pt-BR
nomes no trilho de navegação


L'escheríchia coli (Escherichia coli, pronunciat /eʃe'ɾikia 'kɔli/ i abreujat E. coli habitualment) és una de les principals espècies d'eubacteris que viuen a la part més baixa dels intestins dels animals de sang calenta, incloent-hi ocells i mamífers i són necessaris per a la correcta digestió dels aliments. La seva presència en l'aigua subterrània és un indicador comú de la contaminació fecal ("Entèric" és un adjectiu que descriu els organismes que viuen en els intestins. "Fecal" és l'adjectiu per als organismes que viuen en la femta, de manera que amb freqüència s'empra com a sinònim d'"entèric.") El nom prové del seu descobridor, Theodor Escherich. Pertany a la família de les enterobacteriàcies, i és emprada com a organisme model per als bacteris en general.
De mitjana, un humà treu diàriament amb la femta entre cent mil milions i un bilió d'E. coli. Tots els diferents tipus de coli fecals i tots els bacteris semblants que viuen en el sòl. (en el sòl amb plantes marcint-se el més comú és l'Enterobacter aerogenes) són agrupades juntes sota el nom "coliforme" (que vol dir semblant a coli"). Tècnicament el "grup coliforme" es defineix com el conjunt de bacteris aerobis o anaerobis facultatius, no esporolables, gram-negatius, en forma de bacil i que fermenten lactosa amb producció de gas (els animals eliminen aquest gas en forma de flatulència).
Actualment es considera que l'E coli és l'organisme model i per tant l'ésser viu sobre el que se'n té més coneixement biomolecular. A més a més, gràcies al seguit d'eines desenvolupades al voltant de la seva bioquímica ha demostrat ser altament manipulable al laboratori. Per això mateix és l'eubacteri més emprat per fer als experiments consistents en estudiar les propietats de les biomolècules. Té capacitat per introduir una gran quantitat de proteïnes i fins i tot vies metabòliques senceres dins del seu metabolisme i de produir-les de forma barata en massa, gràcies als seus pocs requeriments del medi. Les seves limitacions són les cadenes d'ADN amb palíndroms, que no és capaç de manipular i que no té capacitat de processar proteïnes eucariotes que requereixin modificacions post-transcripcionals com ara glucosidacions. Actualment és per exemple el principal productor d'insulina.
L'alt coneixement que se'n té ha fet que sigui l'ésser viu que tingui més tecnologia desenvolupada amb la fi de modificar-lo com tota mena de fags i plasmidis. També hi ha moltes eines per tal de conservar i replicar informació com els BAC que es varen fer servir moltíssim en les genoteques implicades en la seqüenciació del genoma humà.
Hi ha, tanmateix, tres situacions en què la E. coli pot causar malalties:
La majoria no produeixen patologia i viuen a l'intestí de l'home i mamífers sense causar malaltia com a flora normal intestinal. El tipus de patologia més freqüent que produeixen són les infeccions del tracte urinari i les gastroenteritis. Moltes poden actuar com a patogen oportunista. E. coli és el principal agent causal de septicèmies i també produeix un tipus de meningitis. Diferents grups de soques produeixen diferents tipus d'infecció i patogènia:
Produeix una diarrea tipus còlera encara que menys severa (diarrea del viatger). La seua patogènia es deu a la producció de toxines. Les ETEC actuen a nivell de mucosa de l'intestí prim a la qual es fixa per mitjà d'unes adhesines fimbriades.
Provoca una diarrea persistent en infants. Forma agregats a la superfície de la mucosa intestinal.
Es desenvolupa a l'intestí prim on forma una alteració de l'estructura de les microvellositats. Els bacteris apareixen enganxats a la mucosa en unes projeccions. La penetració de la mucosa per les cèl·lules succeeix de forma esporàdica i produeix efectes importants sobre la fisiologia de la cèl·lula infectada com l'activació de la tirosina-cinasa i l'increment dels nivells de calci.
Important causa de brots epidèmics de gastroenteritis hemorràgica en països desenvolupats (carn picada). Dosi d'infecció molt baixa (quantitats molt petites poden comportar brots epidèmics seriosos). Similars a la de les EPEC però produeix símptomes semblants a Shigella, produeixen una toxina tipus Shiga (Verotoxina) que pot produir la síndrome hemolítica-urèmica (HUS). S'ha convertit en l'amenaça, a nivell d'infeccions gastrointestinals, més greu als països desenvolupats.
Patologia indistingible de la Shigella però no es produeixen toxines Shiga.
Responsables del 80% d'infeccions urinàries. Freqüents en dones en edat fèrtil. Pot afectar bufeta i uretra (cistitis) o ronyó (pielonefritis). Les infeccions urinàries són el segon tipus d'infecció més freqüent. També són freqüents en infants de menys de 5 anys (d'ambdós sexes). S'ha demostrat l'actuació d'adhesines que permeten adherència a la mucosa del tracte urinari. L'acció inflamatòria localitzada dels lipopolisacàrids (LPS) probablement té un paper important.
En els camps relacionats amb la purificació de l'aigua i tractament d'aigües residuals l'E.coli va ser escollida des de molt d'hora en el desenvolupament de la tecnologia com a "indicador" del nivell de pol·lució de l'aigua mitjançant l'Índex de Coliformes per controlar-ne la contaminació fecal humana. Les raons principals per emprar l'E. coli són que hi ha un munt de coliformes en la femta humana que són patògens (com la Salmonella typhi, que causa el tifus), i l'E. coli és normalment inofensiva, de manera que no es pot escapar de laboratori i fer mal algú. Pot ser enganyós emprar l'E. coli com a indicador de la contaminació fecal humana, ja que hi ha altres ambients en els quals l'E. coli creix bé, com per exemple les fàbriques de paper.
Filogènia de les soques de l'Escherichia coliL'escheríchia coli (Escherichia coli, pronunciat /eʃe'ɾikia 'kɔli/ i abreujat E. coli habitualment) és una de les principals espècies d'eubacteris que viuen a la part més baixa dels intestins dels animals de sang calenta, incloent-hi ocells i mamífers i són necessaris per a la correcta digestió dels aliments. La seva presència en l'aigua subterrània és un indicador comú de la contaminació fecal ("Entèric" és un adjectiu que descriu els organismes que viuen en els intestins. "Fecal" és l'adjectiu per als organismes que viuen en la femta, de manera que amb freqüència s'empra com a sinònim d'"entèric.") El nom prové del seu descobridor, Theodor Escherich. Pertany a la família de les enterobacteriàcies, i és emprada com a organisme model per als bacteris en general.
De mitjana, un humà treu diàriament amb la femta entre cent mil milions i un bilió d'E. coli. Tots els diferents tipus de coli fecals i tots els bacteris semblants que viuen en el sòl. (en el sòl amb plantes marcint-se el més comú és l'Enterobacter aerogenes) són agrupades juntes sota el nom "coliforme" (que vol dir semblant a coli"). Tècnicament el "grup coliforme" es defineix com el conjunt de bacteris aerobis o anaerobis facultatius, no esporolables, gram-negatius, en forma de bacil i que fermenten lactosa amb producció de gas (els animals eliminen aquest gas en forma de flatulència).
Escherichia coli (původním názvem Bacterium coli) je gramnegativní fakultativně anaerobní spory netvořící tyčinkovitá bakterie pohybující se pomocí bičíků. Spadá pod čeleď Enterobacteriaceae, jež také zahrnuje mnoho patogenních rodů mikroorganismů. E. coli patří ke střevní mikrofloře teplokrevných živočichů, včetně člověka. Z tohoto důvodu je její přítomnost v pitné vodě indikátorem fekálního znečištění. Člověku je jako součást přirozené mikroflory prospěšná, jelikož produkuje řadu látek, které brání rozšíření patogenních bakterií (koliciny) a podílí se i na tvorbě některých vitamínů (např. vitamín K). Byla objevena německo-rakouským pediatrem a bakteriologem Theodorem Escherichem v roce 1885[1].
E. coli patří k nejlépe prostudovaným mikroorganismům, jelikož je modelovým organismem pro genové a klinické studie. Joshua Lederberg jako první r. 1947 pozoroval a popsal na bakterii E. coli výměnu genetického materiálu tzv. konjugaci[2].
E. coli je nesporotvorná tyčinka, jež se pohybuje pomocí bičíku. Bakterie dosahuje délky 2–3 μm a šířky 0,6 μm. Některé druhy mohou tvořit slizovité obaly, jež jsou složeny z polysacharidů. Na svém povrchu nese dva typy fimbrií. První typ fimbrií se skládá z kyselého hydrofobního proteinu tzv. fimbrinu. Umožňuje bakterii přichytit se na epitel hostitele a následně jej kolonizovat. Kolonizace je usnadněna vysokým počtem fimbrií prvního typu na buňku (100-1000 ks/buňka). Tyto fimbrie jsou vysoce antigenní, jelikož obsahují tzv. F antigeny. Druhým typem fimbrií jsou tzv. sex pili, jež hrají důležitou úlohu při konjugaci.
Bakterie se může pohybovat pomocí bičíků. Ty jsou složeny z tzv. flagelinu (na lysin bohatý protein). Bičíky jsou stejně jako fimbrie vysoce antigenní, a to díky tzv. H antigenům. Na povrchu bakterie se při stresových podmínkách dále mohou tvořit polysacharidové kapsule, jež obsahují tzv. K a M antigeny.
Vnější membrána je pokryta lipopolysacharidem a skládá se z lipidové dvojvrstvy, kde je ukotveno množství membránových proteinů. Mezi proteiny, které tvoří póry, patří poriny Omp C, Omp F a Pho E. Poriny slouží jako vstupní a výstupní kanály pro buněčné metabolity a pro příjem vitaminů z okolí.
Prostor mezi vnější membránou a buněčnou stěnou se nazývá periplazmatický. Vyskytují se zde např. proteiny vázající aminokyseliny či cukry, enzymy degradující antibiotika (beta-laktamasy).
Buněčná stěna E. coli (jakožto zástupce gramnegativních bakterií) se skládá z tenké vrstvy peptidoglykanu, jenž je zodpovědný za rigidní tvar buňky. Pod vrstvou peptidoglykanu se nalézá cytoplazmatická membrána. Ta se skládá především z proteinů (70%), lipopolysacharidů a fosfolipidů. Je v ní lokalizováno mnoho biochemických pochodů, např. dýchací řetězec a syntéza ATP.
Cytoplazma bakteriální buňky je viskózní vodný roztok, jež obsahuje rozpuštěné anorganické a organické látky. Nachází se zde množství ribozomů (cca 40% hmotnosti celé buňky), díky nimž je proteosyntéza a dělení bakteriálních buněk velice rychlé. Při optimálních podmínkách (37 °C, dostatek živin) je doba generace zhruba 20 min. Bakteriální ribozomy jsou menší než eukaryotní. Mají sedimentační konstantu 70S.
Dále se zde nalézá molekula bakteriální DNA, ve které je uložena veškerá dědičná informace bakterie. Velikost DNA u E. coli K-12 je zhruba 4700 kbp a kóduje cca 4400 proteinů.[3] Cytoplazma bakterií na rozdíl od cytoplazmy eukaryot neobsahuje membránové organely.
E. coli je fakultativní anaerob, tj. využívá respirační i kvasný metabolismus (fermentace) pro přísun energie. E. coli jako chemoheterotrof je schopná využívat množství cukrů i aminokyselin jako zdroj uhlíku, nejrychleji však roste na glukose. Za anaerobních podmínek E. coli utilizuje glukosu za vzniku laktátu, sukcinátu, acetátu i ethanolu. Za aerobních podmínek je glukosa využita efektivněji a konečnými produkty je především oxid uhličitý. E. coli produkuje indol, avšak neroste na citrátu a neprodukuje sirovodík. Je kataláza-pozitivní, oxidáza-negativní. Těchto vlastností se využívá při její identifikaci pomocí tzv. Enterotestu[4]. E. coli je schopná růst za teploty 8 °C-48 °C, avšak optimální teplota je 37 °C. Rozsah pH pro růst je pH6-pH8[3].
E. coli můžeme taxonomicky dělit dle antigenních struktur na sérotypy. Mezi hlavní struktury patří somatické O antigeny (lipopolysacharid), jichž je 170 typů, a kapsulární K antigeny (80 typů), dalšími strukturami jsou např. H antigeny (flagelární proteiny) a F antigeny (bílkoviny fimbrií)[3].
E. coli je jednou z nejčastěji se vyskytujících bakterií v klinických vzorcích. Její přítomnost je u člověka fyziologická pouze ve střevech jako součást střevní mikroflory. Avšak patologicky se může vyskytovat i v krevních vzorcích, a být tak původcem bakterémie. Často způsobuje nosokomiální infekce tj. infekce získané v nemocnicích. Bakteriální infekce se léčí podáváním antibiotik. V dnešní době však nalézáme stále větší množství kmenů bakterií, které jsou k podávaným antibiotikům rezistentní.
Patogenní kmeny způsobují dva typy onemocnění. Prvním je extraintestiální onemocnění, kdy jsou napadeny především močové cesty, dochází k infekci ran a jejich hnisání. Pokud se bakterie dostává do intestinálního traktu člověka, vyvolává infekce provázené průjmy. Extraintestiální formy onemocnění jsou vyvolávány kmeny , jež mají polysacharidový kapsulární K antigen, příp P fimbrie, jimiž adherují na povrch sliznic. Pokud se bakterie E. coli dostane do zažívacího traktu, mluví se o ní jako o enteropatogenním kmenu E. coli. Ty se mohou dále dělit:
EPEC E. coli vyvolává vodnaté průjmy především u novorozenců. Může docházet k vysokému stupni dehydratace a následné smrti. Toto onemocnění je stále problémem v rozvojových zemích. Frekvenci infekce způsobené EPEC E. coli u dospělých jedinců je složité stanovit, jelikož u pacientů starších tři roky se po příčině nezávažného průjmu nepátrá.
ETEC E. coli též vyvolává průjmové stavy, jak u dětí, tak dospělých. Tento kmen se vyskytuje v teplých oblastech (Egypt, Bangladéš), do střední Evropy se může dostat s cestovateli. ETEC se vyznačuje tvorbou dvou typů enterotoxinů-termolabilního (TL) a termostabilního (TS). Informace pro tvorbu těchto toxinů je uložena na bakteriálních plasmidech.
TL enterotoxin se skládá z polypeptidové podjednotky A o molekulární hmotnosti 25 kDa a pěti podjednotek B o molekulární hmotnosti 11,5 kDa. Podjednotka B se váže na epitelové buňky a napomáhá translokaci podjednotky A do buňky. Ta katalyzuje aktivaci cAMP. Enzymatickou kaskádou dochází ke zvýšené sekreci sodíku z buňky, a tím i úniků chloridů a následně i vody. Následkem jsou vodnaté průjmy. ST enterotoxin je slabě imunogenní. Aktivuje guanylátcyklázu, a tím se zvyšuje koncentrace cGMP. Další mechanismus není přesně znám. Je možné, že určitou úlohu hraje vápník[5].
EIEC E. coli pronikají do buněk tlustého střeva, kde se množí. Toto onemocnění připomíná průběh bacilární dysenterie.
O epidemiologii EIEC není mnoho známo. Neexistuje její lidský ani zvířecí rezervoár. V oblastech s nedostatečnou hygienou způsobuje EIEC až 5% všech průjmů. Nejčastější sérologickou skupinou je O124.
EHEC E. coli, stejně jako EPEC, je schopen adheze na stěny endotelu. Na rozdíl od EPEC E. coli se však EHEC váže na endotelie tlustého střeva a produkuje zde toxin, tzv. shigella toxin, či jinak zvaný verotoxin. Verotoxin je zodpovědný za poškození sliznice tlustého střeva, což vede ke krvavým průjmům. Onemocnění, jež tento kmen způsobuje se nazývá hemoragická kolitida. U některých pacientů může dojít k poškození ledvin a onemocnění přechází do hemolyticko-uremického syndromu (HUS), jež bývá smrtelný. Zdrojem infekce je infikované hovězí maso. Identifikace EHEC E. coli se opírá o unikátní neschopnost utilizovat sorbitol[6]. Jedním ze zástupců kmene EHEC je sérotyp E. coli O157:H7.
Poprvé byl izolován v roce 1917 na východní frontě profesorem Alfrédem Nissle. Už on byl toho názoru, že přítomnost tohoto sérotypu ve střevní mikroflóře výrazně snižuje riziko vzniku ulcerózní kolitidy. V nedávné době byl tento objev znovu experimentálně potvrzován, přičemž se ukázalo, že ostatní sérotypy tuto schopnost nemají a dokonce jejich přítomnost ve střeve může snižovat účinnost E. coli nissle. V současnosti se mikrobiologové snaží vyvinout léčbu ulcerózní kolitidy pomocí osazení střeva pacienta E. coli nissle.
Sérotyp E. coli O157: H7 je z hlediska veřejného zdraví nejdůležitějším zástupce E. coli EHEC, jelikož způsobuje HUS. Reservoirem tohoto patogena je především dobytek a přežvýkavci. Na člověka se přenáší kontaminovanou potravou (tepelně neupravené maso a mléko), ale i při fekálním znečištění vody a křížovou kontaminací při přípravě pokrmů. Infekční dávka je velice nízká (jednotky bakterií na gram potravy). Poprvé byl tento serotyp izolován r. 1982[7].
Tento sérotyp byl poprvé izolován r. 1922 na univerzitě ve Stanfordu (USA) z lidské stolice. E. coli K-12 se nejprobádanějším zástupcem E. coli vůbec. Důvodem je jednak přítomnost lysogenního bakteriofága lambda v bakterii, jednak široká vybavenost E. coli K-12 množstvím plasmidů, což dává široké možnosti pro využití v genovém inženýrství.
Sérotyp E. coli K-12 byl použit např. při výzkumu metabolismu dusíku u bakterií, biosyntéze L-tryptofanu z indolu a L-serinu a také při studiích konjugace bakteriích[8].
Bakteriální infekce jsou léčeny antibiotiky (beta-laktamová antibiotika, fluorochinolony a aminoglykosidy). Jelikož E. coli má krátkou generační dobu, může se u ní stejně jako u ostatních bakterií velice rychle vyvinout rezistence k používanému antibiotiku.
Mechanismus účinku beta-laktamových antibiotik (penicilin, ampicilin, cefalosporiny) je založen na skutečnosti, že beta-laktamový kruh antibiotika naruší syntézu buněčné stěny bakterie, a ta, jelikož není chráněná, zahyne.
E. coli může produkovat enzym tzv. beta-laktamázu, jež hydrolyzuje beta-laktamový kruh antibiotika , a tím se stává imunní k působení tohoto antibiotika. Geny pro tuto rezistenci bývají uloženy na plasmidu, a proto je genetická informace velice snadno přístupná k předávání jiným bakteriím (i mezidruhově). Nevýhodou informace uložené na plasmidu, je skutečnost, že nemusí při dělení buňky dojít k jejímu předání oběma dceřiným buňkám. Rezistentní by byla pouze jedna z nově vzniklých buněk, druhá by rezistenci nenesla.
Dnes je známo, že v České republice je E. coli cca ze 60% případů rezistentní k podávání aminopenicilinů a z 15% k cefalosporinům[9].
Fluorochinolony jsou antibiotika, jež inhibují replikaci bakteriální DNA. Pokud si bakterie na ně vyvine rezistenci, může se jednat o dva různé mechanismy. Nejčastějším je tzv. efluxu – tj. po tom, co se antibiotikum dostane do bakteriální buňky, je pomocí membránových pump vyčerpáno mimo buňku.
Druhým způsobem, jak se bakterie brání působení fluorochinolonů je pozměnění (methylace, adenylace) jejich cílových struktur, tedy enzymů, jež jsou zodpovědné za replikaci bakteriální DNA. Tyto enzymy se nazývají DNA gyráza a topoisomeráza IV. V České republice se rezistence kmenů E. coli k fluorochinolonům pohybuje okolo 25% případů, což je zhruba evropský průměr.[9].
Aminoglykosidy jsou antibiotika, která blokují proces translace tj. přepis genetické informace z řeči nukleotidů do řeči aminokyselin. Enzymy, kde translace probíhá se nazývají ribozomy. Rezistence k těmto antibiotikům spočívá v modifikaci ribozomů (methylace, adenylace) tak, že se stanou pro aminoglykosidová antibiotika biologicky inertní.
V České republice se výskyt E. coli rezistentní k aminoglykosidům pohybuje okolo 10% případů, což je v rámci Evropy stále průměrné číslo[9].
Bakterie E. coli se vyskytuje jako součást přirozené mikroflóry teplokrevných živočichů v tlustém střevě a dolní části tenkého střeva. Člověk je touto bakterií kolonizován už od narození (kontaminace z potravy, přenos z již kolonizovaného jedince, nejčastěji matky). E. coli patří k nejlépe prostudovaným známým bakteriím, což je důvodem jejího využití v biotechnologiích a genovém inženýrství. Mezi její klady jakožto zástupce prokaryot patří rychlý růst a levná kultivační media, naopak nevýhodou použití E. coli může být její neschopnost provádět posttranslační modifikace, např. glykosylace. V takovýchto případech je v průmyslu využívána kvasinka Saccharomyces cerevisiae[3].
E. coli je nejběžněji používanou bakterií v metodách molekulární biologie. Její kultivace je úspěšná v médiích bohatých (kromě zdroje uhlíku, dusíku a dalších esenciálních látek tato média obsahují i aminokyseliny a vitaminy) i minimálních (obsahují pouze glukosu jako zdroj uhlíku a energie a soli jako zdroj síry, fosforu apod). Pro klonování se používají speciálně upravené kmeny E. coli, které jsou zbavené schopnosti syntézy restrikčních endonukleáz. Pro účely molekulární genetiky byla připravena řada mutantů E. coli:
Diabetes mellitus I.stupně je onemocnění, kdy slinivka břišní není schopna produkovat insulin. Léčba je substituční, kdy se pacientovi exogenně podává insulin. Dříve byl získáván z jatečních prasat. Docházelo však k alergickým reakcím na takto získaný insulin. Důvodem byla různá lokace glykosylace řetězců insulinu u prasat a u člověka. R. 1982 byl na trh uveden insulin získaný rekombinantně pomocí geneticky modifikované bakterie E. coli pod záštitou firmy Eli Lilly. Insulin se dále získává rekombinantními metodami i pomocí eukaryotních organismů, např. Saccharomyces cerevisiae (pod komerčním názvem Novokin od firmy NovoNordisk)[11].
Roku 1983 byla objevena schopnost bakterie E. coli syntetizovat textilní barvivo indigo. Jeho produkce byla však pro průmyslové účely malá a náklady vysoké (jako výchozí substrát byl používán indol, příp. aminokyselina L-tryptofan), proto byla pomocí rekombinace získána E. coli, jež by mohla jako výchozí substrát používat glukosu. Významným kladem této biotechnologické výroby s využitím modifikované E. coli oproti syntetické výrobě je ekologická nezávadnost, jelikož při syntetické výrobě mohou vznikat toxické meziprodukty[12].
Mezi další produkty rekombinantní E. coli patří např. příprava lidského růstového hormonu, interferonu alfa, interferonu gama, interleukinu-2 či produkce aminokyselin (L-valin, L-threonin). Pod záštitou firmy Eli Lilly se na trh s léčivy dostaly rekombinantní hormony jako jsou parathormon a kalcitonin, jež chrání před osteoporosou). Od r. 1999 je na trhu v USA vakcína proti lymské borelioze s názvem Lymerix. Opět se jedná o rekombinantní protein připravený z E. coli. Tato vakcína je účinná pouze na bakterii Borrelia burgdorferi, jež je majoritním původcem lymské boreloisy v USA (v Evropě je to nejčastěji Borrelia garinii)[13].
Escherichia coli (původním názvem Bacterium coli) je gramnegativní fakultativně anaerobní spory netvořící tyčinkovitá bakterie pohybující se pomocí bičíků. Spadá pod čeleď Enterobacteriaceae, jež také zahrnuje mnoho patogenních rodů mikroorganismů. E. coli patří ke střevní mikrofloře teplokrevných živočichů, včetně člověka. Z tohoto důvodu je její přítomnost v pitné vodě indikátorem fekálního znečištění. Člověku je jako součást přirozené mikroflory prospěšná, jelikož produkuje řadu látek, které brání rozšíření patogenních bakterií (koliciny) a podílí se i na tvorbě některých vitamínů (např. vitamín K). Byla objevena německo-rakouským pediatrem a bakteriologem Theodorem Escherichem v roce 1885.
E. coli patří k nejlépe prostudovaným mikroorganismům, jelikož je modelovým organismem pro genové a klinické studie. Joshua Lederberg jako první r. 1947 pozoroval a popsal na bakterii E. coli výměnu genetického materiálu tzv. konjugaci.
Escherichia coli (abgekürzt E. coli) – auch Kolibakterium genannt – ist ein gramnegatives, säurebildendes und peritrich begeißeltes Bakterium, das normalerweise im menschlichen und tierischen Darm vorkommt. Unter anderem auf Grund dessen gilt dieses Bakterium auch als Fäkalindikator.[1] E. coli und andere fakultativ anaerobe Organismen machen etwa 1 ‰ der Darmflora aus.[2]
Innerhalb der Familie der Enterobakterien (altgriechisch ἕντερον, lateinisch enteron „Darm“) gehört E. coli zur bedeutenden Gattung Escherichia und ist deren Typspezies.[3] Benannt wurde es nach dem deutschen Kinderarzt Theodor Escherich, der es erstmals beschrieb. Coli ist der lateinische Genitiv von colon (zu dt. Kolon), einem Teil des Dickdarms.[4]
Die meisten Angehörigen dieser Spezies sind nicht krankheitsauslösend, jedoch gibt es auch zahlreiche verschiedene pathogene Stämme.[5] Es zählt zu den häufigsten Verursachern von menschlichen Infektionskrankheiten.[1] Die Basensequenz des Genoms einiger Stämme ist vollständig aufgeklärt. Als Modellorganismus zählt es zu den am besten untersuchten Prokaryoten[6] und nimmt in der Molekularbiologie eine wichtige Rolle als Wirtsorganismus ein. Der Nobelpreis für Physiologie oder Medizin wurde an zahlreiche Forscher, die sich mit der Biologie von E. coli beschäftigt haben, vergeben.[7]
E. coli hat die Form gerader, zylindrischer Stäbchen mit runden Enden. Der Durchmesser beträgt 1,1–1,5 µm und die Länge 2,0–6,0 µm. Sie kommen paarweise oder einzeln vor. In der Gram-Färbung verhalten sie sich negativ (gramnegativ).[3] Es bildet keine Bakteriensporen.[6] Die Zellen bestehen überwiegend (70–85 %) aus Wasser, wobei die Trockenmasse zu 96 % aus Polymeren besteht, unter denen die Proteine dominieren. Es sind 4288 unterschiedliche Proteine annotiert. Im Cytoplasma als auch in der Zellhülle (bestehend aus Zellmembran, Periplasma, äußere Membran) erfüllen sie strukturelle, enzymatische und regulatorische Funktionen. Das Genom umfasst etwa 4600 Kilobasenpaare und kommt als kovalent in sich geschlossenes Bakterienchromosom vor.[8]
Viele Stämme besitzen Fimbrien (Pili). Eine Zelle des Stammes K-12 enthält typischerweise etwa 100–500 Typ 1 Fimbrien mit einer Länge von 0,2–2,0 µm und einem Durchmesser von ca. 7 nm. Es gibt mehr als 30 verschiedene Arten von Fimbrien, die in zwei nach ihren adhesiven Eigenschaften an rote Blutkörperchen eingeteilt werden: MS (Mannose-sensitive), die in Anwesenheit von Mannose rote Blutkörperchen nicht verklumpen können (Hämagglutination) und MR (Mannose-resistente), denen die Präsenz des Zuckers nichts ausmacht. Typ 1 Fimbrien, die zu den MS-Fimbrien gehören, kommen sowohl in symbiotischen als auch in pathogenen Stämmen vor und werden daher nicht zur Differenzierung herangezogen. MR-Fimbrien sind serologisch divers und fungieren häufig als Virulenzfaktoren. Ihr Anhaften ist sowohl spezies- als auch organspezifisch.[3] Zusätzlich bildet E. coli auch einen Sexpilus aus (auch F-Pilus, F für Fertilität) mit dem Zell-Zell-Kontakte zum Austausch von genetischer Information (Konjugation) möglich sind. Zudem dient der F-Pilus auch einigen Bakteriophagen als Rezeptor nach deren Bindung die Virus-DNA eingeschleust wird (Transduktion).[9]
Zellen von E. coli können sich durch peritriche Begeißelung aktiv bewegen (sie sind motil) oder sie sind – seltener – zur aktiven Bewegung unfähig. Motile E. coli bewegen sich mit ihrem proteinösen Flagellum fort, wobei sie wiederholt ihre Richtung ändern: Ein Bakterium bewegt sich in eine Richtung fort, indem sich die Flagellen bündeln und zusammenarbeiten. Die Fortbewegung wird zeitweise kurz durch Taumeln unterbrochen, indem sich das Flagellenbündel auflöst und die einzelnen Flagellen sich in verschiedene Richtungen wenden. Danach reformiert sich das Flagellenbündel und beschleunigt das Bakterium in eine neue Richtung. Die Stabilität des Bündels wird durch Chemorezeptoren verstärkt. Bietet man den Bakterien einen Nährstoff an, so wird die Stabilität des Flagellenbündels weiter verstärkt, und die Bakterien akkumulieren.
E. coli ist chemotaktisch: Schwimmen die Individuen in einem Konzentrationsgefälle eines Lockstoffs in Richtung ansteigender Konzentration, so ändern sie weniger häufig ihre Richtung. Schwimmen sie ein Konzentrationsgefälle herunter, ist ihr Bewegungsmuster nicht von dem in einer isotropen Lösung zu unterscheiden, und sie ändern häufiger die Richtung.[10] Neben positiver Chemotaxis kann E. coli sich auch aktiv von Schadstoffen entfernen (negative Chemotaxis), wobei niedrige Konzentrationen an Schadstoffen keine Lockstoffe darstellen und hohe Nährstoffkonzentration nicht abstoßend wirken. Es gibt Mutanten, die gewisse Schadstoffe nicht erkennen, und nicht-chemotaktische Mutanten, die auch keine Lockmittel erkennen können. Der Prozess benötigt L-Methionin.[11]
Die Signaltransduktion für akkurate chemotaktische Reaktionen hat sich im Laufe der Evolution auf optimale Arbeitsleistung bei minimaler Proteinexpression entwickelt. Aufgrund des hohen Selektionsdrucks ist die Chemotaxis bei E. coli sehr sensitiv, besitzt ein schnelles Ansprechverhalten und ist perfekt angepasst. Zudem scheint die Anordnung innerhalb des bakteriellen chemosensorischen Systems hochkonserviert.[12]
Zum Stoffaustausch besitzt E. coli Transportproteine in der Zellmembran. Unter den Porinen dominieren Outer-membrane-Proteine OmpF und OmpC, welche zwar nicht substratspezifisch sind, jedoch kationische und neutrale Ionen bevorzugen und hydrophobe Verbindungen nicht akzeptieren. Die Kopienzahl hängt von der Osmolarität der Umgebung ab und dient der Anpassung an den Lebensraum. Unter den Bedingungen im Dickdarm (Hyperosmolarität, höhere Temperatur) überwiegen OmpC-Kanäle. Verlässt das Bakterium seinen Wirt und findet sich in einem weniger bevorzugten Lebensraum, z. B. einem Gewässer (niedrigere Osmolarität und Temperatur) so wird die OmpF-Synthese gefördert. Für Substrate, die durch die unspezifischen Porine gar nicht oder unzureichend transportiert werden, gibt es substratspezifische Porine. Bei Phosphatmangel exprimiert E. coli das Protein PhoE. Zusammen mit Maltodextrinen entstehen daraus Maltoporine, die auch als Rezeptor für die Lambda-Phage fungieren und daher auch LamB genannt werden. Stämme, die Saccharose verwerten können, nehmen diese über das Kanalprotein ScrY auf. Langkettige Fettsäuren werden mit FadL in die Zelle transportiert.[13]
E. coli ist heterotroph, fakultativ anaerob und besitzt die Fähigkeit, Energie sowohl durch die Atmungskette als auch durch „Gemischte Säuregärung“ zu gewinnen. Die Gärungsbilanz bei E. coli sieht folgendermaßen aus:[14]


Glucose wird von E. coli unter Säurebildung vergoren, was mit Methylrot als pH-Indikator nachgewiesen werden kann. Neben Säure bildet E. coli aus Glucose auch Gas. Der Indol-Test auf Tryptophanase ist positiv. Die Voges-Proskauer-Reaktion zum Nachweis der Acetoin-Bildung fällt negativ aus. Auf Simmons Citrat-Agar ist keine Verfärbung sichtbar, da E. coli Citrat nicht als alleinige Energiequelle nutzen kann. Zudem kann es Malonat nicht verwerten. Acetat und Tartrat können verstoffwechselt werden (Test mit Methylrot nach Jordan). Nitrat kann zu Nitrit reduziert werden. Auf Triple Sugar Iron-Agar wird kein Schwefelwasserstoff gebildet. E. coli kann keinen Harnstoff und keine Gelatine hydrolysieren, einige Stämme jedoch Esculin. Lysin wird von vielen Stämmen decarboxyliert, Ornithin nur von wenigen. Im Kaliumcyanid-Wachstumstest wächst E. coli nicht. Es besitzt keine Phenylalanindeaminase, keine Lipase und keine DNase im engeren Sinne. Der Oxidase-Test mit Kovacs-Reagenz ist stets negativ. Des Weiteren kann von den meisten Stämmen L-Arabinose, Lactose, Maltose, D-Mannitol, D-Mannose, Mucinsäure, D-Sorbitol, Trehalose und D-Xylose fermentativ genutzt werden.[3]
Die Serotypisierung ist eine nützliche Möglichkeit E. coli anhand der zahlreichen Unterschiede in der Antigenstruktur auf der Bakterienoberfläche einzuteilen.[3]
Man unterscheidet vier Gruppen von Serotypen:
Selten zur Diagnostik eingesetzt werden:
E. coli kommt als universeller und kommensaler Begleiter im unteren Darmtrakt warmblütiger Tiere (inklusive des Menschen) vor. Im Stuhl befinden sich etwa 108–109 koloniebildende Einheiten pro g.[6] Es kann auch in anderen Habitaten überleben. Bei Neugeborenen spielt es als Erstbesiedler eine wichtige Rolle. Obwohl es selbst nur in geringer Zahl vorhanden ist, dient es der Ansiedlung obligater Anaerobier, die physiologische Bedeutung bei der Verdauung besitzen.[18] Trotz des geringen Anteils im Darm nimmt E. coli eine beherrschende Stellung im Darm ein, den es bei Menschen innerhalb von 40 Stunden nach der Geburt über Lebensmittel, Wasser oder andere Individuen kolonisiert. Die Fähigkeit zum Anhaften an den Mucus erlaubt E. coli, lange Zeit im Darm zu verweilen. Obwohl sehr viel über den Organismus bekannt ist, weiß man über seine Ökologie im Darm relativ wenig.[19]
Sporadische durch Trinkwasser übertragene Ausbrüche von enterotoxischen Stämmen (ETEC) sind bekannt. Zudem werden ETEC durch Konsum von Weichkäse und rohem Gemüse übertragen. Ausbrüche von enteropathogenen Stämmen (EPEC) werden häufig mit kontaminiertem Trinkwasser und einigen Fleischprodukten in Verbindung gebracht. Infektionen mit enterohämorrhagischen E. coli (EHEC) stammen häufig von Lebensmitteln oder geschehen mittels Wasser. Häufig infizierte Lebensmittel sind nicht durchgegartes Rinderhack, Rohmilch, kalte Sandwiches, Wasser, nicht-pasteurisierter Apfelsaft, Sprossen und rohes Gemüse.[20] Zudem standen Epidemien im Zusammenhang mit Hamburgern, Roastbeef, Kohlrouladen und Rohwurst (Teewurst).[21]
In 1–2 % des Rinderkots kann der für Rinder ungefährliche magensäureresistente Stamm Escherichia coli O157:H7 (EHEC) nachgewiesen werden, welcher bei der Schlachtung auch das Fleisch kontaminieren und bei Menschen schwere Lebensmittelvergiftungen auslösen kann. Der Grund hierfür ist, dass häufig stärkehaltiges Getreide gefüttert wird, das im Pansen unvollständig abgebaut und zu Säure fermentiert wird, so dass sich acidophile Bakterien dort anreichern. Die Fütterung von Heu oder Gras reduziert die Anzahl humanpathogener Stämme.[22]
Da eine komplette EHEC-Sanierung der Tierbestände nicht möglich ist, muss die Prophylaxe bei der Schlachthygiene ansetzen. Rindfleischprodukte sollten mindestens 10 Minuten bei mindestens 70 °C durchgegart werden. Aufgrund der hohen Umweltresistenz der Erreger sollten Lebensmittelhersteller Belastungstests und HACCP-Analysen durchführen. Risikogruppen (Kinder unter 6 Jahren und immunsupprimierte Personen) sollten rohe Produkte nicht verzehren.[21]
Nach der EU-Badegewässerverordnung von 2008 gelten für E.coli folgende Grenzwerte:[23]
Bei Trinkwasser gilt hingegen ein restriktiver Grenzwert von 0 CFU / 100 ml.[24]
Sandstrände können besonders von E.coli betroffen sein, da der Abbau der Abwasserbakterien im Sand länger dauert als im Meerwasser.[25]
Der Stamm Escherichia coli Alfred Nissle 1917 (Handelsname Mutaflor) zählt zu den am häufigsten untersuchten Probiotika. Er wurde während des Balkankriegs vom Stuhl eines Soldaten isoliert, der im Gegensatz zu seinen Kameraden nicht an Durchfall litt. Der Stamm ist mittlerweile sequenziert und besitzt sechs verschiedene Systeme, um Eisen aufzunehmen, wodurch er Konkurrenten aus dem Feld schlägt. Er besitzt Adhesine für eine effektive Kolonisierung und blockiert das Anhaften und Eindringen von pathogenen Bakterien an die Epithelzellen des Darms. Zudem besitzt es einen entzündungshemmenden Effekt auf die T-Zellen-Proliferation. Des Weiteren wird die Produktion von menschlichem β-Defensin 2 angeregt, das als Breitspektrumantibiotikum sowohl grampositive als -negative Bakterien, Pilze und Viren abtötet.[26]
Die meisten Stämme von E. coli sind nicht pathogen und damit harmlos. Einige Serotypen spielen jedoch eine wichtige Rolle bei Erkrankungen innerhalb und außerhalb des Darms.[27] In Wirten mit Immunschwäche ist E. coli ein opportunistischer Erreger, das heißt, erst durch die Schwächung des Wirts kann er wirksam werden.[3] Uropathogene E. coli (UPEC) sind für unkomplizierte Harnwegsinfektionen verantwortlich.[28][29] Neonatale Meningitis auslösende E. coli (NMEC) können die Blut-Hirn-Schranke passieren und bei Neugeborenen eine Hirnhautentzündung auslösen. NMEC und UPEC führen im Blutstrom zur Sepsis.[30]
Es wird vermutet, dass E. coli mit chronisch-entzündlichen Darmerkrankungen wie Morbus Crohn und Colitis ulcerosa assoziiert ist, da neben genetischer Prädisposition und Umweltfaktoren an der Krankheitsentstehung unter anderem auch eine fehlregulierte Immunantwort der Schleimhaut gegen kommensale Bakterien beteiligt sein könnte.[31] Die Schleimhaut der Patienten ist abnormal mit adhärent-invasiven E. coli (AIEC) kolonisiert, welche an den Epithelzellen anhaften und in sie eindringen.[32]
Die darmpathogenen E. coli werden in fünf verschiedene Pathogruppen unterteilt. Weltweit verursachen sie jährlich 160 Mio. Durchfallerkrankungen und 1 Mio. Todesfälle. In den meisten Fällen sind Kinder unter 5 Jahren in den Entwicklungsländern betroffen.[33]
Enteropathogene E. coli (kurz EPEC) sorgen bei Kleinkindern für schwere Durchfälle, die in industrialisierten Gesellschaften selten, in unterentwickelten Ländern jedoch häufig für kindliche Todesfälle verantwortlich sind. Mithilfe des EPEC Adhäsionsfaktor (EAF) heften sich die EPEC an die Epithelzellen des Dünndarms und injizieren dann Toxine in die Enterozyten mit Hilfe eines Typ-III-Sekretionssystems.[1] Es existieren auch sogenannte atypische EPEC. Sie zeigen die bei STEC gängigen Serotypen sowie Virulenz- und Pathogenitätsfaktoren wie beispielsweise das eae-Gen. Den Stx-Prophagen und die damit zugehörigen für STEC charakteristischen stx-Gene haben sie jedoch wahrscheinlich verloren.[34]
Enterotoxische E. coli (kurz ETEC) sind häufiger Erreger der Reisediarrhoe („Montezumas Rache“). Grund für diese Erkrankung ist ein hitzelabiles Enterotoxin vom A/B Typ (LT I und LT II), sowie ein hitzestabiles Enterotoxin (ST). Dieses 73 kDa große Protein besitzt zwei Domänen, von denen sich eine an ein G-Gangliosid der Zielzelle bindet (Bindende Domäne). Die andere Domäne ist die Aktive Komponente, die ähnlich dem Choleratoxin (etwa 80 % Genhomologie) die Adenylatcyclase aktiviert. Das etwa 15–20 Aminosäuren lange ST aktiviert die Guanylatcyclase. Die Aktivierung der Adenylatcyclase und der Guanylatcyclase endet in einer sekretorischen Diarrhoe, bei der viel Wasser und Elektrolyte verloren gehen. Die genetische Information erhält das Bakterium von einem lysogenen Phagen durch Transduktion.[33]
Enteroinvasive E. coli (kurz EIEC) penetrieren die Epithelzellen des Kolons und vermehren sich dort. Innerhalb der Zelle kommt es zur Aktinschweifbildung, womit sie wie Listerien und Shigellen in benachbarte Epithelzellen eindringen. Es kommt zu Entzündungen und Geschwürbildung unter Absonderung von Blut, Schleim und weißen Blutkörperchen (Granulozyten). Zudem können EIEC Enterotoxine abgeben, die zu Elektrolyt- und Wasserverlust führen. Das Krankheitsbild ähnelt einer Bakterienruhr mit Fieber und blutig-schleimigen Durchfällen, wobei häufig eine abgeschwächte Symptomatik mit wässriger Diarrhoe einhergeht.[33]
Enterohämorrhagische E. coli (kurz EHEC) sind Shigatoxin produzierende E. coli (STEC) mit zusätzlichen Pathogenitätsfaktoren. Das Shigatoxin wirkt enterotoxisch und zytotoxisch und zeigt Ähnlichkeiten mit dem von Shigellen gebildeten Toxin. Analog werden VTEC (Verotoxin produzierende E. coli) benannt. Durch EHEC verursachte Darmerkrankungen wurden vornehmlich unter dem Namen enterohämorrhagische Colitis bekannt. EHEC-Infektionen zählen zu den häufigsten Ursachen für Lebensmittelvergiftungen. Der Erreger ist hoch infektiös: 10 – 100 Individuen sind für eine Erkrankung ausreichend. Die niedrige Infektionsdosis begünstigt eine Übertragung von Mensch zu Mensch. Eine Infektion kann jedoch auch durch Tierkontakt (Zoonose) oder durch Verschlucken von Badewasser erfolgen. Typische Krankheitsbilder sind die thrombotisch-thrombozytopenische Purpura (TTP) und ein hämolytisch-urämisches Syndrom (HUS). Gefürchtet ist vor allem HUS aufgrund der Möglichkeit, an einem terminalen Nierenschaden zu sterben. Hierbei sind alle Altersgruppen betroffen, jedoch vor allem Kinder unter 6 Jahren. Das Nierenversagen verläuft in 10 – 30 % der Fälle mit dem Tod des Patienten innerhalb eines Jahres nach Beginn der Erkrankung.[21]
Enteroaggregative E. coli (EAggEC oder EAEC abgekürzt) besitzen die Fähigkeit zur Autoaggregation. Sie heften sich mit spezifischen Fimbrien an das Dünndarmepithel. Charakteristisch ist die erhöhte Schleimproduktion der Mukosazellen, die eine Ausscheidung verzögert. Es kommt zu einer Diarrhoe vom sekretorischen Typ aufgrund von Enterotoxinen (EAST). Durch EAEC werden sowohl akute als auch chronisch rezidivierende Durchfallerkrankungen, die sich über Wochen hinziehen können, verursacht. Neben wässrig schleimigem Durchfall kann es auch zu Fieber und Erbrechen oder blutigem Stuhl kommen. Bei Immunsupprimierten (z. B. HIV-Patienten) ist EAEC der häufigste Erreger einer bakteriellen Enteritis.[33]
E. coli ist für eine Vielzahl von Infektionskrankheiten bei Tieren verantwortlich. Spezifische veterinärmedizinische Krankheitsbilder sind:
Beim Hausschwein lösen extraintestinal pathogene (ExPEC) Stämme eine hämorrhagische Septikämie aus, die als Differentialdiagnose zur klassischen Schweinepest angesehen wird.[35]
Coliforme Bakterien werden als Indikator für die sanitäre Gewässergüte und die Hygiene bei der Lebensmittelverarbeitung verwendet. Fäkalcoliforme Keime gelten insbesondere bei Schalentieren als der Standardindikator für Verunreinigung. E. coli zeigt fäkale Verunreinigung sowie unhygienische Verarbeitungen an. Klassischerweise basieren die biochemischen Methoden zum Nachweis von E. coli, Gesamtcoliformen oder Fäkalcoliformen auf der Verwertung von Lactose. Eine Auszählung ist mittels MPN-Verfahren möglich. Gebräuchlich ist eine Brillantgrün-Galle-Lactose-Bouillon in denen Gasbildung beobachtet wird. Es gibt jedoch auch spezielle Nährböden wie Eosin-Methylen-Blau, VRB-Agar (Kristallviolett-Neutralrot-Galle-Agar), MacConkey-Agar[20] und Endo-Agar, die Lactoseverwertung anzeigen. Zur Differenzierung von anderen Enterobakterien kann der IMViC-Test durchgeführt werden.[36]
Pathogene E. coli werden ebenfalls zunächst angereichert. Das hitzelabile Enterotoxin (LT) der ETEC kann durch einen Y-1-Nebennieren-Zell-Assay, Latexagglutinations-Assay und ELISA nachgewiesen werden. Der Nachweis für das hitzestabile Toxin (ST) der ETEC kann ebenfalls mittels ELISA oder Jungmaus-Assay erfolgen. Die Gene für LT und ST sind bekannt und können mittels PCR oder mittels Gensonde nachgewiesen werden. In Verbindung mit Ausplattierung auf Agarkulturmedien können so ETEC-positive Kolonien gezählt werden. EIEC sind nicht-motil und anaerogen, da sie kein Lysin decarboxylieren und keine Lactose fermentieren. Der invasive Phänotyp von EIEC, der durch ein hochmolekulares Plasmid codiert wird, kann mittels HeLa-Zellen oder Hep-2-Gewebezellkulturen nachgewiesen werden. Alternativ können PCR- und Sondenmethoden für die Invasionsgene eingesetzt werden. Das Protein Intimin der EPEC wird durch ein eae-Gen codiert auf das mit PCR getestet werden kann. Des Weiteren wird der EPEC-adhärent-Faktor (EAF) über ein Plasmid codiert. Das Protein lässt sich mit Hep-2-Zellen nachweisen. EHEC kann über die Shiga-Toxine (Stx) nachgewiesen werden. Insbesondere Stx1 und Stx2 werden mit menschlichen Erkrankungen in Zusammenhang gebracht, wobei zahlreiche Varianten von Stx2 existieren. Die Produktion von Stx1 und Stx2 kann mit Zytotoxizitätstests auf Vero-Zellen oder Hela-Gewebekulturen sowie durch ELISA und Latexaggulationstests erfolgen. Zudem gibt es PCR-Assays für Stx1, Stx2 oder andere charakteristische Marker. EHEC zeichnen sich zudem durch keine oder langsame Fermentation von Sorbitol aus.[20] Eine weitere biochemische Differenzierung ist der LST-MUG-Assay, welcher auf der enzymatischen Aktivität von β-Glucuronidase (GUD) basiert. GUD setzt das Substrat 4-Methylumbelliferyl-β-D-glucuronid (MUG) in 4-Methylumbelliferon um, welches bei 365 nm (UV-Licht) eine blaue Fluoreszenz zeigt. GUD wird von>95 % der E. coli-Stämme produziert (inklusive denen, die kein Gas produzieren), jedoch nicht von EHEC des Serotyps O157:H7, weshalb es hier zur Differenzierung verwendet werden kann.[36]
Die Therapie bei fakultativ pathogenen Stämmen sollte immer gezielt nach Antibiogramm erfolgen. E. coli-Arten (insbesondere ESBL-Stämme im Gegensatz zum Wildtyp, der gut sensibel gegen Cephalosporine ist)[37] besitzen Antibiotikaresistenzen durch die Bildung zahlreicher β-Lactamasen, die in der Lage sind, β-Lactam-Antibiotika zu spalten (ESBL-Stämme, die resistent gegen alle β-Lactama-Antibiotika außer Carbapenem resistent sind, sind zudem häufig multiresistent – auch gegenüber Chinolonen[38]).
Mittel der Wahl sind Aminopenicilline, Ureidopenicilline, Cephalosporine (idealerweise ab der 2. Generation), Carbapeneme, Chinolon-Antibiotika und Cotrimoxazol. Aminoglycoside werden in Ausnahmesituationen kombiniert.
Bei Befall der Harnwege eignen sich insbesondere Cotrimoxazol (bei Sensitivität) und Cefuroxim, alternativ auch Levofloxacin, Ciprofloxacin und Fosfomycin; bei Bakteriämie und Sepsis Cefotaxim und Ceftriaxon, und alternativ Levofloxacin und Ciprofloxacin. Bei einer Meningitis durch E. coli eignen sich Ceftriaxon, Cefotaxim und alternativ Meropenem. Infektionen mit ESBL-positiven Stämmen werden mit Ertapenem, Imipenem, Meropenem, Levofloxacin oder (wenn sensibel) mit Ciprofloxacin therapiert.[38]
Bei obligat pathogenen E.-coli-Stämmen sind die Gastroenteritidien selbstlimitierend. Der starke Flüssigkeitsverlust muss jedoch insbesondere bei Säuglingen und Kleinkindern behandelt werden. Für den Wasser- und Salzverlust bieten sich orale Rehydratationslösungen an. Die zweimalige Gabe von Antibiotika (z. B. Ciprofloxacin und Cotrimoxazol) innerhalb von 24 h kann Dauer und Schwere der Erkrankung lindern.[39]
Eine rasche Keimzahlreduktion kann nur bei sehr frühem Einsatz erzielt werden. Bei der Therapie von enterohämorrhagischen E. coli mit Antibiotika kann es durch vermehrte Ausschüttung von Verotoxin zu Komplikationen kommen. Insbesondere bei der Gabe von Fluorchinolonen, Cotrimoxazol, Aminoglycosiden und Fosfomycin überwiegen die ungünstigen Wirkungen. Dies gilt nicht in gleichem Maße für Carbapeneme. Auch der Einsatz neuerer Makrolide und gegebenenfalls von Clindamycin sowie Rifampicin und Rifaximin bei gegebener Indikation (z. B. Sanierung von Meningokokken-Trägern) scheint nicht kontraindiziert. Jedoch bleibt der Einsatz dieser Substanzen im Sinne einer EHEC-Virulenzabschwächung durch Reduktion der Verotoxinproduktion kontrovers.[40]
In Deutschland ist der direkte oder indirekte Nachweis von Escherichia coli (enterohämorrhagische Stämme (EHEC) und sonstige darmpathogene Stämme) namentlich meldepflichtig nach des Infektionsschutzgesetzes, soweit der Nachweis auf eine akute Infektion hinweist.
Entdeckt wurde E. coli 1885 von Theodor Escherich, der es damals „Bacterium coli commune“ nannte.[41] 1919 wurde es ihm zu Ehren umbenannt.[42] 1892 wurde von Shardinger vorgeschlagen, E. coli als Indikatororganismus für fäkale Verunreinigung zu verwenden. In der Praxis war es jedoch schwierig, mit rein biochemischen Nachweismethoden E. coli von anderen Enterobakterien abzugrenzen, weshalb die nicht taxonomische Bakteriengruppe coliforme Bakterien definiert wurde.[36] 1997 wurde die DNA-Sequenz aufgeklärt.[8] Hierfür wurden 15 Jahre benötigt.[43]
Serotyp O7:K1 (Stamm IAI39 / ExPEC)
Stamm SMS-3-5 / SECEC
Serotyp O127:H6 (Stamm E2348/69 / EPEC)
Serotyp O6:H1 (Stamm CFT073 / UPEC)
Serotyp O45:K1 (Stamm S88 / ExPEC)
Serotyp O1:K1 / APEC
Stamm UTI89 / UPEC
Serotyp O81 (Stamm ED1a)
Serotyp O6:K15:H31 (Stamm 536 / UPEC)
Serotyp O157:H7 (Stamm TW14359 / EHEC)
Serotyp O157:H7 (Stamm EC4115 / EHEC)
Serotyp O157:H7 (Stamm EDL933 / EHEC)
Serotyp O139:H28 (Stamm E24377A / ETEC)
Stamm 55989 / EAEC
Serotyp O8 (Stamm IAI1)
Stamm SE11
Serotyp O103:H2 (Stamm 12009 / EHEC)
Stamm Crooks
Stamm B / BL21-DE3
Stamm B / BL21
Stamm K12 / MC4100 / BW2952
Stamm K12
Stamm K12 / DH10B
Stamm K12 / DH1
Serotyp O9:H4 (Stamm HS)
Serotyp O17:K52:H18 (Stamm UMN026 / ExPEC)
Das Kerngenom, das sich in allen Stämmen wiederfindet, macht lediglich 6 % der Genfamilien aus. Über 90 % der Gene sind variabel. Die Diversität innerhalb der Spezies und die überlappenden Geninhalte mit verwandten Spezies lassen einen Übergang anstelle einer scharfen Speziesabgrenzung innerhalb der Enterobacteriaceae vermuten.[45] Insbesondere zwischen der Gattung Shigella und enteroinvasiven E. coli gibt es eine enge evolutionäre Verwandtschaft sowohl in der chromosomalen DNA als auch bei dem Virulenzplasmid.[46]
Man unterscheidet anhand von phylogenetischen Analysen vier Hauptgruppen (A, B1, B2, und D), wobei die virulenten extraintestinalen Stämme hauptsächlich zu den Gruppen B2 und D gehören.[47] Innerhalb der Gattung Escherichia ist E. fergusonii die nächste verwandte Art.[48]
Der extraintestinal pathogene (ExPEC) Serotyp O7:K1 (Stamm IAI39) löst beim Hausschwein eine hämorrhagische Septikämie aus. Er hat keine ETEC (enterotoxische) oder EDEC (Ödemkrankheit-verursachende E. coli) Virulenzfaktoren. Stattdessen besitzt er P-Fimbrien und Aerobactin[35] Stamm SMS-3-5 / SECEC wurde in einem küstennahen Industriegebiet, mit Schwermetallen verseuchtem Gebiet isoliert. Es ist gegen zahlreiche Antibiotika in hohen Konzentrationen resistent.[49]
Serotyp O127:H6, Stamm E2348/69 war der erste sequenzierte und am besten untersuchte enteropathogene E. coli (EPEC).[50]
Im Vergleich mit anderen pathogenen Stämmen zeigt CFT073 des Serotyps O6:H1 das Fehlen eines Typ-III-Sekretionssystems und keine phagen- oder plasmidcodierten Toxine. Stattdessen besitzt er fimbriale Adhesine, Autotransporter, Eisen-Sequestierungssysteme und Rekombinasen. Schlussfolgernd kann man sagen, dass extraintestinal pathogene E. coli unabhängig voneinander entstanden sind. Die verschiedenen Pathotypen haben hohe Syntänie, die durch vertikalen Gentransfer entstanden ist und ein gemeinsames Rückgrat bildet, das durch zahlreiche Inseln aufgrund von horizontalem Gentransfer unterbrochen wird.[51]
Der Serotyp O45:K1 (Stamm S88 / ExPEC) wurde 1999 aus der Zerebrospinalflüssigkeit eines spät ausbrechenden Meningitisfalles in Frankreich isoliert. Er gehört zur phylogenetischen Gruppe B2.[52] Der Serotyp O1:K1 löst bei Vögeln Krankheiten aus und wird „avian pathogen Escherichia coli“ (APEC) genannt. Er ist mit drei menschlichen uropathogenen (UPEC) eng verwandt.[53] UTI89 ist ein uropathogener E.-coli-Stamm, der aus einem Patienten mit akuter Blaseninfektion isoliert wurde.[54] Der Stamm ED1a ist hingegen apathogen und wurde aus dem Stuhl eines gesunden Mannes isoliert.[55] Der uropathogene Stamm 536 (O6:K15:H31) stammt ursprünglich von einem Patienten mit akuter Pyelonephritis. Er ist ein Modellorganismus für extrainstestinale E. coli. Das Genom enthält fünf gut charakterisierte Pathogenitätsinseln und eine neu entdeckte sechste, die den Schlüsselvirulenzfaktor darstellt.[56]
Der gefürchtete Lebensmittelvergifter E. coli O157:H7 wurde vollständig sequenziert, um seine Pathogenität zu verstehen. Die Erklärung liefert ein massiver lateraler Gentransfer. Über 1000 neue Gene finden sich in diesem stammspezifischen Cluster. Enthalten sind mögliche Virulenzfaktoren, alternative metabolische Fähigkeiten sowie zahlreiche Prophagen, die für die Lebensmittelüberwachung genutzt werden können.[57] Das Virulenzplasmid pO157 besitzt 100 offene Leserahmen. Ein ungewöhnlich großes Gen hat ein mutmaßliches aktives Zentrum, das mit der Familie des großen clostridialen Toxins (LCT) und Proteinen wie ToxA und B von Clostridium difficile verwandt ist.[58] Der Stamm TW14359 wurde 2006 nach einem Ausbruch von E. coli O157:H7 in den USA aus Spinat isoliert. Es wurden neue Genabschnitte festgestellt, welche die erhöhte Fähigkeit zur Auslösung des hämolytisch-urämischen Syndroms oder alternativ die Anpassung an Pflanzen erklären. Zudem enthält der Stamm Gene für intakte anaerobe Nitritreduktasen.[54]
Der Serotyp O139:H28 (Stamm E24377A / ETEC) ist ein enterotoxisches Isolat. Er besitzt das Kolonisationsfaktorantigen (colonization factor antigen, CFA) Pili, um sich anzuheften. 4 % des Genoms bestehen aus Insertionssequenzen. Vermutlich kann der Stamm Propandiol als einzige Kohlenstoffquelle verwerten.[59] Stamm 55989 / EAEC ist ein enteroaggregativer Stamm, der 2002 in der Zentralafrikanischen Republik aus dem Stuhl eines HIV-positiven Erwachsenen, der an stark wässrigem Durchfall litt, isoliert wurde.[60]
Der Stamm IAI1 vom Serotyp O8 ist ein bei Menschen kommensaler Stamm, der aus den Fäzes eines gesunden Franzosen in den 1980ern isoliert wurde.[61] Bakterien des Stammes SE11 vom Serotyp O152:H28 wurden ebenfalls aus den Fäzes eines gesunden Menschen isoliert. Im Vergleich mit dem Laborstamm K-12 MG1655 besitzt der Stamm zusätzliche Gene für Autotransporter und Fimbrien, um sich an den Darmzellen zu befestigen. Zudem besitzt er mehr Gene, die für den Kohlenhydratstoffwechsel von Bedeutung sind. Alles deutet darauf hin, dass sich dieser Stamm an den menschlichen Darm angepasst hat.[62] Bakterien des Serotyps O103:H2 (Stamm 12009) wurden 2001 in Japan von einem Patienten mit sporadisch auftretendem blutigen Durchfall isoliert. Offenbar sind EHEC-Stämme mit den gleichen Pathotypen von verschiedener Abstammung unabhängig voneinander durch Lambda-Phagen, Inserationelemente und Virulenzplasmide entstanden.[63]
Crooks (ATCC 8739 / DSM 1576) ist ein fäkaler Stamm, der dazu verwendet wird, die Effizienz antimikrobieller Wirkstoffe zu testen. Er besitzt ein Insertionselement innerhalb von ompC und kann daher nur ompF als äußeres Membranporin exprimieren.[64] Der Stamm B dient als Forschungsmodell für Phagensensitivität, Restriktionsmodifikationssysteme und bakterielle Evolution. Ihm fehlen Proteasen. Zudem produziert er wenig Acetat, wenn ihm viel Glucose angeboten wird. Aufgrund einer einfachen Zelloberfläche ist die selektive Permeabilität verbessert. Daher wird er gerne zur rekombinanten Proteinexpression im Labormaßstab und in industriellen Dimensionen eingesetzt.[65] Der Stamm K12 von MC4100 ist sehr häufig eingesetzt. Eine Genomsequenzierung zeigte, dass er während seiner Anpassung an die Laborumgebung zusätzliche Unterschiede entwickelt hat, die nicht künstlich herbeigeführt wurden.[66] Der Stamm K12 wurde 1922 von einem Patienten mit Diphtherie isoliert und 1925 in die Stammsammlung von Stanford überführt. Da er prototroph und in definiertem Medium mit kurzen Generationszeiten einfach zu züchten ist, wurde er schnell zum bestuntersuchten Organismus und seit den 1950ern zum Verstehen zahlreicher fundamentaler biochemischer und molekularer Prozesse herangezogen. Zudem besitzt er die unter E-coli-Wildtypen seltene Fähigkeit zur Rekombination.[67] Der Stamm DH10B ist ein Derivat von K12, das aufgrund der leichten Transformation insbesondere großer Plasmide (nützlich bei der Genomsequenzierung) vielfach eingesetzt wird. Die Sequenz wurde aus „Kontaminationssequenzen“ bei der Genomsequenzierung des Rindes zusammengesetzt.[68]
Der Serotyp O9:H4 (Stamm HS) wurde von einem Laborwissenschaftler des Walter-Reed-Militärkrankenhauses isoliert. Im Humanexperiment zeigte sich, dass der Stamm HS den Gastrointestinaltrakt kolonisiert, aber keine Krankheitsbilder verursacht.[69] Der Stamm UMN026 ist ein extraintestinal pathogener Stamm (ExPEC), der 1999 in den USA von einem Patienten mit akuter Cystitis isoliert wurde. Er gehört zur phylogenetischen Gruppe D und besitzt den Serotyp O17:K52:H18. Er ist Repräsentant einer Gruppe resistenter Erreger.[70]
Der Serotyp O104:H4 Klon HUSEC041 (Sequenztyp 678) enthält Virulenzfaktoren, die sowohl für STEC als auch für EaggEC typisch sind. Im Zusammenhang mit der HUS-Epidemie 2011 in Norddeutschland wird dieser Hybrid für die besonders schwere Verlaufsform verantwortlich gemacht.[71]
Seit 1988 führt Richard Lenski ein Langzeitexperiment über die Evolution von E. coli durch. Jeden Tag werden die Kulturen in ein frisches Medium überimpft und tiefgefroren. So können neue besser angepasste Stämme wieder gegen ihre Vorfahren antreten und geprüft werden, ob sie sich besser an ihre Erlenmeyerkolbenumgebung angepasst haben. Es werden parallel die Veränderungen im Genom ermittelt, wobei die Innovationsrate kontinuierlich abnahm, je besser sich die Kulturen angepasst hatten. Es wurden auch Parallelversuche mit Myxococcus xanthus gemacht, das E. coli jagt und frisst, sowie Temperatur und Medium variiert. Unter konstanten Bedingungen ohne Fressfeinde und nur einem Zucker (Glucose) wurde dem Nährmedium auch Citrat zugegeben, obwohl E. coli dieses nicht verwerten kann. Im Jahr 2003 dominierte plötzlich ein Citrat-verwertender Mutant, der nachweislich von der Ursprungskultur abstammte.[72]
Mittels gentechnisch veränderter E. coli ist es möglich, Biosensoren für Schwermetalle wie Arsen herzustellen. Die natürlichen Mechanismen werden hierbei mit Reportergenen wie β-Galactosidase, bakteriellen Luciferasen (lux) oder dem Grün fluoreszierendem Protein (GFP) gekoppelt. Auf diese Weise ist es möglich, kostengünstig Arsenite und Arsenate unterhalb eines Mikrogramms zu detektieren.[73]
Die Kombination von ringförmiger separat in der Bakterienzelle vorliegender DNA, den sogenannten Plasmiden, mit der Entdeckung des Restriktionsenzyms EcoRI, das doppelsträngige DNA spezifisch schneidet und identische überstehende Enden zurücklässt, war die Geburtsstunde der Gentechnik. Um rekombinante DNA (künstlich hergestellte DNA) zu erhalten, wird bei einer typischen Klonierung zunächst das DNA-Fragment der Wahl und anschließend die plasmidische DNA eines so genannten Klonierungsvektors mit Restriktionsenzymen geschnitten. Durch eine Ligation werden die Enden des DNA-Fragmentes und des Plasmides zusammengefügt und anschließend in E. coli transformiert. Auf diese Weise können beispielsweise artfremde Gene in E. coli eingebracht werden, sodass dieses die auf der DNA codierten fremden Proteine synthetisieren kann.[74] Aus E. coli wird auch das Restriktionsenzym EcoRV verwendet.
Moderne künstlich hergestellte Plasmide (Vektoren oder „Genfähren“) wie pUC19 enthalten zusätzliche Antibiotika-Resistenzgene um Bakterienzellen ohne einklonierte Plasmide auf Selektivnährböden abzutöten. Eine weitere Möglichkeit zur Selektion transformierter Kolonien ist das blue-white-screening. Das Gen lacZ codiert das Protein β-Galactosidase, welches in Anwesenheit von IPTG synthetisiert wird und das künstliche Glycosid X-Gal in einen blauen Farbstoff spaltet. Nicht-transformierte Zellen erscheinen also blau auf einen Nährboden mit X-Gal. Schneidet ein Restriktionsenzym innerhalb des lacZ-Gens und wird erfolgreich ein Fremdgen eingebracht, so wird die Sequenz zur Expression von β-Galactosidase dermaßen zerstört, dass keine α-Komplementation mehr möglich ist. Erfolgreich transformierte Zellen erscheinen also weiß. Das lacZ dient hierbei somit als Reportergen. Der Test funktioniert nur mit der Deletions-Mutante lacZΔM15, deren defekte β-Galactosidase auf die α-Komplementation angewiesen ist.[75]
Zwingt man E. coli zur Überexpression von heterologen Proteinen, kann es zu Problemen bei der Proteinfaltung kommen. Es kommt zur Anhäufung fehlgefalteter und somit biologisch inaktiver Proteine (Einschlusskörperchen). Eine Strategie um die Ausbeute an löslichem Protein zu steigern, ist die Kultivierung bei niedrigen Temperaturen. Hierfür koexprimiert man im mesophilen E. coli die Faltungshelferproteine (Chaperonine) Cpn10 and Cpn60 aus dem psychrophilen Bakterium Oleispira antarctica, die bei 4–12 °C arbeiten. Sie werden unter dem Markennamen ArcticExpress von der Firma Agilent vertrieben. Zusätzlich werden Stämme angeboten, die weitere tRNAs besitzen um dem limitierenden Codon Bias bei der Translation DNA fremder Organismen in rekombinante Proteine entgegenzuwirken.[76]
E. coli XL1-Red ist ein Stamm, der in der Molekularbiologie und Gentechnik zur ungerichteten Mutagenese genutzt wird. Durch Defekte im DNA-Reparaturmechanismus zeigt der Stamm eine 5000-fache Mutationsrate gegenüber dem Wildtyp jedoch auch eine deutlich geringere Wachstumsrate (Verdopplung alle 90 min. bei 37 °C). Die Defekte im Reparaturmechanismus der DNA-Vervielfältigung gehen auf drei Mutationen der genomischen DNA zurück. Das Gen MutS enthält Mutationen der DNA-Mismatch-Reparaturproteine. Durch die Mutation erfolgt keine Fehlbasenreparatur nach der DNA-Replikation. MutD löst einen Defekt der 3'-5'-Exonukleaseaktivität der DNA-Polymerase III aus. MutT ist für die Unfähigkeit zur Hydrolyse des Basenanalogons oxo-dGTP verantwortlich.[77] Zur Mutationsauslösung werden also weder Mutagene noch Karzinogene benötigt. Die Generierung von Genmutationen ist ein klassisches Mittel in der molekularbiologischen Forschung, um generelle oder Teilfunktionen des entsprechenden Gens zu charakterisieren.
Die erste kommerzielle biotechnologische Anwendung war die Produktion des menschlichen Hormons Somatostatin von der Firma Genentech mittels genetisch veränderten E. coli. Die großtechnische Herstellung von Insulin und Wachstumshormonen folgte kurz darauf.[74] Das derart hergestellte Insulinpräparat wird in der Behandlung von Diabetes mellitus eingesetzt. Hierfür ist E. coli besonders geeignet, da es zur Darmflora des Menschen gehört und so gut wie keine Allergien verursacht. Auch in der industriellen Herstellung von Aminosäuren, Interferon sowie weiterer Feinchemikalien, Enzyme und Arzneistoffe, werden gentechnisch veränderte E. coli Bakterien verwandt.[78] So wurden neun von 31 therapeutischen Proteinen, die im Zeitraum von 2003 bis 2006 eine Arzneimittelzulassung erhalten haben, in E. coli hergestellt.[79]
Um E. coli in biotechnologischen Anwendungen leichter handhaben zu können, züchtete ein Team um Frederick Blattner (University of Wisconsin) einen Stamm, dessen Genom gegenüber natürlich vorkommenden Varianten um ca. 15 Prozent verkleinert wurde, und der dennoch lebens- und fortpflanzungsfähig ist. Hierfür wurden zwei unterschiedliche E. coli Stämme verglichen und diejenigen Gene entfernt, die kein Homolog im jeweils anderen Stamm haben und somit entbehrlich scheinen.[80]
Die Herstellung von Biotreibstoffen aus Proteinen ist mit genetisch veränderten E. coli, die drei exogene Transaminierungs- und Desaminierungszyklen enthalten, möglich. Die Proteine werden zunächst durch Saccharomyces cerevisiae, Bacillus subtilis, Mikroalgen (zur gleichzeitigen CO2-Fixierung) oder E. coli selbst hergestellt. Die Proteine könnten auch aus Abfällen von Fermentationen, Lebensmittelverarbeitung, und Bioethanolherstellung stammen. Anschließend werden die Hydrolysate in C4- und C5-Alkohole mit 56 % der theoretischen Ausbeute umgewandelt. Der entstehende Stickstoff kann in Düngemittel umgewandelt werden.[81]
Durch Veränderungen im Stoffwechsel von E. coli können höhere Alkohole wie Isobutanol, 1-Butanol, 2-Methyl-1-butanol, 3-Methyl-1-butanol und 2-Phenylethanol aus der C-Quelle Glucose produziert werden. Hierfür wird der hochaktive Biosyntheseweg für Aminosäuren verwendet und die 2-Ketosäureintermediate zur Alkoholsynthese verwendet. Verglichen mit dem klassischen Biotreibstoff Ethanol ist die höhere Energiedichte und die niedrige Hygroskopie hervorzuheben. Zudem haben verzweigtkettige Alkohole höhere Octanzahlen.[82]
Bakterien können sich bei Nährstoffmangel gegenseitig helfen. So wurde bei genetisch veränderten Escherichia coli und Acinetobacter baylyi beobachtet, wie sich E. coli mit Acinetobacter baylyi durch bis zu 14 Mikrometer lange Nanokanäle verbunden hat, um zytoplasmatische Bestandteile auszutauschen. Die beiden Bakterien wurden so verändert, dass sie für sich notwendige Aminosäuren nicht mehr produzieren konnten, die für die andere Art notwendigen Aminosäuren aber produzierten. Darauf hin bildete E. coli Nanokanäle aus, um sich mit Acinetobacter baylyi zu verbinden und zu überleben. Unklar ist noch, ob Bakterien gezielt steuern können, an welche Zelle sie sich anheften und ob diese Verbindung parasitischer Natur ist.[83]
Im Zuge der GeneSat-1-Mission wurden am 16. Dezember 2006 E. coli mittels eines Cubesat in den Orbit befördert, um genetische Änderungen aufgrund von Strahlungen im All und der Schwerelosigkeit zu untersuchen.[84]
Escherichia coli (abgekürzt E. coli) – auch Kolibakterium genannt – ist ein gramnegatives, säurebildendes und peritrich begeißeltes Bakterium, das normalerweise im menschlichen und tierischen Darm vorkommt. Unter anderem auf Grund dessen gilt dieses Bakterium auch als Fäkalindikator. E. coli und andere fakultativ anaerobe Organismen machen etwa 1 ‰ der Darmflora aus.
Innerhalb der Familie der Enterobakterien (altgriechisch ἕντερον, lateinisch enteron „Darm“) gehört E. coli zur bedeutenden Gattung Escherichia und ist deren Typspezies. Benannt wurde es nach dem deutschen Kinderarzt Theodor Escherich, der es erstmals beschrieb. Coli ist der lateinische Genitiv von colon (zu dt. Kolon), einem Teil des Dickdarms.
Die meisten Angehörigen dieser Spezies sind nicht krankheitsauslösend, jedoch gibt es auch zahlreiche verschiedene pathogene Stämme. Es zählt zu den häufigsten Verursachern von menschlichen Infektionskrankheiten. Die Basensequenz des Genoms einiger Stämme ist vollständig aufgeklärt. Als Modellorganismus zählt es zu den am besten untersuchten Prokaryoten und nimmt in der Molekularbiologie eine wichtige Rolle als Wirtsorganismus ein. Der Nobelpreis für Physiologie oder Medizin wurde an zahlreiche Forscher, die sich mit der Biologie von E. coli beschäftigt haben, vergeben.
Escherichia coli (/ˌɛʃəˈrɪkiə ˈkoʊlaɪ/)[1][2] is a Gram-negative, facultative anaerobic, rod-shaped, coliform bacterium of the genus Escherichia that is commonly found in the lower intestine of warm-blooded organisms.[3][4] Most E. coli strains are harmless, but some serotypes such as EPEC, and ETEC are pathogenic and can cause serious food poisoning in their hosts, and are occasionally responsible for food contamination incidents that prompt product recalls.[5][6] Most strains are part of the normal microbiota of the gut and are harmless or even beneficial to humans (although these strains tend to be less studied than the pathogenic ones).[7] For example, some strains of E. coli benefit their hosts by producing vitamin K2[8] or by preventing the colonization of the intestine by pathogenic bacteria. These mutually beneficial relationships between E. coli and humans are a type of mutualistic biological relationship — where both the humans and the E. coli are benefitting each other.[9][10] E. coli is expelled into the environment within fecal matter. The bacterium grows massively in fresh fecal matter under aerobic conditions for three days, but its numbers decline slowly afterwards.[11]
E. coli and other facultative anaerobes constitute about 0.1% of gut microbiota,[12] and fecal–oral transmission is the major route through which pathogenic strains of the bacterium cause disease. Cells are able to survive outside the body for a limited amount of time, which makes them potential indicator organisms to test environmental samples for fecal contamination.[13][14] A growing body of research, though, has examined environmentally persistent E. coli which can survive for many days and grow outside a host.[15]
The bacterium can be grown and cultured easily and inexpensively in a laboratory setting, and has been intensively investigated for over 60 years. E. coli is a chemoheterotroph whose chemically defined medium must include a source of carbon and energy.[16] E. coli is the most widely studied prokaryotic model organism, and an important species in the fields of biotechnology and microbiology, where it has served as the host organism for the majority of work with recombinant DNA. Under favourable conditions, it takes as little as 20 minutes to reproduce.[17]

E. coli is a Gram-negative, facultative anaerobe, nonsporulating coliform bacterium.[18] Cells are typically rod-shaped, and are about 2.0 μm long and 0.25–1.0 μm in diameter, with a cell volume of 0.6–0.7 μm3.[19][20][21]
E. coli stains Gram-negative because its cell wall is composed of a thin peptidoglycan layer and an outer membrane. During the staining process, E. coli picks up the color of the counterstain safranin and stains pink. The outer membrane surrounding the cell wall provides a barrier to certain antibiotics such that E. coli is not damaged by penicillin.[16]
The flagella which allow the bacteria to swim have a peritrichous arrangement.[22] It also attaches and effaces to the microvilli of the intestines via an adhesion molecule known as intimin.[23]
E. coli can live on a wide variety of substrates and uses mixed acid fermentation in anaerobic conditions, producing lactate, succinate, ethanol, acetate, and carbon dioxide. Since many pathways in mixed-acid fermentation produce hydrogen gas, these pathways require the levels of hydrogen to be low, as is the case when E. coli lives together with hydrogen-consuming organisms, such as methanogens or sulphate-reducing bacteria.[24]
In addition, E. coli's metabolism can be rewired to solely use CO2 as the source of carbon for biomass production. In other words, this obligate heterotroph's metabolism can be altered to display autotrophic capabilities by heterologously expressing carbon fixation genes as well as formate dehydrogenase and conducting laboratory evolution experiments. This may be done by using formate to reduce electron carriers and supply the ATP required in anabolic pathways inside of these synthetic autotrophs.[25]
E. coli have three native glycolytic pathways: EMPP, EDP, and OPPP. The EMPP employs ten enzymatic steps to yield two pyruvates, two ATP, and two NADH per glucose molecule while OPPP serves as an oxidation route for NADPH synthesis. Although the EDP is the more thermodynamically favourable of the three pathways, E. coli do not use the EDP for glucose metabolism, relying mainly on the EMPP and the OPPP. The EDP mainly remains inactive except for during growth with gluconate.[26]
When growing in the presence of a mixture of sugars, bacteria will often consume the sugars sequentially through a process known as catabolite repression. By repressing the expression of the genes involved in metabolizing the less preferred sugars, cells will usually first consume the sugar yielding the highest growth rate, followed by the sugar yielding the next highest growth rate, and so on. In doing so the cells ensure that their limited metabolic resources are being used to maximize the rate of growth. The well-used example of this with E. coli involves the growth of the bacterium on glucose and lactose, where E. coli will consume glucose before lactose. Catabolite repression has also been observed in E. coli in the presence of other non-glucose sugars, such as arabinose and xylose, sorbitol, rhamnose, and ribose. In E. coli, glucose catabolite repression is regulated by the phosphotransferase system, a multi-protein phosphorylation cascade that couples glucose uptake and metabolism.[27]
Optimum growth of E. coli occurs at 37 °C (99 °F), but some laboratory strains can multiply at temperatures up to 49 °C (120 °F).[28] E. coli grows in a variety of defined laboratory media, such as lysogeny broth, or any medium that contains glucose, ammonium phosphate monobasic, sodium chloride, magnesium sulfate, potassium phosphate dibasic, and water. Growth can be driven by aerobic or anaerobic respiration, using a large variety of redox pairs, including the oxidation of pyruvic acid, formic acid, hydrogen, and amino acids, and the reduction of substrates such as oxygen, nitrate, fumarate, dimethyl sulfoxide, and trimethylamine N-oxide.[29] E. coli is classified as a facultative anaerobe. It uses oxygen when it is present and available. It can, however, continue to grow in the absence of oxygen using fermentation or anaerobic respiration. Respiration type is managed in part by the arc system. The ability to continue growing in the absence of oxygen is an advantage to bacteria because their survival is increased in environments where water predominates.[16]
The bacterial cell cycle is divided into three stages. The B period occurs between the completion of cell division and the beginning of DNA replication. The C period encompasses the time it takes to replicate the chromosomal DNA. The D period refers to the stage between the conclusion of DNA replication and the end of cell division.[30] The doubling rate of E. coli is higher when more nutrients are available. However, the length of the C and D periods do not change, even when the doubling time becomes less than the sum of the C and D periods. At the fastest growth rates, replication begins before the previous round of replication has completed, resulting in multiple replication forks along the DNA and overlapping cell cycles.[31]
The number of replication forks in fast growing E. coli typically follows 2n (n = 1, 2 or 3). This only happens if replication is initiated simultaneously from all origins of replications, and is referred to as synchronous replication. However, not all cells in a culture replicate synchronously. In this case cells do not have multiples of two replication forks. Replication initiation is then referred to being asynchronous.[32] However, asynchrony can be caused by mutations to for instance DnaA[32] or DnaA initiator-associating protein DiaA.[33]
Although E. coli reproduces by binary fission the two supposedly identical cells produced by cell division are functionally asymmetric with the old pole cell acting as an aging parent that repeatedly produces rejuvenated offspring.[34] When exposed to an elevated stress level, damage accumulation in an old E. coli lineage may surpass its immortality threshold so that it arrests division and becomes mortal.[35] Cellular aging is a general process, affecting prokaryotes and eukaryotes alike.[35]
E. coli and related bacteria possess the ability to transfer DNA via bacterial conjugation or transduction, which allows genetic material to spread horizontally through an existing population. The process of transduction, which uses the bacterial virus called a bacteriophage,[36] is where the spread of the gene encoding for the Shiga toxin from the Shigella bacteria to E. coli helped produce E. coli O157:H7, the Shiga toxin-producing strain of E. coli.
E. coli encompasses an enormous population of bacteria that exhibit a very high degree of both genetic and phenotypic diversity. Genome sequencing of many isolates of E. coli and related bacteria shows that a taxonomic reclassification would be desirable. However, this has not been done, largely due to its medical importance,[37] and E. coli remains one of the most diverse bacterial species: only 20% of the genes in a typical E. coli genome is shared among all strains.[38]
In fact, from the more constructive point of view, the members of genus Shigella (S. dysenteriae, S. flexneri, S. boydii, and S. sonnei) should be classified as E. coli strains, a phenomenon termed taxa in disguise.[39] Similarly, other strains of E. coli (e.g. the K-12 strain commonly used in recombinant DNA work) are sufficiently different that they would merit reclassification.
A strain is a subgroup within the species that has unique characteristics that distinguish it from other strains. These differences are often detectable only at the molecular level; however, they may result in changes to the physiology or lifecycle of the bacterium. For example, a strain may gain pathogenic capacity, the ability to use a unique carbon source, the ability to take upon a particular ecological niche, or the ability to resist antimicrobial agents. Different strains of E. coli are often host-specific, making it possible to determine the source of fecal contamination in environmental samples.[13][14] For example, knowing which E. coli strains are present in a water sample allows researchers to make assumptions about whether the contamination originated from a human, another mammal, or a bird.
A common subdivision system of E. coli, but not based on evolutionary relatedness, is by serotype, which is based on major surface antigens (O antigen: part of lipopolysaccharide layer; H: flagellin; K antigen: capsule), e.g. O157:H7).[40] It is, however, common to cite only the serogroup, i.e. the O-antigen. At present, about 190 serogroups are known.[41] The common laboratory strain has a mutation that prevents the formation of an O-antigen and is thus not typeable.
Like all lifeforms, new strains of E. coli evolve through the natural biological processes of mutation, gene duplication, and horizontal gene transfer; in particular, 18% of the genome of the laboratory strain MG1655 was horizontally acquired since the divergence from Salmonella.[42] E. coli K-12 and E. coli B strains are the most frequently used varieties for laboratory purposes. Some strains develop traits that can be harmful to a host animal. These virulent strains typically cause a bout of diarrhea that is often self-limiting in healthy adults but is frequently lethal to children in the developing world.[43] More virulent strains, such as O157:H7, cause serious illness or death in the elderly, the very young, or the immunocompromised.[43][44]
The genera Escherichia and Salmonella diverged around 102 million years ago (credibility interval: 57–176 mya), an event unrelated to the much earlier (see Synapsid) divergence of their hosts: the former being found in mammals and the latter in birds and reptiles.[45] This was followed by a split of an Escherichia ancestor into five species (E. albertii, E. coli, E. fergusonii, E. hermannii, and E. vulneris). The last E. coli ancestor split between 20 and 30 million years ago.[46]
The long-term evolution experiments using E. coli, begun by Richard Lenski in 1988, have allowed direct observation of genome evolution over more than 65,000 generations in the laboratory.[47] For instance, E. coli typically do not have the ability to grow aerobically with citrate as a carbon source, which is used as a diagnostic criterion with which to differentiate E. coli from other, closely, related bacteria such as Salmonella. In this experiment, one population of E. coli unexpectedly evolved the ability to aerobically metabolize citrate, a major evolutionary shift with some hallmarks of microbial speciation.
In the microbial world, a relationship of predation can be established similar to that observed in the animal world. Considered, it has been seen that E. coli is the prey of multiple generalist predators, such as Myxococcus xanthus. In this predator-prey relationship, a parallel evolution of both species is observed through genomic and phenotypic modifications, in the case of E. coli the modifications are modified in two aspects involved in their virulence such as mucoid production (excessive production of exoplasmic acid alginate ) and the suppression of the OmpT gene, producing in future generations a better adaptation of one of the species that is counteracted by the evolution of the other, following a co-evolutionary model demonstrated by the Red Queen hypothesis.[48]
E. coli is the type species of the genus (Escherichia) and in turn Escherichia is the type genus of the family Enterobacteriaceae, where the family name does not stem from the genus Enterobacter + "i" (sic.) + "aceae", but from "enterobacterium" + "aceae" (enterobacterium being not a genus, but an alternative trivial name to enteric bacterium).[49][50][51]
The original strain described by Escherich is believed to be lost, consequently a new type strain (neotype) was chosen as a representative: the neotype strain is U5/41T,[52] also known under the deposit names DSM 30083,[53] ATCC 11775,[54] and NCTC 9001,[55] which is pathogenic to chickens and has an O1:K1:H7 serotype.[56] However, in most studies, either O157:H7, K-12 MG1655, or K-12 W3110 were used as a representative E. coli. The genome of the type strain has only lately been sequenced.[52]
Many strains belonging to this species have been isolated and characterised. In addition to serotype (vide supra), they can be classified according to their phylogeny, i.e. the inferred evolutionary history, as shown below where the species is divided into six groups as of 2014.[57][58] Particularly the use of whole genome sequences yields highly supported phylogenies.[52] The phylogroup structure remains robust to newer methods and sequences, which sometimes adds newer groups, giving 8 or 14 as of 2023.[59][60]
The link between phylogenetic distance ("relatedness") and pathology is small,[52] e.g. the O157:H7 serotype strains, which form a clade ("an exclusive group")—group E below—are all enterohaemorragic strains (EHEC), but not all EHEC strains are closely related. In fact, four different species of Shigella are nested among E. coli strains (vide supra), while E. albertii and E. fergusonii are outside this group. Indeed, all Shigella species were placed within a single subspecies of E. coli in a phylogenomic study that included the type strain.[52] All commonly used research strains of E. coli belong to group A and are derived mainly from Clifton's K-12 strain (λ+ F+; O16) and to a lesser degree from d'Herelle's "Bacillus coli" strain (B strain; O7).
There have been multiple proposals to revise the taxonomy to match phylogeny.[52] However, all these proposals need to face the fact that Shigella remains a widely used name in medicine and find ways to reduce any confusion that can stem from renaming.[61]
Group B2E. coli SE15 (O150:H5. Commensal)
E. coli E2348/69 (O127:H6. Enteropathogenic)
E. coli ED1a O81 (Commensal)
E. coli CFT083 (O6:K2:H1. UPEC)
E. coli APEC O1 (O1:K12:H7. APEC
E. coli UTI89 O18:K1:H7. UPEC)
E. coli S88 (O45:K1. Extracellular pathogenic)
Group DE. coli UMN026 (O17:K52:H18. Extracellular pathogenic)
E. coli (O19:H34. Extracellular pathogenic)
E. coli (O7:K1. Extracellular pathogenic)
Group EE. coli EDL933 (O157:H7 EHEC)
E. coli Sakai (O157:H7 EHEC)
E. coli EC4115 (O157:H7 EHEC)
E. coli TW14359 (O157:H7 EHEC)
Shigella Group B1E. coli E24377A (O139:H28. Enterotoxigenic)
E. coli 11368 (O26:H11. EHEC)
E. coli 11128 (O111:H-. EHEC)
E. coli IAI1 O8 (Commensal)
E. coli 53638 (EIEC)
E. coli SE11 (O152:H28. Commensal)
E. coli 12009 (O103:H2. EHEC)
E. coli GOS1 (O104:H4 EAHEC) German 2011 outbreak
E. coli 55989 (O128:H2. Enteroaggressive)
Group AE. coli HS (O9:H4. Commensal)
E. coli ATCC8739 (O146. Crook's E.coli used in phage work in the 1950s)
K‑12 strain derivativesE. coli K-12 W3110 (O16. λ− F− "wild type" molecular biology strain)
E. coli K-12 DH10b (O16. high electrocompetency molecular biology strain)
E. coli K-12 DH1 (O16. high chemical competency molecular biology strain)
E. coli K-12 MG1655 (O16. λ− F− "wild type" molecular biology strain)
E. coli BW2952 (O16. competent molecular biology strain)
E. coli 101-1 (O? H?. EAEC)
B strain derivativesE. coli B REL606 (O7. high competency molecular biology strain)
E. coli BL21-DE3 (O7. expression molecular biology strain with T7 polymerase for pET system)

The first complete DNA sequence of an E. coli genome (laboratory strain K-12 derivative MG1655) was published in 1997. It is a circular DNA molecule 4.6 million base pairs in length, containing 4288 annotated protein-coding genes (organized into 2584 operons), seven ribosomal RNA (rRNA) operons, and 86 transfer RNA (tRNA) genes. Despite having been the subject of intensive genetic analysis for about 40 years, many of these genes were previously unknown. The coding density was found to be very high, with a mean distance between genes of only 118 base pairs. The genome was observed to contain a significant number of transposable genetic elements, repeat elements, cryptic prophages, and bacteriophage remnants.[62]
More than three hundred complete genomic sequences of Escherichia and Shigella species are known. The genome sequence of the type strain of E. coli was added to this collection before 2014.[52] Comparison of these sequences shows a remarkable amount of diversity; only about 20% of each genome represents sequences present in every one of the isolates, while around 80% of each genome can vary among isolates.[38] Each individual genome contains between 4,000 and 5,500 genes, but the total number of different genes among all of the sequenced E. coli strains (the pangenome) exceeds 16,000. This very large variety of component genes has been interpreted to mean that two-thirds of the E. coli pangenome originated in other species and arrived through the process of horizontal gene transfer.[63]
Genes in E. coli are usually named in accordance with the uniform nomenclature proposed by Demerec et al.[64] Gene names are 3-letter acronyms that derive from their function (when known) or mutant phenotype and are italicized. When multiple genes have the same acronym, the different genes are designated by a capital later that follows the acronym and is also italicized. For instance, recA is named after its role in homologous recombination plus the letter A. Functionally related genes are named recB, recC, recD etc. The proteins are named by uppercase acronyms, e.g. RecA, RecB, etc. When the genome of E. coli strain K-12 substr. MG1655 was sequenced, all known or predicted protein-coding genes were numbered (more or less) in their order on the genome and abbreviated by b numbers, such as b2819 (= recD). The "b" names were created after Fred Blattner, who led the genome sequence effort.[62] Another numbering system was introduced with the sequence of another E. coli K-12 substrain, W3110, which was sequenced in Japan and hence uses numbers starting by JW... (Japanese W3110), e.g. JW2787 (= recD).[65] Hence, recD = b2819 = JW2787. Note, however, that most databases have their own numbering system, e.g. the EcoGene database[66] uses EG10826 for recD. Finally, ECK numbers are specifically used for alleles in the MG1655 strain of E. coli K-12.[66] Complete lists of genes and their synonyms can be obtained from databases such as EcoGene or Uniprot.
The genome sequence of E. coli predicts 4288 protein-coding genes, of which 38 percent initially had no attributed function. Comparison with five other sequenced microbes reveals ubiquitous as well as narrowly distributed gene families; many families of similar genes within E. coli are also evident. The largest family of paralogous proteins contains 80 ABC transporters. The genome as a whole is strikingly organized with respect to the local direction of replication; guanines, oligonucleotides possibly related to replication and recombination, and most genes are so oriented. The genome also contains insertion sequence (IS) elements, phage remnants, and many other patches of unusual composition indicating genome plasticity through horizontal transfer.[62]
Several studies have experimentally investigated the proteome of E. coli. By 2006, 1,627 (38%) of the predicted proteins (open reading frames, ORFs) had been identified experimentally.[67] Mateus et al. 2020 detected 2,586 proteins with at least 2 peptides (60% of all proteins).[68]
Although much fewer bacterial proteins seem to have post-translational modifications (PTMs) compared to eukaryotic proteins, a substantial number of proteins are modified in E. coli. For instance, Potel et al. (2018) found 227 phosphoproteins of which 173 were phosphorylated on histidine. Interestingly, the majority of phosphorylated amino acids were serine (1,220 sites) with only 246 sites on histidine and 501 phosphorylated threonines and 162 tyrosines.[69]
The interactome of E. coli has been studied by affinity purification and mass spectrometry (AP/MS) and by analyzing the binary interactions among its proteins.
Protein complexes. A 2006 study purified 4,339 proteins from cultures of strain K-12 and found interacting partners for 2,667 proteins, many of which had unknown functions at the time.[70] A 2009 study found 5,993 interactions between proteins of the same E. coli strain, though these data showed little overlap with those of the 2006 publication.[71]
Binary interactions. Rajagopala et al. (2014) have carried out systematic yeast two-hybrid screens with most E. coli proteins, and found a total of 2,234 protein-protein interactions.[72] This study also integrated genetic interactions and protein structures and mapped 458 interactions within 227 protein complexes.
E. coli belongs to a group of bacteria informally known as coliforms that are found in the gastrointestinal tract of warm-blooded animals.[49] E. coli normally colonizes an infant's gastrointestinal tract within 40 hours of birth, arriving with food or water or from the individuals handling the child. In the bowel, E. coli adheres to the mucus of the large intestine. It is the primary facultative anaerobe of the human gastrointestinal tract.[73] (Facultative anaerobes are organisms that can grow in either the presence or absence of oxygen.) As long as these bacteria do not acquire genetic elements encoding for virulence factors, they remain benign commensals.[74]
Due to the low cost and speed with which it can be grown and modified in laboratory settings, E. coli is a popular expression platform for the production of recombinant proteins used in therapeutics. One advantage to using E. coli over another expression platform is that E. coli naturally does not export many proteins into the periplasm, making it easier to recover a protein of interest without cross-contamination.[75] The E. coli K-12 strains and their derivatives (DH1, DH5α, MG1655, RV308 and W3110) are the strains most widely used by the biotechnology industry.[76] Nonpathogenic E. coli strain Nissle 1917 (EcN), (Mutaflor) and E. coli O83:K24:H31 (Colinfant)[77][78]) are used as probiotic agents in medicine, mainly for the treatment of various gastrointestinal diseases,[79] including inflammatory bowel disease.[80] It is thought that the EcN strain might impede the growth of opportunistic pathogens, including Salmonella and other coliform enteropathogens, through the production of microcin proteins the production of siderophores.[81]
Most E. coli strains do not cause disease, naturally living in the gut,[82] but virulent strains can cause gastroenteritis, urinary tract infections, neonatal meningitis, hemorrhagic colitis, and Crohn's disease. Common signs and symptoms include severe abdominal cramps, diarrhea, hemorrhagic colitis, vomiting, and sometimes fever. In rarer cases, virulent strains are also responsible for bowel necrosis (tissue death) and perforation without progressing to hemolytic-uremic syndrome, peritonitis, mastitis, sepsis, and Gram-negative pneumonia. Very young children are more susceptible to develop severe illness, such as hemolytic uremic syndrome; however, healthy individuals of all ages are at risk to the severe consequences that may arise as a result of being infected with E. coli.[73][83][84][85]
Some strains of E. coli, for example O157:H7, can produce Shiga toxin (classified as a bioterrorism agent). The Shiga toxin causes inflammatory responses in target cells of the gut, leaving behind lesions which result in the bloody diarrhea that is a symptom of a Shiga toxin-producing E. coli (STEC) infection. This toxin further causes premature destruction of the red blood cells, which then clog the body's filtering system, the kidneys, in some rare cases (usually in children and the elderly) causing hemolytic-uremic syndrome (HUS), which may lead to kidney failure and even death. Signs of hemolytic uremic syndrome include decreased frequency of urination, lethargy, and paleness of cheeks and inside the lower eyelids. In 25% of HUS patients, complications of nervous system occur, which in turn causes strokes. In addition, this strain causes the buildup of fluid (since the kidneys do not work), leading to edema around the lungs, legs, and arms. This increase in fluid buildup especially around the lungs impedes the functioning of the heart, causing an increase in blood pressure.[86][84][85]
Uropathogenic E. coli (UPEC) is one of the main causes of urinary tract infections.[87] It is part of the normal microbiota in the gut and can be introduced in many ways. In particular for females, the direction of wiping after defecation (wiping back to front) can lead to fecal contamination of the urogenital orifices. Anal intercourse can also introduce this bacterium into the male urethra, and in switching from anal to vaginal intercourse, the male can also introduce UPEC to the female urogenital system.
Enterotoxigenic E. coli (ETEC) is the most common cause of traveler's diarrhea, with as many as 840 million cases worldwide in developing countries each year. The bacteria, typically transmitted through contaminated food or drinking water, adheres to the intestinal lining, where it secretes either of two types of enterotoxins, leading to watery diarrhea. The rate and severity of infections are higher among children under the age of five, including as many as 380,000 deaths annually.[88]
In May 2011, one E. coli strain, O104:H4, was the subject of a bacterial outbreak that began in Germany. Certain strains of E. coli are a major cause of foodborne illness. The outbreak started when several people in Germany were infected with enterohemorrhagic E. coli (EHEC) bacteria, leading to hemolytic-uremic syndrome (HUS), a medical emergency that requires urgent treatment. The outbreak did not only concern Germany, but also 15 other countries, including regions in North America.[89] On 30 June 2011, the German Bundesinstitut für Risikobewertung (BfR) (Federal Institute for Risk Assessment, a federal institute within the German Federal Ministry of Food, Agriculture and Consumer Protection) announced that seeds of fenugreek from Egypt were likely the cause of the EHEC outbreak.[90]
Some studies have demonstrated an absence of E. coli in the gut flora of subjects with the metabolic disorder Phenylketonuria. It is hypothesized that the absence of these normal bacterium impairs the production of the key vitamins B2 (riboflavin) and K2 (menaquinone) – vitamins which are implicated in many physiological roles in humans such as cellular and bone metabolism – and so contributes to the disorder.[91]
Carbapenem-resistant E. coli (carbapenemase-producing E. coli) that are resistant to the carbapenem class of antibiotics, considered the drugs of last resort for such infections. They are resistant because they produce an enzyme called a carbapenemase that disables the drug molecule.[92]
The time between ingesting the STEC bacteria and feeling sick is called the "incubation period". The incubation period is usually 3–4 days after the exposure, but may be as short as 1 day or as long as 10 days. The symptoms often begin slowly with mild belly pain or non-bloody diarrhea that worsens over several days. HUS, if it occurs, develops an average 7 days after the first symptoms, when the diarrhea is improving.[93]
Diagnosis of infectious diarrhea and identification of antimicrobial resistance is performed using a stool culture with subsequent antibiotic sensitivity testing. It requires a minimum of 2 days and maximum of several weeks to culture gastrointestinal pathogens. The sensitivity (true positive) and specificity (true negative) rates for stool culture vary by pathogen, although a number of human pathogens can not be cultured. For culture-positive samples, antimicrobial resistance testing takes an additional 12–24 hours to perform.
Current point of care molecular diagnostic tests can identify E. coli and antimicrobial resistance in the identified strains much faster than culture and sensitivity testing. Microarray-based platforms can identify specific pathogenic strains of E. coli and E. coli-specific AMR genes in two hours or less with high sensitivity and specificity, but the size of the test panel (i.e., total pathogens and antimicrobial resistance genes) is limited. Newer metagenomics-based infectious disease diagnostic platforms are currently being developed to overcome the various limitations of culture and all currently available molecular diagnostic technologies.
The mainstay of treatment is the assessment of dehydration and replacement of fluid and electrolytes. Administration of antibiotics has been shown to shorten the course of illness and duration of excretion of enterotoxigenic E. coli (ETEC) in adults in endemic areas and in traveller's diarrhea, though the rate of resistance to commonly used antibiotics is increasing and they are generally not recommended.[94] The antibiotic used depends upon susceptibility patterns in the particular geographical region. Currently, the antibiotics of choice are fluoroquinolones or azithromycin, with an emerging role for rifaximin. Rifaximin, a semisynthetic rifamycin derivative, is an effective and well-tolerated antibacterial for the management of adults with non-invasive traveller's diarrhea. Rifaximin was significantly more effective than placebo and no less effective than ciprofloxacin in reducing the duration of diarrhea. While rifaximin is effective in patients with E. coli-predominant traveller's diarrhea, it appears ineffective in patients infected with inflammatory or invasive enteropathogens.[95]
ETEC is the type of E. coli that most vaccine development efforts are focused on. Antibodies against the LT and major CFs of ETEC provide protection against LT-producing, ETEC-expressing homologous CFs. Oral inactivated vaccines consisting of toxin antigen and whole cells, i.e. the licensed recombinant cholera B subunit (rCTB)-WC cholera vaccine Dukoral, have been developed. There are currently no licensed vaccines for ETEC, though several are in various stages of development.[96] In different trials, the rCTB-WC cholera vaccine provided high (85–100%) short-term protection. An oral ETEC vaccine candidate consisting of rCTB and formalin inactivated E. coli bacteria expressing major CFs has been shown in clinical trials to be safe, immunogenic, and effective against severe diarrhoea in American travelers but not against ETEC diarrhoea in young children in Egypt. A modified ETEC vaccine consisting of recombinant E. coli strains over-expressing the major CFs and a more LT-like hybrid toxoid called LCTBA, are undergoing clinical testing.[97][98]
Other proven prevention methods for E. coli transmission include handwashing and improved sanitation and drinking water, as transmission occurs through fecal contamination of food and water supplies. Additionally, thoroughly cooking meat and avoiding consumption of raw, unpasteurized beverages, such as juices and milk are other proven methods for preventing E. coli. Lastly, cross-contamination of utensils and work spaces should be avoided when preparing food.[99]

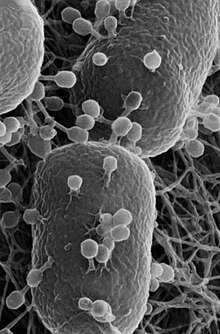
Because of its long history of laboratory culture and ease of manipulation, E. coli plays an important role in modern biological engineering and industrial microbiology.[101] The work of Stanley Norman Cohen and Herbert Boyer in E. coli, using plasmids and restriction enzymes to create recombinant DNA, became a foundation of biotechnology.[102]
E. coli is a very versatile host for the production of heterologous proteins,[103] and various protein expression systems have been developed which allow the production of recombinant proteins in E. coli. Researchers can introduce genes into the microbes using plasmids which permit high level expression of protein, and such protein may be mass-produced in industrial fermentation processes. One of the first useful applications of recombinant DNA technology was the manipulation of E. coli to produce human insulin.[104]
Many proteins previously thought difficult or impossible to be expressed in E. coli in folded form have been successfully expressed in E. coli. For example, proteins with multiple disulphide bonds may be produced in the periplasmic space or in the cytoplasm of mutants rendered sufficiently oxidizing to allow disulphide-bonds to form,[105] while proteins requiring post-translational modification such as glycosylation for stability or function have been expressed using the N-linked glycosylation system of Campylobacter jejuni engineered into E. coli.[106][107][108]
Modified E. coli cells have been used in vaccine development, bioremediation, production of biofuels,[109] lighting, and production of immobilised enzymes.[103][110]
Strain K-12 is a mutant form of E. coli that over-expresses the enzyme Alkaline Phosphatase (ALP).[111] The mutation arises due to a defect in the gene that constantly codes for the enzyme. A gene that is producing a product without any inhibition is said to have constitutive activity. This particular mutant form is used to isolate and purify the aforementioned enzyme.[111]
Strain OP50 of Escherichia coli is used for maintenance of Caenorhabditis elegans cultures.
Strain JM109 is a mutant form of E. coli that is recA and endA deficient. The strain can be utilized for blue/white screening when the cells carry the fertility factor episome.[112] Lack of recA decreases the possibility of unwanted restriction of the DNA of interest and lack of endA inhibit plasmid DNA decomposition. Thus, JM109 is useful for cloning and expression systems.
E. coli is frequently used as a model organism in microbiology studies. Cultivated strains (e.g. E. coli K12) are well-adapted to the laboratory environment, and, unlike wild-type strains, have lost their ability to thrive in the intestine. Many laboratory strains lose their ability to form biofilms.[113][114] These features protect wild-type strains from antibodies and other chemical attacks, but require a large expenditure of energy and material resources. E. coli is often used as a representative microorganism in the research of novel water treatment and sterilisation methods, including photocatalysis. By standard plate count methods, following sequential dilutions, and growth on agar gel plates, the concentration of viable organisms or CFUs (Colony Forming Units), in a known volume of treated water can be evaluated, allowing the comparative assessment of materials performance.[115]
In 1946, Joshua Lederberg and Edward Tatum first described the phenomenon known as bacterial conjugation using E. coli as a model bacterium,[116] and it remains the primary model to study conjugation.[117] E. coli was an integral part of the first experiments to understand phage genetics,[118] and early researchers, such as Seymour Benzer, used E. coli and phage T4 to understand the topography of gene structure.[119] Prior to Benzer's research, it was not known whether the gene was a linear structure, or if it had a branching pattern.[120]
E. coli was one of the first organisms to have its genome sequenced; the complete genome of E. coli K12 was published by Science in 1997.[62]
From 2002 to 2010, a team at the Hungarian Academy of Science created a strain of Escherichia coli called MDS42, which is now sold by Scarab Genomics of Madison, WI under the name of "Clean Genome E. coli",[121] where 15% of the genome of the parental strain (E. coli K-12 MG1655) were removed to aid in molecular biology efficiency, removing IS elements, pseudogenes and phages, resulting in better maintenance of plasmid-encoded toxic genes, which are often inactivated by transposons.[122][123][124] Biochemistry and replication machinery were not altered.
By evaluating the possible combination of nanotechnologies with landscape ecology, complex habitat landscapes can be generated with details at the nanoscale.[125] On such synthetic ecosystems, evolutionary experiments with E. coli have been performed to study the spatial biophysics of adaptation in an island biogeography on-chip.
In other studies, non-pathogenic E. coli has been used as a model microorganism towards understanding the effects of simulated microgravity (on Earth) on the same.[126][127]
Since 1961, scientists proposed the idea of genetic circuits used for computational tasks. Collaboration between biologists and computing scientists has allowed designing digital logic gates on the metabolism of E. coli. As Lac operon is a two-stage process, genetic regulation in the bacteria is used to realize computing functions. The process is controlled at the transcription stage of DNA into messenger RNA.[128]
Studies are being performed attempting to program E. coli to solve complicated mathematics problems, such as the Hamiltonian path problem.[129]
A computer to control protein production of E. coli within yeast cells has been developed.[130] A method has also been developed to use bacteria to behave as an LCD screen.[131][132]
In July 2017, separate experiments with E. coli published on Nature showed the potential of using living cells for computing tasks and storing information.[133] A team formed with collaborators of the Biodesign Institute at Arizona State University and Harvard's Wyss Institute for Biologically Inspired Engineering developed a biological computer inside E. coli that responded to a dozen inputs. The team called the computer "ribocomputer", as it was composed of ribonucleic acid.[134][135] Meanwhile, Harvard researchers probed that is possible to store information in bacteria after successfully archiving images and movies in the DNA of living E. coli cells.[136][137] In 2021, a team led by biophysicist Sangram Bagh realized a study with E. coli to solve 2 × 2 maze problems to probe the principle for distributed computing among cells.[138][139]
In 1885, the German-Austrian pediatrician Theodor Escherich discovered this organism in the feces of healthy individuals. He called it Bacterium coli commune because it is found in the colon. Early classifications of prokaryotes placed these in a handful of genera based on their shape and motility (at that time Ernst Haeckel's classification of bacteria in the kingdom Monera was in place).[98][140][141]
Bacterium coli was the type species of the now invalid genus Bacterium when it was revealed that the former type species ("Bacterium triloculare") was missing.[142] Following a revision of Bacterium, it was reclassified as Bacillus coli by Migula in 1895[143] and later reclassified in the newly created genus Escherichia, named after its original discoverer, by Aldo Castellani and Albert John Chalmers.[144]
In 1996, the world's worst to date outbreak of E. coli food poisoning occurred in Wishaw, Scotland, killing 21 people.[145][146] This death toll was exceeded in 2011, when the 2011 Germany E. coli O104:H4 outbreak, linked to organic fenugreek sprouts, killed 53 people.
E. coli has several practical uses besides its use as a vector for genetic experiments and processes. For example, E. coli can be used to generate synthetic propane and recombinant human growth hormone.[147][148]
Escherichia coli (/ˌɛʃəˈrɪkiə ˈkoʊlaɪ/) is a Gram-negative, facultative anaerobic, rod-shaped, coliform bacterium of the genus Escherichia that is commonly found in the lower intestine of warm-blooded organisms. Most E. coli strains are harmless, but some serotypes such as EPEC, and ETEC are pathogenic and can cause serious food poisoning in their hosts, and are occasionally responsible for food contamination incidents that prompt product recalls. Most strains are part of the normal microbiota of the gut and are harmless or even beneficial to humans (although these strains tend to be less studied than the pathogenic ones). For example, some strains of E. coli benefit their hosts by producing vitamin K2 or by preventing the colonization of the intestine by pathogenic bacteria. These mutually beneficial relationships between E. coli and humans are a type of mutualistic biological relationship — where both the humans and the E. coli are benefitting each other. E. coli is expelled into the environment within fecal matter. The bacterium grows massively in fresh fecal matter under aerobic conditions for three days, but its numbers decline slowly afterwards.
E. coli and other facultative anaerobes constitute about 0.1% of gut microbiota, and fecal–oral transmission is the major route through which pathogenic strains of the bacterium cause disease. Cells are able to survive outside the body for a limited amount of time, which makes them potential indicator organisms to test environmental samples for fecal contamination. A growing body of research, though, has examined environmentally persistent E. coli which can survive for many days and grow outside a host.
The bacterium can be grown and cultured easily and inexpensively in a laboratory setting, and has been intensively investigated for over 60 years. E. coli is a chemoheterotroph whose chemically defined medium must include a source of carbon and energy. E. coli is the most widely studied prokaryotic model organism, and an important species in the fields of biotechnology and microbiology, where it has served as the host organism for the majority of work with recombinant DNA. Under favourable conditions, it takes as little as 20 minutes to reproduce.
Escherichia coli (pronunciado /eske'rikia 'koli/) es una bacteria miembro de la familia de las enterobacterias y forma parte de la microbiota del tracto gastrointestinal de animales homeotermos, como por ejemplo el ser humano.[3] Es un bacilo gramnegativo, no exigente, oxidasa negativo, catalasa positivo, anaerobio facultativo, cuya temperatura de crecimiento preferente es a 37 °C (mesófilo), fimbriado y comúnmente es móvil por flagelos perítricos.[4][5]
E. coli es la bacteria anaerobia facultativa comensal más abundante de la microbiota del tracto gastrointestinal en donde junto con otros microorganismos es esencial para el funcionamiento correcto del proceso digestivo,[cita requerida] E. coli además participa en la producción de las vitaminas B y K.[6] Sin embargo, se han descrito diferentes clonas que por procesos de patoadaptación, han adquirido elementos genéticos o mutaciones que funcionan como factores de virulencia y aptitud, estos determinan la patogenicidad y virulencia de las cepas en diferentes tipo de enfermedades.[7] Las cepas asociadas a las infecciones gastrointestinales, son denominadas E. coli Diarreogénicas o Intestinales, mientras que las asociadas a infecciones en otros aparatos y sistemas, como por ejemplo, el tracto urinario, sanguíneo y nervioso, son denominadas E. coli Patogénicas Extraintestinales (ExPEC). La alta morbilidad, así como la variedad en síndromes y cuadros clínicos asociados a las infecciones por E. coli, hacen de esta bacteria uno de patógenos más versátiles y de gran relevancia para el ser humano.
Escherichia coli es un organismo modelo utilizado frecuentemente en el laboratorio por su velocidad de crecimiento, pocos requerimientos nutricionales y a su amplia bibliografía. Además, E. coli es usada en experimentos de genética y biología molecular debido a que la estructura de su genoma es altamente flexible, permitiendo la movilidad de material genético por medio de transposones, secuencias de inserción, bacteriófagos y plásmidos.[4][8]
Fue descrita por primera vez en 1885 por Theodore von Escherich, bacteriólogo alemán, quien la denominó Bacterium coli commune. Posteriormente la taxonomía le adjudicó el nombre de Escherichia coli, en honor a su descubridor.[3][4][9]
A pesar de que ambas compartan abreviatura, no se debe confundir la bacteria Escherichia coli con la ameba Entamoeba coli (E. coli).
Theodore von Escherich, pediatra y microbiólogo alemán, estudió en el año 1884 los microbios que se encontraban en el intestino infantil, a partir de sus heces, para establecer su rol en la digestión y la enfermedad. En el año 1885 publicó los resultados de su estudio, en cuyo reporte describe el aislamiento y caracterización de bacterias de rápido crecimiento con forma de varas cortas y delgadas que llamó Bacterium coli commune. Aunque posteriormente el microorganismo fue descrito por varios otros investigadores con múltiples sinónimos, Escherich fue reconocido como el primero estableciéndose el nombre definitivo del microbio como Escherichia coli en 1954.[1][4][10]
En 1933, Alfred Adam mostró que ciertos serotipos de «dyspepsie koli» (como le llamaba a las cepas de E. coli diarreogénicas) estaban implicados en epidemias de diarrea pediátrica. En 1944, Kauffman propuso un esquema de clasificación que aún se usa con el fin de diferenciar los tipos comensales de los patógenos y subclasificarlos.[11] [12] [13]
Escherichia coli, en su hábitat natural, vive en los intestinos de la mayor parte de los mamíferos sanos. Es el principal organismo anaerobio facultativo del sistema digestivo. En individuos sanos, es decir, si la bacteria no adquiere elementos genéticos que codifican factores virulentos, la bacteria actúa como un comensal formando parte de la microbiota intestinal y ayudando así a la absorción de nutrientes. En humanos, Escherichia coli coloniza el tracto gastrointestinal de un neonato adhiriéndose a las mucosidades del intestino grueso dentro de pocas horas de nacido. Desde entonces permanece en una relación de mutuo beneficio. No obstante, estas cepas comensales pueden producir infecciones en el paciente inmunodeprimido. Las cepas patógenas de E. coli, por el contrario, en cuanto colonizan un huésped sano, pueden producir infecciones de diversa severidad en el intestino, las vías urinarias, meningitis, sepsis, entre otras infecciones.[11]
E. coli se puede aislar e identificar a partir de una muestra si se siembra en medios selectivos bajo condiciones de esterilidad aeróbicas. Los medios selectivos de elección son, por ejemplo, el agar MacConkey, en donde forman colonias rosas (lactosa positivas), opacas, circulares, de 2 a 4 mm de diámetro, convexas, de bordes enteros y suaves, y el agar eosina azul de metileno (EMB) donde forma colonias negras verde metálico, opacas, brillantes, con un halo de precipitado de bilis, las colonias aisladas oxidasa negativas, catalasa positivas deben ser evaluadas por pruebas bioquímicas.
En un frotis teñido por la técnica de Gram, E. coli se presenta como bacilos gramnegativos, con la tinción de verde de malaquita se observa que no forma esporas, es un anareobio facultativo, que fermenta la glucosa y la lactosa, con la producción de ácido y gas a las veinticuatro horas, reduce los nitratos a nitritos, descarboxila la L-ornitina y su prueba de IMViC es ++--.[5]
En el mundo microbiano se puede establecer una relación de depredación similar a la que observamos en el mundo animal. Recientemente, se ha observado que E. coli es presa de múltiples depredadores generalistas, como puede ser Myxococcus xanthus. En esta relación depredador-presa se observa una evolución paralela de ambas especies mediante modificaciones genómicas y fenotípicas, en el caso de E. coli dichas modificaciones se producen en dos rasgos implicados en su virulencia como son la producción de mucoidía (excesiva producción del exopolosicárido alginato) y la supresión del gen OmpT, produciendo en las sucesivas generaciones una mejor adaptación de una de las especies que es contrarrestada por la evolución de la otra, siguiendo un modelo co-evolutivo denominado hipótesis de la Reina Roja.[14]
Es miembro de la familia enterobacteria y estas afectan al tracto gastrointestinal (Aparato Digestivo)
La clasificación de las cepas de E. coli, se realizaba comúnmente mediante la identificación serológica según el esquema de Kauffman, basada en el uso de anticuerpos, que determinan las variaciones naturales en la composición de los antígenos somático "O" determinado por el lipopolisacárido, LPS, el flagelar "H" determinado por las proteína fliC del flagelo y capsular "K" determinado por la envoltura y la cápsula, termoestables y termolábiles de la bacteria. Alrededor de ciento ochenta y seis tipos diferentes de antígeno O y cincuenta y tres antígenos H han sido reconocidos. La presencia de antígenos capsulares "K" se demuestra en las cepas que no se aglutinan por el antisuero O solo hasta que se calientan. Posterior a Kauffman, se precisó la definición de antígenos K solo para los polisacáridos acídicos, dejando fuera los antígenos proteicos de las fimbrias, que se denominan antígenos F. Los serotipos de E. coli son determinados por combinaciones específicas de los antígenos O y H.[13][11][15]
La fagotipificación, con líneas de bacteriófagos específicos era utilizada como una metodología complementaria a la serotipificación y tuvieron un papel relevante en la epidemiología y la clasificación de las clonas patógenas de E. coli.
Los avances y el uso generalizado actual de las técnicas de la biología molecular, han permitido el desarrollo de nuevas aproximaciones a la tipificación. Clermont y colaboradores en el año 2000[16] propusieron una metodología para la clasificación de E. coli mediante ensayos de PCR múltiple para la amplificación de secuencias específicas utilizando oligonucleótidos alelo específicos, siendo una metodología ampliamente utilizada en estudios epidemiológicos. En esta clasificación se propone la división de las cepas de E. coli en siete filogrupos A, B1, B2, C, D, E y F, que interesantemente, parecen estar relacionados con su estilo de vida y a su tipo de hospedero. Los genes arpA, chuA y yjA, y el fragmento críptico TSPE4.C2 son amplificados, esto permite determinar los filogrupos A, B1, B2 y F. Se requieren la amplificación adicional de las secuencias Gp.E y Gp.C, para determinar los filogrupos E y C respectivamente y siendo complementaria para la determinación del filogrupo D.[17]
Las técnicas para la secuenciación del genoma completo han permitido que se encuentren disponibles genomas de E. coli de diferentes filogrupos y patotipos, permitiendo establecer un genoma núcleo (core en inglés) de E. coli. Esto permitió comparar los genomas de cepas patógenas contra el de las comensales, encontrando que en promedio, el genoma de las cepas patógenas contiene un millón más de pares de bases más, dentro de estas regiones se han localizado las secuencias asociadas a factores de virulencia, resistencia y aptitud que contribuyen a la patogenicidad de E. coli.
Las cepas patogénicas de E. coli productoras de diarrea en el hombre se han clasificado según el factor genético que define su virulencia y que contribuye al cuadro clínico asociado, definiendo a diferentes tipos patogénicos, conocidos como patotipos o virotipos; de esta forma se han definido al menos siete tipos de patotipos intestinales son: E. coli enteropatogénica (EPEC), E. coli enterotoxigénica (ETEC), E. coli enteroinvasiva (EIEC), E. coli enteroagregativa (EAEC), E. coli productora de toxina Shiga (STEC), E. coli de adherencia difusa (ADEC) y E. coli enterohemorrágica (EHEC). El patotipo EIEC se ha asociado con la enfermedad de Crohn, mientras que el patotipo DAEC se asocia con colitis ulcerativa.[18] Hay tipos híbridos, como la E. coli enteroagregativa hemorrágica (ECEAH) que lleva genes de virulencia de la EAEC y la ECST. Las cepas de E. coli son altamente heterogéneas y continuaran adaptándose.[15][3]
La cepa capaz de provocar enfermedad fuera del tracto intestinal, se denomina E. coli patógena extraintestinal (ExPEC), son responsables de una variedad de enfermedades, la más común es la infección de vías urinarias, causadas por cepas de E. coli uropatogénica (UPEC), pero pueden colonizar el tracto respiratorio, el sistema nervioso central (E. coli asociada a meningitis neonatal, NMEC) y otras infecciones ya sea generalizadas en bacteriemias, o en diversas ubicaciones (articulaciones, globos oculares, glándulas, peritoneo, hígado, hueso, cerebro, corazón, próstata, venas y otros). Estas cepas pueden ser portadas por animales, tales como las aves de crianza, cerdos, ganado vacuno, animales de compañía, de tal forma que la enfermedad extraintestinal puede ser adquirida por el contacto con estos animales o sus subproductos. Una cepa en particular, produce brotes de enfermedad relevantes en la industria de las aves y se denomina E. coli patógena aviar (APEC). Todas estos tipos, comparten diversos factores de virulencia que les permiten adherirse a las células infectadas (p. ej., fimbrias tipo 1 y fimbrias tipo P), factores que les permiten evitar o sobrevivir a los sistemas de defensa del huésped (como cápsulas y lipopolisacáridos), mecanismos de adquisición de nutrientes (sideróforos), proteasas, invasinas y toxinas (hemolisina y factor citotóxico necrosante).[15][19] [20]
La E. coli enteropatogénica, ECEP, se caracteriza por adherirse a la mucosa del intestino, produciendo lesiones distintivas en dicho epitelio. El término se usó por primera vez en 1955 en relación con una serie de brotes de diarrea infantil en los años 1940 y 1950, para diferenciarla de la E. coli comensal.[4][21]
Esta cepa causa diarrea en humanos, monos, conejos, perros y ovejas, al igual que la enterotoxigénica, pero la etiología y los mecanismos moleculares de colonización son diferentes. No produce las toxinas termoestables (ST) ni termolábiles (LT), pero utilizan la proteína intimina, una adhesina, para adherirse a las células intestinales. Produce una lesión característica denominada «adhesión y eliminación», destruyendo las microvellosidades intestinales en el lugar donde la bacteria se adhiere al epitelio intestinal, el cual toma la forma de "pedestales" para las colonias.[4][11]
La ECEP se subclasifica en típica y atípica, según la presencia o no del plásmido del factor de adherencia de E. coli (FAE). Este plásmido codifica la formación de fimbrias tipo IV conocidas como «pelos formadores de penachos» (en inglés: "bundle-forming pilus" o BFP).[4][22]
Las fases del proceso patogénico de la ECEP son: adherencia inicial, inyección de factores y transducción de señales, y contacto íntimo.
La adherencia de la ECEP comienza con una adherencia entre las mismas bacterias, seguido por una adherencia al epitelio intestinal. Para que este fenómeno se lleve a cabo, es necesaria la presencia de dos factores de virulencia: los pelos formadores de penachos y el flagelo. Los pelos permiten a las ECEP unirse entre sí formando una microcolonia. El flagelo permite estrechar el contacto entre las bacterias y los enterocitos.[11][21]
Completada la fase anterior, la ECEP inyecta al enterocito una serie de proteínas mediante el sistema de secreción tipo III (SSTT). Este sistema funciona como una "jeringa molecular" y se conoce como «inyectisoma» o «complejo aguja». Por este sistema se inyectan en el enterocito las proteínas efectoras que producirán los efectos necesarios para continuar con la infección. La mayor parte de estas proteínas está codificada en el «locus de la eliminación del enterocito» o LEE del cromosoma de la ECEP.[21][23] [24]
En esta etapa se estructuran los pedestales en el epitelio intestinal, por debajo de las bacterias. Las microvellosidades se destruyen y la función normal del enterocito cesa. Estos pedestales contienen altas concentraciones de actina filamentosa polimerizada o actina F, además de actinina alfa, talina, ezrina y cadenas livianas de miosina.[21][11]
La consecuencia principal de la infección por la NBA es la diarrea secundaria a la alteración en la absorción y secreción de iones y solutos a lo largo del epitelio intestinal, que son acompañadas de movimiento de agua. La pérdida de las microvellosidades disminuye al área de intercambio contribuyendo a la diarrea. Además se suma el aumento de permeabilidad y la inflamación.[23]
La ECET se caracteriza por producir al menos una de dos toxinas: la enterotoxina resistente al calor o ST y la enterotoxina termolábil o LT. Se descubrió primero en cerdos donde la infección sigue siendo letal en los recién nacidos.[11]
La toxina LT se parece mucho, tanto en estructura como en función, a la enterotoxina producida por el Vibrio cholerae. Se adhiere a los enterocitos, siendo endocitada y traslocada al interior de la célula. Produce una activación permanente de la adenilil ciclasa, aumentando la concentración de AMP cíclico intracelular lo cual activa a la proteína quinasa dependiente de cAMP (PKA) generando una fosforilación por sobre lo normal de los canales de calcio ubicados en la cara apical de los enterocitos. El resultado de esto es el aumento de secreción de calcio por las células criptales y la inhibición de la reabsorción de cloruro de sodio por las microvellosidades. El aumento neto de iones en el intestino genera un arrastre pasivo de agua.[11]
La toxina ST que produce la ECET es la misma ST producida en las infecciones por Yersinia enterocolítica y Vibrio cholerae no O1. En la cara apical de los enterocitos existe un receptor para esta toxina, llamado guanilil ciclasa C (GC-C). Su activación genera un aumento en el GMP cíclico lo que produce un aumento en la secreción de cloro y una inhibición en la absorción de cloruro de sodio, contribuyendo de esta forma a la producción de diarrea. Otro tipo de ST presente en algunas ECET, produce daño directo a los enterocitos y aumentando la secreción de bicarbonato.[11]
La ECET se adhiere y coloniza la mucosa del intestino delgado gracias a fimbrias o pili de distinto tipo, que le permiten adherirse a los enterocitos, y luego secretar las toxinas descritas.[11]
El cuadro clínico en humanos puede ser una diarrea infantil o la diarrea del viajero, especialmente en temporada cálida y húmeda. Comienza de forma abrupta tras un corto período de incubación (14 a 50 horas). Produce diarrea acuosa no sanguinolenta en niños y adultos, sobre todo en países en vías de desarrollo, aunque los desarrollados también se ven afectados. Generalmente es leve y autolimitada, pero puede llegar a ser letal en niños pequeños, si la hidratación no compensa las grandes pérdidas hídricas que puede producir la ECET.[11]
Es inmóvil, no fermenta la lactosa. Invade el epitelio intestinal causando diarrea sanguinolenta en niños y adultos. Libera el calcio en grandes cantidades impidiendo la solidificación ósea, produciendo artritis y en algunos casos arterioesclerosis. Es una de las E. coli que causa más daño debido a la invasión que produce en el epitelio intestinal.
La convención internacional de nomenclatura de patógenos ha recomendado el uso de STEC (Shiga Toxin Escherichia coli) para este grupo, debido a que estas bacterias producen una toxina citotóxica para células Vero de cultivo de similitud estructural a la toxina producida por Shigella dysenteriae.
Este virotipo posee una serie de factores de virulencia que son similares a los que se encuentran en Shigella, como la toxina shiga.[25][26]
Las STEC producen verotoxinas que actúan en el colon. Sus síntomas son: primero colitis hemorrágica, luego síndrome urémico hemolítico (lo anterior más afección del riñón, posible entrada en coma y muerte), y por último, púrpura trombocitopénica trombótica (lo de antes más afección del sistema nervioso central). Esta cepa no fermenta el sorbitol y posee un fago, donde se encuentran codificadas las verotoxinas, también llamadas «Toxinas Shiga», no posee una fimbria formadora de mechones, en vez de esto posee una fimbria polar larga que usa para adherencia.
Solo encontrada en humanos. Son llamadas enteroagregativas porque tienen fimbrias con las que aglutinan células en los cultivos de tejidos. Se unen a la mucosa intestinal causando diarrea acuosa sin fiebre. No son invasivas. Producen hemolisina y una enterotoxina ST similar a la de las enterotoxigénicas. se le asocian dos toxinas:
Se adhiere a la totalidad de la superficie de las células epiteliales y habitualmente causa enfermedad en niños inmunológicamente no desarrollados o malnutridos. No se ha demostrado que pueda causar diarrea en niños mayores de un año de edad, ni en adultos y ancianos.
La Escherichia coli O157:H7 es una de los cientos de cepas de Escherichia coli. Aunque la mayoría de las cepas son inocuas y viven en los intestinos de los seres humanos y animales saludables, esta cepa produce una potente toxina y puede ocasionar enfermedades graves como el síndrome urémico hemolítico.
Escherichia coli O157:H7 fue reconocida inicialmente como causa de enfermedad en 1982 durante un brote de diarrea aguda con sangre en Estados Unidos Se determinó que el brote se debía a hamburguesas contaminadas. Desde entonces, la mayoría de las infecciones han provenido de comer carne de vacuno picada insuficientemente cocinada. El escritor Robin Cook escribió una novela sobre el tema titulada Toxina.
En 1996, cerca de Seattle se produjo un brote a causa de esta bacteria, que se encontró en botellas de jugo de manzana de la marca Odwalla Inc.. Muchas personas, entre ellas bebés y niños, murieron después de tomar este jugo de frutas La bacteria entró en las botellas porque las manzanas que se exprimieron contenían excrementos de venados de la zona y no hubo ningún tipo de pasteurización.
Se diferencia de las otras Escherichia coli en que no fermenta el sorbitol, no crece a 44 °C y no produce β-glucoronidasa.
La diarrea producida por cepas patogénicas de E. coli es una causa importante de muerte en niños menores de 5 años, especialmente en las regiones de África subsahariana y Asia del Sur donde es una de las cuatro causas más importantes de diarrea moderada y severa, potencialmente letales. Un incremento en la fatalidad se asocia con las cepas enteropatogénica, ECEP, y enterotoxigénica, ECET.[4]
En Alemania en el año 2011 se informó de un gran brote epidémico producido por el serotipo enterohemorrágico Escherichia coli O104:H4.[27][28] Se trata de cerca de 3255 casos, según la OMS, con 33 fallecidos.[29] Se encontraron indicios de que pudiera haber surgido en una explotación alemana de brotes de soja, pero se descartó como foco de la intoxicación.[30] Sin embargo el gobierno alemán señaló en principio a España como origen de los productos vegetales implicados en la intoxicación alimentaria, dando dos fuentes de producción en Málaga y Almería. Países Bajos es otro país señalado. Tras el análisis de las heces de los pacientes, se descartó el origen español.[31] Pero se desconoce el origen de la bacteria en toda la cadena hasta el consumidor, desde la producción, el transporte y la distribución, por lo que no se puede descartar la manipulación de productos en Alemania, que fue señalada como el país de origen de la intoxicación ocurrida en viajeros que tocaron suelo alemán. No se ha descrito jamás ningún caso en España de una infección por la bacteria implicada en el brote alemán. En España existe un posible caso de infección por el consumo de verduras frescas en Alemania.[32][33] La cepa de la bacteria E. coli aparecida en Alemania es resistente a ocho tipos distintos de antibiótico, según un equipo de investigadores chinos, que lograron secuenciar el genoma del organismo. Los investigadores del Instituto de Genómica de Pekín (BGI) descartaron antibióticos como la penicilina, las sulfamidas, la cefalotina o la estreptomicina como posible solución a la enfermedad debido a los genes que la hacen resistente.[34]
Escherichia coli puede causar infecciones intestinales y extra intestinales generalmente graves, tales como infecciones del aparato excretor, vías urinarias, cistitis, Uretritis, meningitis, peritonitis, mastitis, septicemia y neumonía Gram-negativa.
Escherichia coli está dividida por sus propiedades virulentas, pudiendo causar diarrea en humanos y otros animales. Otras cepas causan diarreas hemorrágicas por virtud de su agresividad, patogenicidad y toxicidad. En muchos países ya hubo casos de muerte por esta bacteria. Generalmente le pasa a niños entre uno y ocho años. Causado generalmente por la contaminación de alimentos, y posterior mala cocción de los mismos, es decir, a temperaturas internas y externas menores de 70 °C.
Son más comunes en mujeres por la corta longitud de la uretra (25 a 50 mm), en comparación con los hombres (unos 15 cm). Entre los ancianos, las infecciones urinarias tienden a ser de la misma proporción entre hombres y mujeres. Debido a que la bacteria invariablemente entra al tracto urinario por la uretra (una infección ascendente), los malos hábitos sanitarios pueden predisponer a una infección, sin embargo, otros factores cobran importancia, como el embarazo, hipertrofia benigna o maligna de próstata, y en muchos casos el evento iniciante de la infección es desconocido. Aunque las infecciones ascendentes son las causantes de infecciones del tracto urinario bajo y cistitis, no es necesariamente esta la causa de infecciones superiores como la pielonefritis, que puede tener origen hematógeno.
El uso de antibióticos es poco eficaz y casi no se prescribe. Para la diarrea se sugiere el consumo de abundante líquido y evitar la deshidratación. Cuando una persona presenta diarrea no debe ir a trabajar o asistir a lugares públicos para evitar el contagio masivo.
En algunas patologías como la pielonefritis hay que considerar el uso de alguna cefalosporina endovenosa.
|número-autores= (ayuda) Escherichia coli (pronunciado /eske'rikia 'koli/) es una bacteria miembro de la familia de las enterobacterias y forma parte de la microbiota del tracto gastrointestinal de animales homeotermos, como por ejemplo el ser humano. Es un bacilo gramnegativo, no exigente, oxidasa negativo, catalasa positivo, anaerobio facultativo, cuya temperatura de crecimiento preferente es a 37 °C (mesófilo), fimbriado y comúnmente es móvil por flagelos perítricos.
E. coli es la bacteria anaerobia facultativa comensal más abundante de la microbiota del tracto gastrointestinal en donde junto con otros microorganismos es esencial para el funcionamiento correcto del proceso digestivo,[cita requerida] E. coli además participa en la producción de las vitaminas B y K. Sin embargo, se han descrito diferentes clonas que por procesos de patoadaptación, han adquirido elementos genéticos o mutaciones que funcionan como factores de virulencia y aptitud, estos determinan la patogenicidad y virulencia de las cepas en diferentes tipo de enfermedades. Las cepas asociadas a las infecciones gastrointestinales, son denominadas E. coli Diarreogénicas o Intestinales, mientras que las asociadas a infecciones en otros aparatos y sistemas, como por ejemplo, el tracto urinario, sanguíneo y nervioso, son denominadas E. coli Patogénicas Extraintestinales (ExPEC). La alta morbilidad, así como la variedad en síndromes y cuadros clínicos asociados a las infecciones por E. coli, hacen de esta bacteria uno de patógenos más versátiles y de gran relevancia para el ser humano.
Escherichia coli es un organismo modelo utilizado frecuentemente en el laboratorio por su velocidad de crecimiento, pocos requerimientos nutricionales y a su amplia bibliografía. Además, E. coli es usada en experimentos de genética y biología molecular debido a que la estructura de su genoma es altamente flexible, permitiendo la movilidad de material genético por medio de transposones, secuencias de inserción, bacteriófagos y plásmidos.
Fue descrita por primera vez en 1885 por Theodore von Escherich, bacteriólogo alemán, quien la denominó Bacterium coli commune. Posteriormente la taxonomía le adjudicó el nombre de Escherichia coli, en honor a su descubridor.
A pesar de que ambas compartan abreviatura, no se debe confundir la bacteria Escherichia coli con la ameba Entamoeba coli (E. coli).
Kolibakteeri eli Escherichia coli (E. coli) on yksi niistä bakteerilajeista, jotka elävät tasalämpöisten eläinten (muun muassa nisäkkäiden ja lintujen) ruoansulatuskanavan alaosassa. Jos pohjavedessä on kolibakteeria, se kertoo yleensä ulostesaastumisesta. Ihmisen päivittäisessä ulostemäärässä on tavallisesti 1011–1013 kolibakteeria. Bakteeri on saanut tieteellisen nimensä löytäjänsä Theodor Escherichin mukaan. E. coli kuuluu enterobakteereihin.
E. coli on sauvamainen noin kahden mikrometrin pituinen ja puolen mikrometrin levyinen. Sen soluseinä koostuu peptidoglykaaneista. Sen kaksinkertaisen solukalvon ulompi kerros on peptidoglykaanikerroksen ulkopuolella eli se on gram-negatiivinen bakteeri. Lisäksi se on fakultatiivisesti anaerobinen organismi ja luokitellaan ei-sisäitiöitä-ryhmään. E. colilla on noin 15 000 ribosomia, tuhat erilaista entsyymiä, noin kahdentuhannen mikrometrin mittainen rengasmainen DNA-molekyyli ja mahdollisesti plasmideja.
E. colin proteiineja koodaava haploidinen genomi on 4 639 221 eli noin 4,64 miljoonaa emästä pitkä, ja se koodaa 4 377 proteiinia. Ihmisessä oleva E. colin versio O157:H7 sisältää noin 5,44 miljoonaa emästä, jotka koodaavat 5 416 proteiinia.[1] Jos genomin pituudeksi oletetaan 4 miljoonaa emäsparia, genomi painaa 4,4×10−18 kg, ja genomin pituus oikaistuna on 1,36 mm[2]. Kromosomi on pitkä (noin 500 kertaa solun pituus) ja siksi voimakkaasti kiertynyt ja laskostunut.
E. coli jakautuu suotuisissa lämpötilaoloissa kerran 20 minuutissa, ja hieman keskimääräisemmissä oloissa kerran 30 minuutissa[3]. Näin E. coleja syntyisi jakautumisen kautta 2 potenssiin 48 eli yli 280 000 miljardia kappaletta vuorokaudessa, mikäli ympäristö ei tätä rajoittaisi. Monesti jakautuminen on hitaampaa, esimerkiksi kerran 108 minuutissa[4].
Joidenkin aiempien arvioiden mukaan olisi 4,64 miljoonan emäksen kolibakteerissa 4 300 geeniä, joista 180 liittyisi solun rakenteeseen, 650 liittyisi aineenvaihduntaan, 240 energia-aineenvaihduntaan, 530 geeniä toimisi DNA-jakautumisen ohjaajina, ja kuljettajaproteiinien geenejä olisi noin 240[5].
Talousveden puhdistamisen tarkkailussa ja jätteidenkäsittelyssä kolibakteeri on jo hyvin varhain valittu veden saastuneisuusasteen mittariksi. Pääasialliset syyt sen käyttöön ovat se, että ihmisulosteessa on paljon enemmän koliformisia bakteereita kuin patogeeneja (kuten lavantautia aiheuttava Salmonella typhii), koliformisia bakteereita on nopea ja edullinen analysoida sekä se, että kolibakteeri on tavallisesti vaaraton eli se ei voi ”karata käsistä” laboratoriossa ja tuottaa vahinkoa.
E. coli on hyvin yleisesti käytetty bakteeri geenitekniikassa ja synteettisessä biologiassa. Bakteeriin voidaan helposti siirtää geenejä. Geenejä voidaan esimerkiksi monistaa liittämällä niitä E. colin DNA:han, jolloin geeni monistuu samalla kun E. coli jakautuu. Kolibakteereja voidaan käyttää myös proteiinin tuottamiseen: bakteeriin liitetään halutun proteiinin geeni, jolloin bakteeri alkaa tuottaa proteiinia.
Vaarattomuudestaan huolimatta kolibakteeri voi aiheuttaa tulehduksen, jos se pääsee leviämään ruoansulatuskanavasta muualle elimistöön. Tartunta tapahtuu usein saastuneiden käsien tai käytettyjen välineiden kautta. Yleisimmin kolibakteeri aiheuttaa infektion virtsaputkeen tai vatsaonteloon, mutta sairaalaoloissa se voi levitä esimerkiksi keuhkoihin. Infektio todennetaan laboratoriokokeissa bakteeriviljelyllä, joka tehdään infektioalueelta otetusta näytteestä (esimerkiksi virtsanäyte tai keuhkoista yskös- tai imulimanäyte). Kolibakteeri on kuitenkin hyvin herkkä antibiooteille, joten tulehdukset ovat helppohoitoisia.
Jotkin muunnokset bakteerista tuottavat myös toksiineja, kuten Shiga-toksiineja[6] ja saattavat aiheuttaa ruokamyrkytyksen.[7] Yksi suolistotulehduksia aiheuttavista E. coli -ryhmistä on Enterohemorraaginen Escherichia coli -bakteeri eli EHEC-bakteeri, ja niistä on 2000-luvulla tullut yleinen ruokamyrkytysten aiheuttaja.[6] Pahimmillaan EHEC voi aiheuttaa voimakasta veriripulia, ja noin 10 prosentilla sairastuneista saattaa tulla vakava munuaisten toimintahäiriö, ureeminen oireyhtymä HUS (yleensä lapsilla). Se voi johtaa myös tromboottiseen trombosytopeeniseen purppuraan TTP (TTP tarkoittaa ihoverenvuotoa eli purppurana ilmenevää verenvuototaipumusta) (yleensä vanhuksilla). Tauti saattaa johtaa kuolemaan. EHEC leviää varsinkin jauhelihan ja pastöroimattoman maidon välityksellä, jolloin taudin alkuperä on yleensä nautakarjan suolisto. Tämä on yleistä länsimaissa muun muassa Pohjois-Amerikassa, jossa bakteeri on levinnyt huonosti kuumennettujen hampurilaisten tai muun pikaruoan välityksellä. Tätä vakavampaa tautityyppiä on tavattu silloin tällöin Suomestakin. Ensimmäinen EHEC-epidemia (kanta 0157:H7) ja myös ensimmäiset kuolemantapaukset diagnosoitiin Suomessa vuonna 1997. Suomessa todetaan vuosittain noin 10-30 EHEC-tartuntaa.[8]
Ruotsissa myrkyllistä 0157:H7-muunnosta on kymmenessä prosentissa maitotiloista, Hollannissa jopa joka neljännellä tilalla. Verosytotoksiinia muodostavista muunnoksista käytetään lyhennettä VTEC (verosytotoksiinia muodostavat E. kolibakteerit. Ryhmän uudet muunnokset, bakteerit O21 ja O103, ovat levinneet Ruotsissa yhtä laajalle kuin 0157:H7, ja joissakin naapurimaissa aiheuttaneet kuolemantapauksia[7].
EHEC-tartunta voi olla hengenvaarallinen ja tämänhetkiset hoidot perustuvat pääosin oireiden lievittämiseen. EHEC-bakteerin tuottamien toksiinien takia antibioottien käyttöä ei suositella taudin hoitomenetelmänä. Antibioottien vaikutuksesta kuolevat bakteerisolut voivat erittää toksiineja ympäristöönsä ja näin pahentaa taudin oireita.[9]
Kesäkuussa 2011 ilmeni Saksassa 1 700 EHEC-tapausta, joista kuoli ainakin 30. Suomessa tapauksia ilmeni yksi, joka oli todennäköisesti peräisin Saksasta, sillä se muistutti saksalaista kantaa.[10][11] Tartunnan lähteenä ovat olleet Saksassa kasvatetut idut.[12]
Richard Lenski aloitti Michigan State Universityssä Yhdysvalloissa 12. helmikuuta 1988 Escherichia colin evoluutiotutkimuksen jolloin 12 koeputkeen kylvettiin lähes identtiset E. coli -populaatiot. Koetta kutsutaan nimellä LTEE eli E. coli long-term evolution experiment.
Bakteereille annettiin minimiravinto, jolloin 500 bakteerisukupolvea syntyi 75 päivässä. Yksistään tämä tuottaisi valtavan määrän bakteereita, jos bakteerit saisivat lisääntyä rajatta. Mutta bakteerien rajallinen ravintomäärä aiheuttaa sen, että ne bakteerit, jotka pystyvät lisääntymään nopeiten, yleistyvät. Tarkoitus oli tutkia, tapahtuuko bakteereissa hyödyllisiä mutaatioita, jotka edistävät bakteerien kykyä selviytyä hengissä ja tuottaa jälkeläisiä.
Parinkymmenen vuoden kuluttua, vuonna 2008, oli tapahtunut 40 000 solunjakautumista. Tähän mennessä oli näkynyt joitain muutoksia bakteereissa. Kun noin 20 000 bakteerisukupolvea oli käyty läpi, monia geneettisiä muutoksia oli tapahtunut kaikissa maljoissa, ja bakteerien kasvu oli nopeutunut 70 %:lla. Kaikista bakteereista oli kehittynyt suurempia ja harvemmassa eläviä, ja ne erikoistuivat elämään glukoosissa. Suurin osa koon kasvusta tapahtui 2 000 ensimmäisen sukupolven aikana[13]. Neljällä populaatiolla heikkeni DNA:n korjausmekanismi, mikä kasvatti mutaatioiden määrää. Vaikka bakteerit läpikävivät maljaa kohden miljoonia mutaatioita 20 000 ensimmäisen solunjakautumisen aikana, Lenskin mukaan vain 10–20 mutaatioista oli hyödyllisiä, ja vain 100 kaikista hyödyllisistä ja selviämisen kannalta yhdentekevistä mutaatioista jäi geeneihin. Kokoa kasvattaneista populaatioista Ara-1 ja Ara+1 kaikki koon määränneet samat 59 geeniä olivat muuttuneet samaan suuntaan toisistaan riippumatta[14].
33 127 jakautumisen jälkeen eräs populaatio, joka on ristitty Ara-3:ksi pystyi hyödyntämään kasvuaineen sitraattia, mihin normaali E. coli ei pysty[15]. Tämä kasvatti bakteerien määrää ja näytti selvästi, että hyödyllinen mutaatio oli tapahtunut laboratoriossa.[16]. Sitraattikyky vaati bakteerilta ainakin kaksi peräkkäistä mutaatiota, ja siksi sitä ei ilmestynyt kaikkiin maljoihin mutaatioiden suhteellisen harvinaisuuden takia[17].
Epidemioiden yhteydessä on tavallisesti puhe virotyypeistä, joiden aiheuttama taudinkuva on tyypillisesti toisesta poikkeava. Kyseessä ei ole eri mikrobilaji. Ihmisillä esiintyviä ovat
Kolibakteeri eli Escherichia coli (E. coli) on yksi niistä bakteerilajeista, jotka elävät tasalämpöisten eläinten (muun muassa nisäkkäiden ja lintujen) ruoansulatuskanavan alaosassa. Jos pohjavedessä on kolibakteeria, se kertoo yleensä ulostesaastumisesta. Ihmisen päivittäisessä ulostemäärässä on tavallisesti 1011–1013 kolibakteeria. Bakteeri on saanut tieteellisen nimensä löytäjänsä Theodor Escherichin mukaan. E. coli kuuluu enterobakteereihin.
Escherichia coli, également appelée colibacille et abrégée en E. coli, est une bactérie intestinale (Gram négatif) des mammifères, en forme de bâtonnet, très commune chez l'être humain. E. coli est une bactérie anaérobie facultative que l'on trouve dans l'intestin des vertébrés. En effet, elle compose environ 80 % de notre flore intestinale aérobie. Découverte en 1885 par Theodor Escherich, dans des selles de chèvres, c'est un coliforme fécal généralement commensal. Cependant, certaines souches d’E. coli peuvent être pathogènes, entraînant alors des gastro-entérites, infections urinaires, méningites ou sepsis. Sous certaines conditions, E. coli serait capable de produire du méthane.
Escherichia coli est un bacille gram négatif radiorésistant de la famille des Enterobacteriaceae[1]. Sa taille varie en fonction des conditions de croissance (entre 0,5 à 3 µm), pesant de 0,5 à 5 picogrammes[2] les bactéries en croissance rapide étant plus allongées et donc plus grandes que les bactéries quiescentes. Le diamètre d'E. Coli est de 0,5 µm en moyenne[1].
C’est un hôte commun du microbiote intestinal (anciennement appelé microflore commensale intestinale) de l’humain et des animaux homéothermes[3]. Son établissement dans le tractus digestif s’effectue durant les premières heures ou journées qui suivent l’accouchement [4]. Escherichia coli constitue alors tout au long de la vie de l’hôte l’espèce bactérienne dominante de la flore aérobie facultative intestinale. E. coli est sans doute l’organisme vivant le plus étudié à ce jour : en effet, l'ancienneté de sa découverte et sa culture aisée (division cellulaire toutes les vingt minutes à 37 °C dans un milieu riche) en font un outil d'étude de choix. La profusion de publications scientifiques qui la mentionnent en témoigne, et elle joue le rôle de « cheval de labour » dans tous les laboratoires de biologie moléculaire.
On a récemment montré qu'en présence de fer libre, elle peut produire du méthane[5].
Theodor Escherich, en observant la fréquence des diarrhées néonatales, avait déjà posé la question de l’implication du colibacille dans les entérites. Après la Seconde Guerre mondiale, les connaissances ont convergé pour établir le concept de virulence de certaines souches de E. coli. Dans les années 1950, de nombreuses souches d’E. coli ont été incriminées en tant qu’agent étiologique de diarrhées infantiles[6].
En prévention, une surveillance des SHU a lieu au Centre National de Référence des E. coli[7], situé dans l’unité de Biodiversité des Bactéries Pathogènes Émergentes à l’Institut Pasteur (France), qui est chargé d’étudier les souches pathogènes.
Depuis les années 1950, les bactériologistes ont essayé, grâce aux différences antigéniques de E. coli, de subdiviser l’espèce en sérotypes en immunisant des lapins avec des antigènes somatiques et flagellaires. Le sérogroupage reste la méthode la plus utilisée actuellement.
Le sérotype est la combinaison des deux antigènes, somatique O et flagellaire H, (exemples : O157:H7 et O111:H8), alors que le sérogroupe n'est déterminé que par l’antigène O (exemple : O157, O111). Cependant le sérotype n’est pas suffisant pour caractériser les E. coli pathogènes. Chaque sérotype n’est pas nécessairement corrélé à la pathogénicité.
Depuis 2010, un laboratoire de Californie (LS9[8]) a expliqué qu'il utilise cette bactérie pour produire des hydrocarbures, technique qui sera peut-être à l'origine de la synthèse du pétrole[réf. nécessaire]. Cette bactérie est encore depuis 2010 l'objet d'études en xénobiologie.
L’antigène somatique O, définissant le sérogroupe, est contenu dans les lipopolysaccharides présents sur la paroi bactérienne des souches à Gram négatif. L’antigène flagellaire H est de nature protéique entrant dans la structure du flagelle (cilliature péritriche) permettant la mobilité de la bactérie. L'antigène K de surface n'est pas toujours présent mais s'il est présent, il bloque l'agglutinabilité de l'antigène O.
Il en existe plus de 150. Les antigènes somatiques sont composés de lipopolysaccharides complexes. Actuellement certains laboratoires d'analyses médicales utilisent l'agglutination avec des sérums pour déterminer le sérogroupe, mais cette technique est limitée par le nombre de plus en plus élevé de sérums à fabriquer, par la présence d'agglutinations croisées entre les antigènes O de E. coli, Shigella et ceux de Salmonella, et par le passage de la consistance crémeuse de la colonie à une consistance rugueuse ayant pour conséquence l’absence de synthèse de l'antigène O. C'est pour cette raison qu'une technique de sérotypage moléculaire a été développée.
L'antigène O fait partie du lipopolysaccharide (LPS) de la membrane externe (faisant partie intégrante de la paroi cellulaire fine) des bactéries à Gram négatif. Il contient un grand nombre d’unités répétées d’oligosaccharides de trois à six sucres dont la combinaison détermine la diversité des antigènes O. Les gènes codant les enzymes impliquées dans la synthèse de l’antigène O sont regroupés dans le groupe de gènes rfb.
Ce groupe rfb peut être amplifié spécifiquement grâce à un système d’amorces puis, après restriction par l’endonucléase MboII, un profil noté « R » peut être obtenu par électrophorèse, correspondant à un sérogroupe de E. coli[10]. Un profil d'électrophorèse est fonction de l’emplacement des sites de restriction propre à MboII. Ainsi tous les groupes de gènes correspondant à un antigène somatique auront un profil de restriction qui lui est propre. Ce profil R sera ensuite analysé avec le logiciel Taxotron puis comparé à une base de données, en perpétuel développement. Par exemple, le profil R aura un numéro R111, correspondant au sérogroupe O111 obtenu avec le sérum.
Les antigènes H ne servent pas à l'identification des E. coli pathogènes mais présentent un grand intérêt au point de vue épidémiologique : l'identité de l'antigène H constitue un élément pour assurer qu'il s'agit d'une même souche.
La diversité des antigènes H est due aux différents types de flagelline composant la structure du flagelle. C'est le flagelle qui permet la mobilité bactérienne. Le typage s'effectue également par séroagglutination, mais n’est développé que dans de très rares laboratoires dans le monde. Cependant, certaines souches perdent leur mobilité et sont classées comme non mobiles (NM ou H-). Une technique de sérotypage moléculaire a donc été également développée pour déterminer l'antigène H.
L'antigène H est codé par le gène fliC. Les parties N et C terminales de la flagelline sont très conservés et c'est la partie médiane, plus variable, qui donne la spécificité de l'antigène H. Les E. coli immobiles possèdent également le gène fliC mais sont incapables de synthétiser un flagelle fonctionnel. Après amplification et restriction du gène fliC, il est possible de typer l'antigène H en comparant le profil obtenu à une base de données de profil-type[11]. Par exemple, le profil fliC (noté F) aura un numéro F8, correspondant au type H8 obtenu avec le sérum.
Il existe trois types d'antigène K désignés par les lettres L, A ou B :
Différence entre l'antigène B et les antigènes A ou L, dans une population homogène sur boîte de Petri :
C'est une bactérie de la famille des Enterobacteriaceae ne possédant pas de désaminase, ce qui exclut les genres Proteus, Morganella et Providencia (typiquement ex-tribu des Proteae).
Elle fermente le glucose par la voie des acides mixtes (rouge de méthyle +, VP -) ce qui exclut les genres Klebsiella, Enterobacter, Hafnia et Serratia (typiquement groupe des KEHS, ex-tribu des Klebsielleae).
De plus,
Le patrimoine génétique de la souche E. coli de laboratoire non pathogène a été entièrement séquencé en 1997. Son génome comprend 4,6 millions de paires de bases codant environ 4 200 protéines[12].
En 2001, le génome d'une souche d’E. coli entérohémorragique[13] (provoquant la maladie du hamburger) a été séquencé. Il comprend 5,5 millions de paires de bases codant 5 400 protéines. L'année suivante, le génome d'une souche d'E. coli provoquant des infections urinaires (cystite, pyélonéphrite) et des méningites néonatales, a été séquencé. Il comprend 5,2 millions de paires de bases codant 5 300 protéines.
La comparaison des génomes de ces trois souches d’E. coli révèle que seulement 40 % de leurs gènes sont communs — à titre de comparaison, 99 % des gènes de l'humain et des grands singes sont communs. Ceci témoigne du remarquable potentiel évolutif et de la versatilité de ce taxon bactérien. En effet, les souches d’E. coli pathogènes ont acquis au cours de l’évolution un répertoire de gènes de virulence, qui leur permettent de coloniser de nouvelles niches écologiques en contournant les mécanismes de défense de l’hôte. L’expression d’un répertoire spécifique de facteurs de virulence est corrélée à une pathologie particulière et permet de définir différents pathovars (voir infra).
Le concept de la pathogénicité bactérienne résultant d’un processus multifactoriel, impliquant une myriade de gènes, dont l’expression est chorégraphiée par des processus de régulation est maintenant bien accepté. L’expression de ces gènes permet une adhésion plus efficace, ou l’invasion des tissus de l’hôte, et permet ainsi la colonisation de niches inaccessibles ou inhospitalières pour les E. coli commensaux. En ce sens, la pathogénicité peut être considérée comme un avantage sélectif, et le succès d’une souche d'E. coli en tant que pathogène requiert probablement l’acquisition et la sélection de gènes de virulence, envers des recombinaisons et des transferts génétiques non spécifiques. La plasticité du génome d’E. coli est à la base de ce processus. La séquence complète du génome de plusieurs souches d’E. coli montre la présence de nombreuses Séquence d'insertion (IS), de séquences bactériophagiques, ainsi que d'autres plages de séquences inusuelles qui témoignent de l’extraordinaire plasticité du génome de ce genre bactérien. Ce sont les isolats cliniques d’E. coli qui possèdent les plus grands génomes, alors que celui de l’E. coli de laboratoire, non pathogène, fait 4,63 Mb. Il apparaît ainsi que le fossé qui sépare les E. coli commensales des E. coli pathogènes est dû à l'acquisition de répertoires de gènes de virulence. Il se pourrait que l’acquisition de ces gènes soit facilitée par une importante aptitude à muter. En effet, plus d'1 % des isolats d’E. coli ou de Salmonella impliqués dans des infections alimentaires sont des « mutateurs » qui présentent une forte tendance à muter, un phénomène corrélé à une déficience dans certains systèmes de réparation de l’ADN. Les gènes de virulence sont le plus souvent localisés sur des éléments génétiques transmissibles comme des transposons, des plasmides ou des bactériophages. De plus, ils peuvent être regroupés sur de grands blocs d’ADN chromosomique appelés « îlots de virulence » ou «îlots de pathogénicité».
Les recherches génétiques sur la bactérie ont permis la découverte de l'ARNI ou ARN I[14] (à ne pas confondre avec l'ARN interférent, ARNi). C'est un ARN non codant qui est un répresseur antisens de la réplication de certains plasmides d'Escherichia coli, dont ColEl[15]. Celui-ci explique la neutralisation de certains gènes.
Certaines souches spécialisées d’E. coli sont associées à des pathologies très diverses tant chez l’être humain que chez l’animal ; diarrhées, gastro-entérites, infections urinaires, méningites, septicémies, etc. Les techniques modernes de la biochimie, de la génétique, de la biologie moléculaire et de la microbiologie cellulaire ont permis d’identifier et d’analyser les mécanismes impliqués dans l’interaction des E. coli pathogènes avec leur hôte. Malgré la diversité des affections provoquées par les souches d’E. coli pathogènes, toutes ces souches utilisent une stratégie classique d’infection, commune à de nombreux autres agents pathogènes.
Comme la plupart des pathogènes des muqueuses, les souches d’E. coli responsables de diarrhées et d'infections extra-intestinales utilisent une stratégie d'infection dont les points clés sont les suivants: colonisation des muqueuses, éventuellement invasion des cellules, multiplication, évasion des défenses de l’hôte, dommages à l’hôte.
Pour survivre et se multiplier dans le tractus intestinal, les colibacilles doivent surmonter les premières lignes de défense de l’organisme hôte, à savoir le péristaltisme et l’effet de barrière de la microflore commensale. Cette microflore accapare les nutriments, produit des inhibiteurs et occupe les surfaces des muqueuses. L’effet de barrière est surmonté par les E. coli pathogènes grâce à des mécanismes qui sont connus en termes généraux : la compétition pour les sources de carbone, de fer, d’énergie sous des conditions anaérobies, la production de bactériocines, ainsi qu’un fort taux de croissance. L’étape de colonisation implique aussi la capacité à adhérer à la surface de la muqueuse intestinale. Virtuellement toutes les souches d’entérobactéries pathogènes ou non possèdent des systèmes d’adhésion, et il est bien établi que ce pouvoir d’adhésion est la caractéristique la plus conservée chez les E. coli pathogènes. Les structures bactériennes responsables de l’adhésion aux cellules épithéliales sont des adhésines fimbriaires (fimbriae ou pili) ou afimbriaires. Exposées à la surface des bactéries, ces adhésines interagissent avec des récepteurs de la membrane des cellules cibles. C’est ainsi que des souches d'E. coli pathogènes sont capables en partie grâce à leurs adhésines de coloniser des biotopes qui ne sont normalement peu ou pas colonisés par les E. coli commensales. Par exemple, les E. coli responsables d'infections urinaires déploient des pili « P » (pili associés aux pyélonéphrites) qui reconnaissent des glycolipides à la surface des cellules épithéliales du tractus urinaire.
La multiplication est essentielle dans le processus de pathogénicité ; on conçoit en effet qu’une multiplication rapide est un avantage pour la colonisation, ainsi que pour causer des dommages avant que le système immunitaire n'entre en action. Une multiplication lente, voire son arrêt, peut aussi constituer un avantage dans la persistance des pathogènes qui causent des maladies chroniques.
Un autre point essentiel dans le processus de pathogénicité est l’interférence des E. coli pathogènes avec le système immunitaire de l’hôte. On sait par exemple que certains types de lipo-polysaccharides (LPS ; antigène « O ») présents à la surface des bactéries les protègent de l’action lytique du complément, de la fixation des anticorps et de la phagocytose. Les capsules polysaccharidiques (antigènes « K ») qui sont sécrétées à la surface de certaines souches d’E. coli pathogènes (principalement celles causant des affections extra-intestinales) peuvent participer à l’évasion des défenses de l’hôte. Les capsules K1 et K5, qui comportent des homologies avec des molécules eucaryotes (les adhésines n-CAM et les héparanes), présentent ainsi une faible immunogénicité. Les variations antigéniques de certaines molécules protéiques de surface (comme les pili), peuvent également participer à l’évitement des défenses immunitaires.
La première étape de colonisation effectuée, certaines souches pathogènes produisent de puissantes toxines, ces dernières pouvant être responsables à elles seules des dommages infligés à l’hôte. D’autres souches pathogènes détournent à leur avantage des fonctions cellulaires essentielles, afin de survivre et persister. Ainsi, en altérant le cytosquelette cellulaire, elles peuvent adhérer très fortement à la surface cellulaire (on parle d’adhésion « intime »), voire pénétrer dans les cellules des muqueuses et s’y multiplier, telles Shigella flexneri ou Salmonella typhimurium.
Sur la base de ces modes d’interaction et des signes cliniques de l’infection, les souches d’E. coli inductrices de diarrhées peuvent être actuellement classées en cinq pathovars : E. coli entérotoxigéniques (ETEC), E. coli entéroinvasives (EIEC), E. coli entéropathogènes (EPEC), E. coli entérohémorragiques[13] (EHEC) et E. coli entéroaggrégatives (EAggEC). Outre les E. coli induisant des diarrhées, on distingue aussi le pathovar des E. coli pathogènes extraintestinales (ExPEC) impliquées dans des affections non-intestinales: infections urinaires, méningites, septicémies, mammites...
Une dizaine de types particuliers d'E. coli, caractérisés par leurs antigènes, peuvent causer un syndrome entéritique grave avec toxicose et déshydratation chez les jeunes enfants. On admet généralement que ces colibacilles ne sont pathogènes qu'en dessous de l'âge de 2 ans mais c'est surtout chez les nouveau-nés et plus particulièrement chez les prématurés que ces germes se manifestent de la façon la plus sévère. L'allaitement maternel confère une certaine protection contre ces infections[réf. souhaitée] (rôle des bifides de la flore intestinale).
Dans les collectivités de nourrissons (maternités, services pédiatriques), la maladie prend généralement une allure épidémique : il s'agit dans ces cas de souches à la fois virulentes et multirésistantes réalisant le tableau d'une véritable infection nosocomiale.
Les ECET sont une cause majeure de diarrhée aqueuse chronique avec déshydratation chez les enfants de bas âge (moins de 3 ans) dans les pays en voie de développement, et sont aussi responsables de la « diarrhée des voyageurs » (ou « turista »). Des ECET sont également une cause fréquente de diarrhées néonatales souvent fatales chez les animaux d’élevage (veau, agneau, porcelet).
Les ECET colonisent essentiellement la partie proximale de l’intestin grêle, grâce à leurs « facteurs de colonisation » (CFAx et CSx) qui sont des adhésines fimbriaires. Les ECET n’induisent pas d’altérations histologiques marquées de la muqueuse. Le pouvoir pathogène des ECET s’explique principalement par la sécrétion des toxines thermostables (ST) et/ou thermolabiles (LT). La toxine LT, après endocytose, ADP-ribosyle la sous-unité alpha de la protéine hétérotrimérique Gs. Il s’ensuit l’hyper-activation de l’adénylate cyclase, l’augmentation de la concentration du second messager AMPc, et la phosphorylation de transporteurs membranaires - particulièrement le « CFTR », le régulateur de la conductance membranaire impliqué dans la mucoviscidose. Cette action se traduit par une sécrétion d’ions chlorure et une inhibition de l’absorption de chlorure de sodium par les cellules intestinales, ce qui provoque la diffusion osmotique d’eau vers la lumière intestinale. L’action des toxines ST est moins connue. En se fixant à leur récepteur à la surface des cellules intestinales (une guanylate cyclase), elles induisent des concentrations accrues en GMPc, ce qui résulte également en l’activation du CFTR, l’altération de l’homéostasie intestinale, et une diarrhée osmotique. Ainsi, c’est l’action des toxines ST et LT qui explique le tableau clinique de l’infection : diarrhée aqueuse peu fébrile, nausées et crampes abdominales.
Les ECEI sont responsables de syndromes dysentériques caractérisés par une forte fièvre, des crampes abdominales et des nausées, accompagnés d’une diarrhée aqueuse qui évolue rapidement en une dysenterie (selles contenant du sang et du mucus). Les ECEI ont des caractères biochimiques, antigéniques, génétiques et fonctionnels très proches de ceux des Shigella, et mettent en œuvre un mécanisme de pathogénicité similaire. Les ECEI et les Shigella envahissent la muqueuse intestinale au niveau du côlon, s’y multiplient, provoquent la mort cellulaire et déclenchent une intense réaction inflammatoire. Le processus d’invasion est complexe et multifactoriel, sous la dépendance de loci chromosomiques et d’un plasmide de virulence (pInv ; ~220 kb). En ce qui concerne Shigella, le processus d’invasion peut être résumé ainsi : lors du contact avec les cellules épithéliales, les bactéries sécrètent des « invasines » (Ipa), qui interagissent avec la surface cellulaire et provoquent un réarrangement localisé du cytosquelette aboutissant à la pénétration de la bactérie dans la cellule. Une fois en position intracellulaire, la membrane de la vacuole est rapidement lysée grâce à une hémolysine de contact, libérant les bactéries dans le cytoplasme où elles peuvent se multiplier. Puis, les bactéries induisent la polymérisation de l’actine cellulaire à un de leurs pôles (grâce à IcsA) pour se mouvoir et se disséminer de cellule en cellule. Le processus d’invasion mis en jeu par les EIEC reste à être élucidé, mais les données actuelles indiquent qu’il est probablement identique à celui de Shigella. Les ECEI et les Shigella élaborent également une ou plusieurs entérotoxines qui seraient impliquées dans l'épisode de diarrhée aqueuse qui précède la dysenterie.
Les ECEP sont responsables de gastro-entérites infantiles (GEI). On admet généralement que ces colibacilles ne sont pathogènes qu'en dessous de l'âge de deux ans. Les principaux sérotypes impliqués sont O111 B4 et O119 B14. Le plus fréquent dans l'UE ces dernières années est l'O111 B4 mais on commence à parler d'autres types (O55 et O26). Chez l'adulte, en principe, les E. coli de GEI ne sont pas pathogènes. Certains avancent que certaines diarrhées du voyageur seraient dues à des types d'E. coli particuliers inconnus dans la région où vit le voyageur.
Ce n'est que chez les nourrissons en bas âge que la maladie prend une allure grave et épidémique (surtout et presque uniquement en milieu hospitalier). Il s'agit de diarrhées avec déséquilibre de la balance ionique ; d'où le plus important est de rétablir la balance ionique ; l'antibiothérapie interviendra secondairement.
Les ECEH sont responsables de colites hémorragiques[16]. Le principal réservoir de ces bactéries est le tube digestif des bovins ; la contamination humaine se fait par l'intermédiaire d'aliments, principalement la viande de bœuf hachée et le lait cru. Le sérotype O157 est le plus fréquent (ex : sérotype O157:H7 pour la « maladie du hamburger »[17]). Il est responsable d'épidémies. Les ECEH produisent une verotoxine (ou Shiga-toxine) qui peut entraîner un syndrome hémolytique et urémique (SHU). Des intoxications à ECEH se sont déclarées à la suite de l'ingestion de viande contaminée et insuffisamment cuite (hamburger). Une intoxication a eu lieu en France en 2005.
Les cytotoxines (vérotoxines) sont à l'origine de la destruction des cellules intestinales. Les symptômes peuvent aller de la diarrhée simple à une diarrhée sanglante et abondante. Les manifestations sont plus graves chez les enfants de moins de 8 ans et chez les personnes de plus de 65 ans.
Le syndrome hémolytique et urémique (SHU) se manifeste entre autres par une anémie hémolytique, une thrombopénie et une insuffisance rénale aiguë.
Jusqu’au début des années 1980, les souches d’E. coli inductrices de diarrhées étaient classées en trois catégories ; les ECET, les ECEI et les ECEP, ces dernières étant alors caractérisées essentiellement par leur appartenance à des sérotypes distinctifs. Au début des années 1980, il a été constaté que la plupart des souches classées dans les ECEP adhèrent sur des cellules de lignée. Par la suite, trois modes distincts d’adhésion aux cellules ont été décrits : l’adhésion « localisée », l’adhésion « diffuse », et l’adhésion « agrégative », ce qui a permis de différencier un nouveau pathovar d’E. coli diarrhégéniques, les EAggEC. Les ECEAgg sont actuellement définies comme des souches qui ne sécrètent pas les entérotoxines LT ou ST, et qui adhèrent aux cellules de culture en formant des images « d’amas de briques » (adhésion agrégative). Il est probable que cette définition inclut des souches non pathogènes ; l’hétérogénéité de ce groupe a d’ailleurs été confirmée par des études épidémiologiques et des infections expérimentales d’adultes volontaires. Néanmoins, les ECEAgg sont de plus en plus reconnues comme étant responsables de retards de croissance et de diarrhées persistantes dans les pays en voie de développement ainsi que dans les pays industrialisés. Des fimbriae (AAF/I et AAF/II) responsables de l’adhésion agrégative ont été décrits, mais ils sont présents dans une minorité d’isolats d’ECEAgg. Ceci suggère que la colonisation du tractus digestif serait due à un ou plusieurs fimbriae, comme dans le cas des ECET. Les ECEAgg adhèrent sur la muqueuse intestinale, enchâssées dans un biofilm de mucus produit par les cellules en gobelets de la muqueuse. Environ 40 % des souches d’ECEAgg produisent l’entérotoxine EAST1 (pour « EAggEC ST-like toxin »), qui présente environ 50 % d’homologie avec la toxine ST des ECET et 30 % avec les toxines amnésiantes de guanylate cyclase de coquilles STJ. La contribution de ces toxines au pouvoir pathogène n’est pas encore établie. Le gène codant la toxine EAST1 ou ses variants a été trouvé dans des souches ECEP et ECET, ainsi que dans des souches d’E. coli pathogènes ou non, qui ne possèdent pas les gènes marqueurs des pathovars classiques.
Les ECDA ont initialement été classées avec les E.Coli entéro-pathogènes du fait de leur adhésion aux cellules Hep-2 (lignée cellulaire d'origine tumorale) mais forment actuellement un groupe à part. En effet, avec la découverte des ECEAgg (E.Coli entéroaggrégatives) les ECDA ont été différenciées par leur phénotype d'adhésion n'impliquant pas d'agrégats microbiens et formant des lésions diffuses sur les cellules Hep-2. Les ECDA sont essentiellement responsables de pathologies extra-intestinales (infections urinaires : cystites, pyélonephrites). Cependant, les études menées ont démontré que selon la tranche d'âge les ECDA pourraient également induire des signes cliniques intestinaux (diarrhée) - la sensibilité aux ECDA serait étroite, située entre un et cinq ans.
Les E. coli sont pathogènes dans des localisations extra-intestinales : méningites, abcès, péritonites, septicémies et surtout infections urinaires (UPEC).
Les germes charriés par la circulation (bactériémie) viennent se fixer au niveau de tractus urinaire si une cause favorisante permet leur implantation : rétrécissement, malformation, calcul. Ces causes entraînent une stagnation.
Cette origine est la plus fréquente. Des germes d'origine fécale en provenance de la région péri-anale remontent dans la vessie, surtout chez les femmes. L'origine explique la fréquence des infections urinaires par germes fécaux surtout les entérobactéries : colibacilles, klebsiella, proteus mais aussi les entérocoques.
Sondage, cathétérisme. Ces manœuvres peuvent introduire des germes à partir de l'extérieur, en particulier le pyocyanique.
Le prélèvement devrait idéalement se faire par ponction sus-pubienne ou par sondage. Comme la première méthode n'est pas toujours réalisable en pratique et qu'il est déconseillé d'effectuer systématiquement la seconde (risque d'infections iatrogènes), on se contente généralement d'utiliser un échantillon récolté à la miction (partie moyenne du jet) après toilette des organes génitaux externes. Cette façon de procéder n'empêche cependant pas l'urine d'être souillée par des germes des orifices externes, gênants pour l'interprétation des résultats. De ce fait, ce prélèvement n'est valable que moyennant une analyse bactériologique quantitative. On admet généralement que la présence de moins de 10 000 germes par ml d'urine correspond à une contamination externe, alors que plus de 100 000 germes traduisent une bactériurie significative. Entre ces deux nombres, le résultat est plus difficile à interpréter. Pour que cette analyse quantitative soit faite dans de bonnes conditions, il faut que l'échantillon soit frais. Une méthode simple de triage quantitatif est la culture sur lames gélosées (lame de verre tapissée de gélose) plongées dans l'urine : le nombre de colonies apparaissant après incubation reflète le nombre de germes par ml d'urine. Cette méthode doit être complétée par l'identification des germes et la réalisation de l'antibiogramme. Des méthodes plus rigoureuses peuvent être appliquées au laboratoire par l'ensemencement d'un volume déterminé d'urine diluée sur une boîte de Petri et par la numération des colonies obtenues.
Les EHEC résistent au froid, mais sont détruites par la cuisson.
Il faut donc rappeler, notamment pour les populations sensibles :
Il est impératif :
Dans certains pays, et en particulier aux États-Unis ou au Royaume-Uni, les autorités sanitaires préconisent une cuisson des viandes hachées à au moins 70 °C à cœur maintenus pendant deux minutes.
Pour une pièce de viande non hachée, les bactéries, présentes uniquement à la surface, sont facilement détruites lors de la cuisson par un passage, même rapide, à la poêle. Mais, dans le cas de la viande hachée, des bactéries sont susceptibles d’être présentes en profondeur et ces dernières ne seraient pas détruites par une cuisson rapide (viande bleue ou saignante) : une cuisson plus longue est indispensable.
Pour que la cuisson des steaks hachés surgelés destinés aux enfants permette d’atteindre en quelques minutes une température à cœur suffisante pour détruire les bactéries, il est recommandé de décongeler les steaks avant leur cuisson. La décongélation doit alors être faite soit en conservant les steaks le temps nécessaire dans la partie la plus froide du réfrigérateur dans leur conditionnement protecteur d’origine, soit dans un four à micro-ondes. La décongélation ne doit jamais être faite à température ambiante.
De plus, les légumes, mais aussi les fruits et les herbes aromatiques, en particulier ceux qui vont être consommés crus, doivent être soigneusement lavés puis épluchés si nécessaire avant leur préparation et leur consommation.
Les aliments crus doivent être conservés séparément des aliments cuits ou prêts à être consommés pour éviter les contaminations croisées (bonne séparation de la viande crue des autres aliments dans le réfrigérateur[20]).
Enfin, les recommandations d’hygiène courantes doivent également être appliquées afin d’éviter la transmission de personne à personne et la contamination croisée des aliments : lavage soigneux des mains et séchage avec un torchon propre avant de préparer le repas, plus particulièrement pour les jeunes enfants ; lavage soigneux des couteaux, planches à découper et autres ustensiles avant de cuisiner ; et lavage soigneux des mains après être allé aux toilettes[21].
Résistances naturelles : Escherichia coli est sensible à toutes les bêta-lactamines malgré la production d'une céphalosporinase chromosomique non inductible de type AmpC qui peut entraîner chez certaines souches une réduction de la sensibilité aux aminopénicillines, à leurs associations au clavulanate et/ou au C1G.
La phagothérapie est utilisée chez l'humain dans certains pays comme la Russie et la Géorgie, mais la société ne communique ni sur la composition de ses préparations ni sur ses résultats. Des données regroupant différents petits tests cliniques montreraient une éradication du pathogène dans 40 % des cas, avec un taux de récupération des patients de 18 %. Globalement, ce secteur de la recherche souffre d'un manque d'essais contrôlés et du désintérêt de l'industrie pharmaceutique, seule l'industrie alimentaire ayant déposé des brevets de phages qualifiés d'additifs alimentaires, sans doute pour contourner une réglementation contraignante. L'autre contrainte est le manque de connaissances sur les mécanismes d'action[22].
Des chercheurs de l'université du Michigan (États-Unis) ont publié le 18 septembre 2009 dans la revue PLoS Pathogens les résultats prometteurs obtenus sur des souris qui créent une forte résistance immunitaire contre la bactérie Escherichia coli.
L'équipe dirigée par le Dr Mobley a analysé 5 379 protéines bactériennes susceptibles d'induire une réponse immunitaire. Sur les six substances retenues comme candidats vaccins et injectées par voie nasale à des souris, trois ont permis de ne pas contracter d'infection.
Les chercheurs américains ont établi que ces protéines ciblent des récepteurs du fer situés à la surface de la bactérie et qui jouent un rôle crucial dans la propagation de l'infection. Surtout, la réponse immunitaire est induite aussi bien dans la muqueuse nasale, où l'injection est pratiquée, que dans celle de l'appareil urinaire.
L'équipe du Dr Mobley a également montré que ses candidats vaccins sont efficaces sur plusieurs souches d'E. coli et envisage de procéder à des essais cliniques de phase 1 sur des volontaires humains.
La souche E. coli ST131, multirésistante aux antibiotiques, est impliquée dans des infections urinaires. Le gène qui leur confère cette grande résistance se transmet facilement entre individus E. coli par plasmides[23],[24]. Il code l'enzyme aminoglycoside-(6')-N-acetyltransferase (aac(6’)-Ib-cr) qui inactive la ciprofloxacine, un antibiotique à large spectre de la classe des fluoroquinolone (FQ).
Escherichia coli étant une bactérie, elle est sensible aux bactériophages comme les phages T4 et lambda.
Escherichia coli est très utilisée pour sa capacité à produire l'enzyme de restriction EcoRI.
En 2008, après un premier pilote industriel (installé en 2007 dans la raffinerie de Pomacle-Bazancourt près de Reims), E. coli est également utilisée pour produire de l'acide succinique à partir de sucres et résidus lignocellulosiques fermentés en atmosphère enrichie en CO2[25]. Cette raffinerie dispose d'une capacité de production de 2 000 t/an.
L'acide succinique est notamment utilisé comme excipient dans le vaccin Prevenar13 de Wyeth Lederle Vaccines SA (Pfizer)[26].
Escherichia coli est aussi utilisé comme hôte hétérologue pour produire de nombreuses protéines[27].
Escherichia coli, également appelée colibacille et abrégée en E. coli, est une bactérie intestinale (Gram négatif) des mammifères, en forme de bâtonnet, très commune chez l'être humain. E. coli est une bactérie anaérobie facultative que l'on trouve dans l'intestin des vertébrés. En effet, elle compose environ 80 % de notre flore intestinale aérobie. Découverte en 1885 par Theodor Escherich, dans des selles de chèvres, c'est un coliforme fécal généralement commensal. Cependant, certaines souches d’E. coli peuvent être pathogènes, entraînant alors des gastro-entérites, infections urinaires, méningites ou sepsis. Sous certaines conditions, E. coli serait capable de produire du méthane.
Escherichia coli (Migula, 1895) Castellani & Chalmers, 1919 è un batterio Gram-negativo ed è la specie tipo del genere Escherichia: se ne distinguono almeno 171 sierotipi, ognuno con una diversa combinazione degli antigeni O, H, K, F. Il nome deriva dal suo scopritore, il tedesco-austriaco Theodor Escherich. Appartiene al gruppo degli enterobatteri ed è usato come organismo modello dei batteri.
È una delle specie principali di batteri che vivono nella parte inferiore dell'intestino di animali a sangue caldo (uccelli e mammiferi, incluso l'uomo). Sono necessari per la digestione corretta del cibo. La sua presenza nei corpi idrici segnala la presenza di condizioni di fecalizzazione (è il principale indicatore di contaminazione fecale, insieme con gli enterococchi).
Il numero di cellule di E. coli nelle feci che un umano espelle in un giorno va da 10 a 100 milioni di unità formanti colonia (UFC) per grammo di feci[1]. Il genere Escherichia, insieme ad altri generi (Enterobacter, Klebsiella, Citrobacter, Serratia, ecc.), viene raggruppato sotto il nome di coliformi. Tecnicamente il "gruppo dei coliformi" comprende batteri aerobi e anaerobi non sporigeni.
Nel gruppo dei coliformi la specie Escherichia coli è ampiamente rappresentata ed è in esclusivo rapporto col tratto gastrointestinale dell'uomo e degli altri animali a sangue caldo, a differenza dei microrganismi appartenenti a diversi generi, tra cui Enterobacter, Klebsiella e Citrobacter (che si caratterizzano per una potenziale capacità di ricrescita una volta pervenuti nell'ambiente).
La specie Escherichia coli è un microrganismo a forma di bastoncello, gram-negativo, aerobio e anaerobio facoltativo, non sporigeno, che cresce alla temperatura di 44,5 °C, lattosio-fermentante, indolo-positivo in terreni contenenti triptofano, beta-D-glucuronidasi-positivo. In letteratura, la presenza di questo enzima è stata evidenziata nel 94-99,5 % dei biotipi di Escherichia coli, con l'eccezione dei sierotipi O157:H7. Alcuni ceppi di Escherichia coli enterotossigeni possono portare alla cosiddetta "diarrea del viaggiatore".
Nelle acque destinate al consumo umano, nelle acque di fonti termali, nelle acque adibite alla balneazione e in altri tipi di matrici (per es. alimenti, cosmetici) è prescritta l'assenza di Escherichia coli in quanto indicatore primario di contaminazione fecale. La mancata rispondenza al valore parametrico stabilito costituisce una non-conformità del prodotto (acqua, alimento, ecc.). Per la sua ricerca nell'ambiente sono stati elaborati, negli anni più recenti, metodi basati sull'attività enzimatica della beta-D-glucuronidasi (Saggio GUS), evidenziabile dall'idrolisi di beta-glucuronidi cromogeni o fluorogeni con rilascio di composti colorati o fluorescenti; o, allo stesso modo, si usa l'X-gal, facendo leva sull'enzima beta-galattosidasi. Queste caratteristiche, eliminando spesso la necessità di svolgere prove di conferma, permettono di ottenere risultati in tempi più rapidi e di giungere con maggiore accuratezza alla determinazione del microrganismo ricercato.
Anche se rappresenta un comune simbionte dell'intestino e ha un ruolo nel processo digestivo, ci sono situazioni in cui E. coli può provocare malattie nell'uomo e negli animali. Alcuni ceppi di E. coli sono l'agente eziologico di malattie intestinali ed extra-intestinali, come infezioni del tratto urinario, meningite, peritonite, setticemia e polmonite. Nel bovino da latte è noto per causare forme acute di mastite. Alcuni ceppi di E. coli sono tossigenici, producono cioè tossine che possono essere causa di diarrea. La dissenteria da E. coli è una comune tossinfezione alimentare, poiché viene contratta principalmente da alimenti contaminati. La contaminazione può avvenire da carni infette non adeguatamente cotte, da latte non pastorizzato e formaggi derivati, e da altri alimenti contaminati da feci. E. coli produce quattro tipi di tossine che si distinguono, per la diversa sensibilità al trattamento termico, in termolabile e termostabile, e per l'azione tossigena (tossine shiga e tossine emolitiche, HlyA).
La tossina termolabile, denominata LT, è molto simile nella struttura e nelle funzioni alla tossina del colera. Contiene una subunità 'A' e cinque subunità 'B' in un'olotossina. Le subunità B contribuiscono all'aderenza e all'entrata della tossina nelle cellule intestinali dell'ospite, dove la subunità A stimola le cellule a rilasciare acqua, provocando diarrea.
E. coli è il batterio più comune isolato nelle emocolture nei casi di batteriemia; la colonizzazione del torrente ematico può avvenire a causa di traumi intestinali, tumori del colon e del tenue. Le scarse misure igieniche nel posizionamento di cateteri o di accessi venosi centrali sono comuni cause di batteriemie da E. coli.
E. coli è comune causa di uretrite e cistite in persone anziane, diabetiche e cateterizzate. Grazie alle fimbrie formanti fasci, ai pili P e alle fimbrie Dr, E. coli riesce ad ancorarsi saldamente all'epitelio di uretra e vescica, resistendo al flusso urinario. Questa condizione è necessaria ma non sufficiente all'instaurarsi dell'infezione. Questa è infatti provocata maggiormente dall'emolisina A (HlyA), in grado di ledere l'epitelio urinario e promuovere chemiotassi, infiammazione e invasione dei tessuti.
E. coli, soprattutto il sierotipo K1, è il principale isolato (insieme con gli streptococchi di gruppo B) nelle emocolture di bambini con età inferiore a 5 anni affetti da meningite.
Un "ceppo" di E. coli è un gruppo con caratteristiche particolari in grado di renderlo riconoscibile da altri ceppi di E. coli, analogamente a come si riescono a distinguere cani appartenenti a razze diverse. Diversi ceppi di E. coli vivono in differenti specie animali, così è possibile stabilire se il materiale fecale nelle acque proviene, ad esempio, da uomini o da uccelli.
Nuovi ceppi di E. coli sorgono continuamente dal processo biologico naturale della mutazione, e alcuni di questi ceppi hanno caratteristiche che possono essere nocive per un animale ospite. Sebbene nella maggior parte degli umani adulti un ceppo patogeno non provocherebbe probabilmente altro che diarrea, e potrebbe non dare alcun sintomo, in bambini piccoli o in persone malate o debilitate da malattie recenti, o in persone sotto particolari cure, un nuovo ceppo potrebbe provocare malattie serie e persino la morte. Un esempio di ceppo particolarmente virulento di E. coli è E.coli O157:H7.
Le infezioni del tratto urinario (UTI) rappresentano una delle più comuni infezioni causate da batteri in grado di risalire l'uretra fino alla vescica. L'infezione della vescica prende il nome di cistite. Se l'infezione batterica si diffonde ai reni e agli ureteri, si parla di pielonefrite. La cistite è considerata un'infezione delle basse vie urinarie, la pielonefrite interessa le alte vie urinarie ed è più grave. I ceppi di E. coli uropatogeni (UPEC) sono responsabili di circa il 90% delle cistiti non complicate acquisite in comunità. Nel primo stadio del processo infettivo, le fimbrie rappresentano il fattore adesivo che consente a E. coli di aderire alle cellule uro-epiteliali. Alcuni studi hanno suggerito che il consumo di prodotti a base di mirtillo (Vaccinium macrocarpon) sono utili nella prevenzione delle infezioni del tratto urinario. Le proantocianidine presenti nel mirtillo contengono legami di tipo A e sono responsabili dell'azione preventiva nei confronti dell'adesione dei ceppi di E. coli, che posseggono fimbrie di tipo P, alle cellule uro-epiteliali.
L'E. coli enteropatogeno (EPEC) fu il primo patogeno associato con la malattia diarroica ed è tuttora una causa importante nei pazienti in età pediatrica. La tipizzazione sierologica degli antigeni O non risulta utile nel caso di EPEC. La malattia è causata dal fatto che questi ceppi sono in grado di aderire all'epitelio dell'intestino tenue e di interferire con l'assorbimento delle sostanze; questo provoca la formazione di un ambiente iperosmolare nel lume dell'intestino, un conseguente richiamo d'acqua e, infine, la diarrea. Il quadro di adesione all'epitelio e la distruzione dei microvilli giustifica la definizione di istopatologia A/E (attachment/effacement). I ceppi di E. coli che esprimono bfp possono stabilire dei blandi legami con la tonaca mucosa, consentendo al sistema di secrezione di tipo 3 di installare sulla membrana degli enterociti il recettore traslocato per l'intimina. Quest'ultimo permette il legame tenace di E. coli, grazie all'intimina espressa sulla membrana del batterio.
Questi ceppi (E. coli enterotossigeno) sono in grado di provocare gastroenteriti molto gravi, soprattutto nei viaggiatori e nelle persone che hanno ingerito cibi o liquidi contaminati da feci. Questi ceppi producono sia tossine labili al calore (LT-1 e LT-2), sia tossine stabili al calore (StA e Stb). LT-1 è formata da una subunità A e da cinque subunità B; queste ultime sono in grado di legare un ganglioside con un residuo di acido sialico (GM1) particolarmente espresso dagli enterociti. Il legame promuove l'internalizzazione della subunità A, una proteina con attività ADP - ribosiltransferasica nei confronti di una proteina G stimolatoria, in grado di attivare l'adenilato ciclasi. Gli elevati livelli di amp ciclico, portano a un rapido rilascio dei soluti nel lume intestinale; questi richiamano acqua e il quadro clinico connesso è la diarrea. Analogamente a LT-1, StA lega GM1; tuttavia la sua attività influenza la guanilato ciclasi e non l'adenilato ciclasi. Si noti come l'attività di LT-1 assomigli all'attività della tossina di Vibrio cholerae; tuttavia, LT-1 ha un'attività molto minore. I geni che codificano per LT-1 e per StA si trovano in un plasmide associati ai geni che codificano per AAF/1, AAF/2 e AAF/3, importanti fattori di adesione in grado di garantire una colonizzazione più duratura dei ceppi ETEC (i fattori di virulenza sono quindi trasmissibili). Negli USA vengono registrati circa 80.000 casi l'anno.
I ceppi di E. coli enteroemorragico sono i principali responsabili di malattia nei paesi industrializzati. Si calcola che questi batteri causino circa 73.000 casi d'infezione e quasi 60 morti ogni anno negli USA. Circa 50 sierotipi causano malattia; tuttavia il sierogruppo principalmente responsabile è O157:H7. L'ingestione di meno di 100 bacilli può causare la malattia; questa è stata associata al consumo di carne di manzo o pollo[2] non ben cotta, di latte non pastorizzato, di succhi di frutta contaminati (ad esempio da feci bovine) e di verdura cruda.
La malattia si manifesta a carico dell'intestino crasso dopo un periodo di incubazione di 3-4 giorni durante i quali comincia a comparire una diarrea non sanguinolenta. Circa al terzo giorno compaiono forti dolori addominali accompagnati da diarrea sanguinolenta. I ceppi EHEC sono in grado di promuovere un'istopatologia A/E (vedi EPEC) e di secernere la tossina Stx-1 (identica alla tossina di Shigella) insieme con la tossina Stx-2 (60% di analogia alla tossina di Shigella).
Entrambe le tossine sono codificate da fagi lisogeni e hanno una subunità A e cinque subunità B, in grado di legarsi al globotriaosilceramide o Gb3. Il legame con questo recettore promuove l'internalizzazione della subunità A nell'enterocita; questa è in grado di legarsi al frammento di RNA ribosomiale 28s, bloccando la sintesi proteica. La distruzione degli enterociti, accompagnata da una diminuzione della capacità di assorbimento, comporta la presenza di una diarrea molto liquida e sanguinolenta. La produzione di Stx-2 si associa spesso (nel 10% dei ragazzi con diarrea da EHEC) alla sindrome emolitico-uremica (HUS in inglese o SEU in italiano), caratterizzata da:
Infatti Stx-2 è in grado di legarsi con maggior affinità al Gb3 espresso dalle cellule renali, provocandone la distruzione. Inoltre le tossine Stx sono in grado di stimolare la produzione di TNF-α e interleuchina-6 che, oltre a sostenere il quadro infiammatorio, promuovono l'esposizione di Gb3. E. coli O157:H7, al contrario degli altri sierotipi, non fermenta il sorbitolo; questo permette l'identificazione nei terreni agar Mac Conkey contenenti sorbitolo (si valutano le colonie che mancano dell'attività fermentativa.
Tuttavia l'esame colturale deve essere affiancato dall'identificazione delle tossine mediante test immunoenzimatici commerciali. I ceppi O124, O143 e O164 (E. coli enteroinvasivi), strettamente simili a Shigella, sono in grado di provocare diarrea sanguinolenta (sono inoltre presenti leucociti), crampi addominali e febbre. Una serie di geni espressi in un plasmide, denominati pInv, codificano per una serie di proteine di membrana in grado di promuovere la fagocitosi e la lisi intracellulare del vacuolo fagocitico, con proliferazione del batterio all'interno del citoplasma degli enterociti dell'intestino crasso. Questo comporta depolimerizzazione dell'actina (come listeria), lisi cellulare ed emorragia. I quadri più gravi evolvono in ulcerazioni dell'intestino.
E. coli enteroaggreganti sono coinvolti in una diarrea acquosa persistente nei viaggiatori e negli infanti dei paesi in via di sviluppo. Questi ceppi esprimono sia Bfp sia AAF/1, AAF/2 e AAF/3 (tutti codificati da un plasmide), fattori di adesione in grado di promuovere la colonizzazione dell'intestino tenue, con stimolazione della produzione di muco. Questo forma un biofilm in grado di isolare e aggregare i batteri. In seguito all'aggregazione si ha riduzione della lunghezza dei microvilli, infiltrazione mononucleata ed emorragia.
E. coli diffusamente aderenti provocano una diarrea acquosa benigna nei bambini di età compresa tra 1 e 5 anni. Il quadro è provocato da un allungamento dei microvilli degli enterociti dell'intestino tenue, con inglobamento dei batteri negli enterociti.
Trattandosi di un batterio molto studiato e con DNA facilmente modificabile è molto utilizzato in processi biotecnologici, cioè di recombineering del DNA con l'inserimento di DNA di altri batteri per ottenere organismi non presenti e non ottenibili in natura, allo scopo di produrre sostanze con processi biotecnologici.
Escherichia coli (Migula, 1895) Castellani & Chalmers, 1919 è un batterio Gram-negativo ed è la specie tipo del genere Escherichia: se ne distinguono almeno 171 sierotipi, ognuno con una diversa combinazione degli antigeni O, H, K, F. Il nome deriva dal suo scopritore, il tedesco-austriaco Theodor Escherich. Appartiene al gruppo degli enterobatteri ed è usato come organismo modello dei batteri.
È una delle specie principali di batteri che vivono nella parte inferiore dell'intestino di animali a sangue caldo (uccelli e mammiferi, incluso l'uomo). Sono necessari per la digestione corretta del cibo. La sua presenza nei corpi idrici segnala la presenza di condizioni di fecalizzazione (è il principale indicatore di contaminazione fecale, insieme con gli enterococchi).
Il numero di cellule di E. coli nelle feci che un umano espelle in un giorno va da 10 a 100 milioni di unità formanti colonia (UFC) per grammo di feci. Il genere Escherichia, insieme ad altri generi (Enterobacter, Klebsiella, Citrobacter, Serratia, ecc.), viene raggruppato sotto il nome di coliformi. Tecnicamente il "gruppo dei coliformi" comprende batteri aerobi e anaerobi non sporigeni.
Nel gruppo dei coliformi la specie Escherichia coli è ampiamente rappresentata ed è in esclusivo rapporto col tratto gastrointestinale dell'uomo e degli altri animali a sangue caldo, a differenza dei microrganismi appartenenti a diversi generi, tra cui Enterobacter, Klebsiella e Citrobacter (che si caratterizzano per una potenziale capacità di ricrescita una volta pervenuti nell'ambiente).
La specie Escherichia coli è un microrganismo a forma di bastoncello, gram-negativo, aerobio e anaerobio facoltativo, non sporigeno, che cresce alla temperatura di 44,5 °C, lattosio-fermentante, indolo-positivo in terreni contenenti triptofano, beta-D-glucuronidasi-positivo. In letteratura, la presenza di questo enzima è stata evidenziata nel 94-99,5 % dei biotipi di Escherichia coli, con l'eccezione dei sierotipi O157:H7. Alcuni ceppi di Escherichia coli enterotossigeni possono portare alla cosiddetta "diarrea del viaggiatore".
Escherichia coli is een gramnegatieve staafvormige bacterie en is een van de meest voorkomende facultatief anaerobe bacteriën in de dikke darmen van warmbloedige dieren, zoals zoogdieren en is nodig voor het verteren van voedsel. Het is een enterobacterie die vaak gebruikt wordt als model voor bacteriën in het algemeen. De bacterie is genoemd naar de Duitse microbioloog Theodor Escherich.
Gemiddeld komen zo'n tien miljard van deze bacteriën per dag via de ontlasting van de mens naar buiten. Als E. coli (de gebruikelijke afkorting) in water wordt aangetroffen is dat een indicatie dat het water met uitwerpselen vervuild is.
Behalve vertering heeft de symbiose van warmbloedige dieren met E. coli nog een andere functie: het produceren van vitamine K. Deze stof is nodig om in de lever trombinogeen te maken en zodoende de bloedstolling te laten functioneren. Ook speelt deze vitamine een rol in de calciumhuishouding. Een overdosis van deze stof is vrijwel onmogelijk, daarom kan E. coli ook in zo groten getale voorkomen, maar een gebrek is wel degelijk mogelijk. Na langdurig gebruik van antibiotica (bacteriedodende middelen) kan deze bacterie flink uitgedund worden, waardoor een gebrek aan vitamine K optreedt, wat vervolgens leidt tot ontwrichting van de bloedstolling. Bloedneuzen en zelfs darmbloedingen kunnen het gevolg zijn.
Escherichia coli mag dan "goedaardig" genoemd worden, maar als deze bacteriën op de verkeerde plaatsen in het lichaam komen kunnen ze wel degelijk gevaar opleveren:
Een andere mogelijkheid voor gevaar is wanneer gevaarlijke, gemuteerde, soorten van deze bacterie het lichaam binnendringen.
Een voorbeeld is Escherichia coli O157:H7, een vrij courante veroorzaker van een bacteriële voedselvergiftiging, onder andere via niet goed doorbakken vlees. Jaarlijks zijn er volgens schattingen in de Verenigde Staten alleen al, gemiddeld zo'n 73.000 gevallen, waarvan 61 dodelijk. Een 2 tot 7 procent van de patiënten ontwikkelen ernstige bijkomende symptomen als nierfalen en bloedarmoede het hemolytisch-uremisch syndroom (HUS) - ook wel 'hamburger disease' genoemd.
Ook zijn er pathogene serotypevariaties van E. coli zoals EHEC, ETEC, EIEC, EAEC en EPEC. De letters in de afkortingen betekenen:
E. coli wordt al heel lang gebruikt voor allerlei onderzoek (de variant die tegenwoordig het meest in laboratoriumonderzoek wordt gebruikt, werd al in 1927 geïsoleerd). Daar zijn verschillende redenen voor:
De Universiteit van Chicago heeft een computersimulatie van E. coli gemaakt om tot een groter begrip te komen van het verband tussen de interne biochemie en het gedrag van het organisme. Deze simulatie heeft de naam AgentCell.
Een nadeel van E. coli voor de productie van transgene eiwitten kan zijn dat ze niet zo gevouwen zijn als in een eukaryoot, en daardoor niet actief zijn. Met name eiwitten die geglycosyleerd zijn (dat wil zeggen dat er suikergroepen aan vast zitten) kunnen door de veranderde hydrofiliteit een andere structuur aannemen. Om zulke suikergroepen te krijgen kan een eukaryoot, meestal een gistcel als Saccharomyces cerevisiae of Pichia pastoris, gebruikt worden.
Wegens de lange geschiedenis in de laboratoriumcultuur en het gemak om de bacterie te manipuleren, speelt E.coli een belangrijke rol in de hedendaagse biotechnologie. Het werk van Stanley Norman Cohen en Herbert Boyer met E. coli, waarbij ze gebruikmaken van plasmiden en restrictie enzymen om zo recombinant DNA te creëren, diende als basis voor de biotechnologie.
E.coli is een zeer veelzijdig organisme en is een goede gastheer voor de productie van verschillende proteïnen. Onderzoekers kunnen genen van andere micro-organismen binnenin de bacterie brengen met behulp van plasmides, om zo massale hoeveelheden proteïnen te genereren in fermentatieprocessen. Een van de eerste bruikbare toepassingen van de recombinant DNA technologie was het manipuleren van E.coli om het menselijke insulinehormoon te produceren.[2]
Genetisch gewijzigde E.coli worden verder ook gebruikt in de ontwikkeling van vaccins, in de bioremediatie en in de productie van geïmmobiliseerde enzymen.[3] E. coli kan daarentegen niet gebruikt worden om sommige grotere en meer complexe proteïnen te produceren, zoals proteïnen die vele zwavelbruggen bevatten.
Met behulp van de klassieke microbiologie wordt deze bacterie ook als indicator in de voedingsindustrie gebruikt. Een hoge concentratie E. coli in een product duidt namelijk op een grote kans dat het product ook andere pathogenen bevat.
Escherichia coli is een gramnegatieve staafvormige bacterie en is een van de meest voorkomende facultatief anaerobe bacteriën in de dikke darmen van warmbloedige dieren, zoals zoogdieren en is nodig voor het verteren van voedsel. Het is een enterobacterie die vaak gebruikt wordt als model voor bacteriën in het algemeen. De bacterie is genoemd naar de Duitse microbioloog Theodor Escherich.
Gemiddeld komen zo'n tien miljard van deze bacteriën per dag via de ontlasting van de mens naar buiten. Als E. coli (de gebruikelijke afkorting) in water wordt aangetroffen is dat een indicatie dat het water met uitwerpselen vervuild is.
Escherichia coli (ofte forkortet som E. coli) er en gram-negativ bakterie, først beskrevet av (og oppkalt etter) den tyske barnelegen Theodor Escherich, og er fakultativt anaerob, den kan leve i oksygenrike og oksygenfattige miljøer. Escherichia coli finnes hovedsakelig i vanlig tarmflora i tykktarmen hos pattedyr inkludert hos mennesker. Der har den en viktig funksjon i fordøyelsen. Den kan likevel også være årsak til sykdommer, som ved infeksiøse gastroenteritter (diarésykdom) og urinveisinfeksjoner. E. coli er sannsynligvis den bakterien som er best dokumentert i den vitenskapelige litteratur. Dette har sammenheng med at den er lett å dyrke i laboratorier, til forskjell fra en del andre tarmbakterier.
Noen stammer produserer giftstoffer (enterotoksiner). Noen dyrearter kan være immune mot en slik gift, mens andre kan bli syke av den. Enkelte E.colistammer som finnes i tarmen hos friske storfe kan for eksempel føre til alvorlig sykdom dersom de slår seg ned i tarmen hos mennesker.
Når en først utvikler symptomer på E. Coli-forgiftning, utvikler sykdommen seg raskt med nedsatt immunforsvar og nyre- og leversvikt. Kun 3 av 10 personer under 18 år overlever sykdommen.
Escherichia coli benyttes som indikator på avføringsforurensing av drikkevann. Escherichia coli er eutrof og vokser derfor dårlig i vann. Påvisning av Escherichia coli i drikkevann indikerer derfor nylig forurensing. Escherichia coli er sjelden selv patogen, men påvisning av den kan tyde på at det finnes andre patogene mikroorganismer og parasitter i vannet.
Noen ganger kan imidlertid Escherichia coli ikke brukes som indikator på forurensning med avføring, som f.eks. i miljøer der Escherichia coli gror godt, bl.a. ved papirfabrikker.
Det er tre muligheter for at den ellers harmløse Escherichia coli kan forårsake sykdom:
Utdypende artikkel: E. coli-saken i 2006
I 2006 ble til sammen 18 personer, hvorav 16 barn, i Norge syke etter å ha blitt smittet av E. coli O103 fra infisert morrpølse. Ett av barna døde.
Også i 2009 ble fire barn i Norge syke etter å ha blitt smittet av Escherichia coli O157:H7. En toåring i Sogn og Fjordane døde. Smittekilden ble ikke funnet.[2][3]
Utdypende artikkel: E. coli-utbruddet i 2011
I mai 2011 oppsto et sykdomsutbrudd i Tyskland som skyldtes smitte av Escherichia coli O104:H4.
Escherichia coli (ofte forkortet som E. coli) er en gram-negativ bakterie, først beskrevet av (og oppkalt etter) den tyske barnelegen Theodor Escherich, og er fakultativt anaerob, den kan leve i oksygenrike og oksygenfattige miljøer. Escherichia coli finnes hovedsakelig i vanlig tarmflora i tykktarmen hos pattedyr inkludert hos mennesker. Der har den en viktig funksjon i fordøyelsen. Den kan likevel også være årsak til sykdommer, som ved infeksiøse gastroenteritter (diarésykdom) og urinveisinfeksjoner. E. coli er sannsynligvis den bakterien som er best dokumentert i den vitenskapelige litteratur. Dette har sammenheng med at den er lett å dyrke i laboratorier, til forskjell fra en del andre tarmbakterier.
Noen stammer produserer giftstoffer (enterotoksiner). Noen dyrearter kan være immune mot en slik gift, mens andre kan bli syke av den. Enkelte E.colistammer som finnes i tarmen hos friske storfe kan for eksempel føre til alvorlig sykdom dersom de slår seg ned i tarmen hos mennesker.
Når en først utvikler symptomer på E. Coli-forgiftning, utvikler sykdommen seg raskt med nedsatt immunforsvar og nyre- og leversvikt. Kun 3 av 10 personer under 18 år overlever sykdommen.
Pałeczka okrężnicy (łac. Escherichia coli) – Gram-ujemna względnie beztlenowa bakteria należąca do rodziny Enterobacteriaceae. Wchodzi w skład fizjologicznej flory bakteryjnej jelita grubego człowieka oraz innych zwierząt stałocieplnych. W jelicie ta symbiotyczna bakteria spełnia pożyteczną rolę, uczestnicząc w rozkładzie pokarmu, a także przyczyniając się do produkcji witamin z grupy B i K. Pałeczka okrężnicy w określonych warunkach wykazuje chorobotwórczość dla człowieka, wywołując głównie schorzenia: układu pokarmowego i moczowego.
Nazwa bakterii pochodzi od nazwiska jej odkrywcy, austriackiego pediatry i bakteriologa Theodora Eschericha.
Escherichia coli spotyka się w jelicie zwierząt, w tym człowieka, jak również powszechnie w glebie i wodzie, gdzie trafiają z wydzielinami i kałem. Obecność Escherichia coli w wodach powierzchniowych (tzw. miano Coli) jest często stosowanym wskaźnikiem ich zanieczyszczenia[1]. Bakterie Escherichia coli mogą kolonizować skórę i błony śluzowe jamy ustnej oraz układu oddechowego.
W wodzie zwykle przeżywa krótko, m.in. ze względu na brak właściwego pożywienia i działanie promieniowania ultrafioletowego. W warunkach eksperymentalnych imitujących warunki spotykane w wodach powierzchniowych regionu Wielkich Jezior populacje Escherichia coli o małym zagęszczeniu zanikają w znaczącym stopniu po 6 godzinach, a o dużym zagęszczeniu – po dwóch dniach. W obecności mat tworzonych przez glony nitkowate (gałęzatkę), które wydzielają substancje organiczne, którymi może się pożywiać Escherichia coli, a ponadto zatrzymują część promieniowania, populacja Escherichia coli może przetrwać kilkakrotnie dłużej (od kilkunastu godzin do kilkunastu dni)[2].
Escherichia coli jest Gram-ujemną, względnie beztlenową pałeczką o długości około 2 μm i średnicy ok. 0,8 μm. Wewnątrz komórki tej bakterii znajduje się 1-4 identycznych łańcuchów DNA (w zależności od jej aktywności podziałowej) oraz od 15 tys. do 30 tys. rybosomów. Inne organella komórkowe pałeczki okrężnicy to m.in.: wakuole, ziarnistości i drobiny substancji tłuszczowych. Bakteria ta posiada wici, fimbrie, pilusy oraz fimbrie płciowe (pod warunkiem posiadania plazmidów płciowych – F+). Ważne klinicznie geny oporności zlokalizowane są na plazmidach w tym często na plazmidach F+, co sprzyja ich poziomemu transferowi. Podział komórek w sprzyjających warunkach trwa około 20 minut.
Wytrzymałość Escherichia coli na czynniki środowiskowe jest stosunkowo mała. Ginie ona po 20 minutach ogrzewania w temperaturze 60 °C, wrażliwa jest na wszystkie znane środki dezynfekcyjne. Jednakże w środowisku o temp. niższej i odpowiedniej wilgotności utrzymuje się miesiącami. W kale o temp. 0 °C może zachować żywotność ponad rok.
Escherichia coli należy do organizmów modelowych wśród bakterii. Jej budowa, genetyka i metabolizm są dobrze poznane i wykorzystywane w badaniach genetycznych. Znalazła szczególne wykorzystanie w modyfikacjach genetycznych w biotechnologii, wykorzystywana w celach przemysłowych (np. w produkcji ludzkiego hormonu – insuliny).
Wykorzystywana jest ze względu na tanią hodowlę przy jednoczesnym otrzymywaniu dużej liczby bakterii na niewielkiej powierzchni. W ciągu 20 minut uzyskuje się następne pokolenie, a powstające mutacje są łatwe do identyfikacji i badań. Kolisty chromosom został kilkudziesięciokrotnie zsekwencjonowany. Najczęściej badanym szczepem jest K-12 posiadający 4,639,221 par zasad kodujący 4,377 genów[potrzebny przypis]. Proteom określono w 2014 na 4303 białek[3]. Pangenom od ~25 Mlat[4] uzbierał> 16k genow gdzie ponad połowa sekwencji pochodziła z HTG.
Dzięki Escherichia coli zrozumiano takie procesy jak:
Gatunek Escherichia coli dzieli się na serotypy na podstawie zróżnicowania budowy antygenów.
Szczepy bakterii różnicuje się na podstawie cech antygenowych:
Wyróżnia się 171 antygenów O, około 80 antygenów K i ponad 50 antygenów H, dzięki którym wyszczególniono ponad 180 typów serologicznych tego drobnoustroju[potrzebny przypis].
Pod względem oddziaływania na organizm człowieka oraz przebiegu infekcji rozróżnia się 6 szczepów Escherichia coli[5][6]:
Czynniki zjadliwości Escherichia coli dotyczą głównie ich zdolności do przylegania i adhezji do nabłonka przewodu pokarmowego oraz wytwarzania toksyn przez niektóre szczepy. Adhezji sprzyjają fimbrie, z czego najgroźniejsze są typy I oraz P (powinowactwo do dróg moczowych) i typu S (powinowactwo do naczyń mózgowych) Innym czynnikiem zjadliwości jest otoczka o właściwościach antyfagocytarnych, zwana antygenem K, adhezyny CEA, endotoksyna (LPS) oraz α-hemolizyny. Niektóre szczepy wytwarzają egzotoksyny oraz toksynę typu Shiga. Szczepy hemolityczne bakterii są bardziej chorobotwórcze niż szczepy niehemolityczne. Hemolizyna ma bowiem właściwości toksyny o działaniu nie tylko hemolitycznym, ale także dermonekrotycznym i koagulującym plazmę.
Młode organizmy zwierzęce są szczególnie narażone na zachorowania – przyjmuje się, że około połowa padnięć zwierząt młodych jest spowodowana kolibakteriozami. Wynika to z mniejszej odporności młodzieży. Chorobotwórczość Escherichia coli zależy od jej inwazyjności i możliwości wytwarzania toksyn. Inwazyjne szczepy tej bakterii mają zdolność wnikania do tkanek i wywoływania odczynów zapalnych, dlatego są przyczyną zapaleń przewodu pokarmowego i posocznic. Szczepy toksynotwórcze produkują w jelitach duże ilości toksyn, prowadząc do enterotoksemii. W organizmie zwierząt chorych mogą znajdować się także różne serotypy zarazka. Enterotoksyna działając na śluzówkę jelit w połączeniu z endotoksyną powoduje zapalenie jelit i objawy zatrucia. Neurotoksyna wchłonięta do organizmu z przewodu pokarmowego prowadzi do wystąpienia charakterystycznych objawów ze strony układu nerwowego oraz przyczynia się do powstania chorób, np. choroby obrzękowej u świń.
Bakterie Escherichia coli, które są nieszkodliwe w jelicie, mogą powodować schorzenia innych układów:
Wobec bakterii Escherichia coli aktywność zachowują:
Podczas operacji na jelicie grubym zaleca się podać profilaktycznie antybiotyk, np. metronidazol z cefazoliną. Antybiotyk zazwyczaj jest podawany 2 godziny przed operacją (doustny) lub 2 godziny po jej rozpoczęciu (dożylny)[8].
Pałeczka okrężnicy (łac. Escherichia coli) – Gram-ujemna względnie beztlenowa bakteria należąca do rodziny Enterobacteriaceae. Wchodzi w skład fizjologicznej flory bakteryjnej jelita grubego człowieka oraz innych zwierząt stałocieplnych. W jelicie ta symbiotyczna bakteria spełnia pożyteczną rolę, uczestnicząc w rozkładzie pokarmu, a także przyczyniając się do produkcji witamin z grupy B i K. Pałeczka okrężnicy w określonych warunkach wykazuje chorobotwórczość dla człowieka, wywołując głównie schorzenia: układu pokarmowego i moczowego.
Nazwa bakterii pochodzi od nazwiska jej odkrywcy, austriackiego pediatry i bakteriologa Theodora Eschericha.
Escherichia coli (/eʃe'ɾikia 'kɔli/, mais conhecida pela abreviatura E. coli), é uma bactéria bacilar Gram-negativa que se encontra normalmente no trato gastrointestinal inferior dos organismos de sangue quente (endotérmicos). A maioria das estirpes de E. coli são inofensivas, mas alguns sorotipos podem causar graves intoxicações alimentares nos seres humanos, e são ocasionalmente responsáveis pela recolha de produtos alimentícios devido à sua contaminação.[1][2] As estirpes inofensivas constituem parte da microbiota intestinal humana normal, e podem ser benéficas para os seus hospedeiros ao produzirem vitamina K2,[3] e impedirem que ali se estabeleçam bactérias patogénicas.[4][5]
E. coli e as bactérias relacionadas constituem cerca de 0,1% da microbiota intestinal,[6] e a transmissão fecal-oral é a principal via utilizada pelas cepas patogénicas que causam doenças. As células desta bactéria podem sobreviver fora do corpo por um tempo bastante limitado, o que faz com que sejam um organismo indicador ideal para comprovação da contaminação fecal em amostras quando extraídas para o meio ambiente.[7] Entretanto, existe um crescente número de investigações que identificaram a E. coli persistentes no meio ambiente, capazes de sobreviver por um longo período de tempo fora de um hospedeiro.[8]
A bactéria também pode crescer e ser cultivada facilmente e a baixo custo em laboratório, e tem sido intensamente investigada há mais de 60 anos. Assim, pode-se dizer que a E. coli é o organismo modelo procariota mais estudado, e uma importante espécie no campo da biotecnologia e microbiologia, onde serviu como organismo hóspede para a maioria dos trabalhos sobre o ADN recombinante. Em condições favoráveis, leva apenas 20 minutos para se reproduzir.[9]
Juntamente com o Staphylococcus aureus é a mais comum e uma das mais antigas bactérias simbiontes da humanidade. Foi descoberta pelo alemão-austríaco Theodor Escherich, em 1885.
A E. coli assume a forma de um bacilo e pertence à família das Enterobacteriaceae. São aeróbias e anaeróbias facultativas. O seu habitat natural é o lúmen intestinal dos seres humanos e de outros animais de sangue quente. Possui múltiplos flagelos dispostos em volta da célula.[10] Algumas espécies sao tao similares ao Shigella e tao diferentes entre si que alguns biólogos recomendam que ambos sejam re-classificados. [11]
A E. coli é um dos poucos seres vivos capazes de produzir todos os componentes de que são feitos, a partir de compostos básicos e fontes de energia suficientes. Ela é lactase positiva, uma enzima fermentadora de açúcares que é grandemente responsável pela flatulência de cada pessoa, especialmente após o consumo de leite e seus derivados.
Possuem fímbrias ou adesinas que permitem a sua fixação, impedindo o arrastamento pela urina ou diarreia. Muitas produzem exotoxinas. São susceptiveis aos ambientes secos, aos quais não resistem. Possuem lipopolissacarídeo (LPS), como todas as bactérias Gram-negativas. Esta molécula externa ativa o sistema imunitário de forma desproporcionada e a vasodilatação excessiva provocada pelas citocinas produzidas pode levar ao choque séptico e morte em casos de septicémia.
Na E. coli, o genoma tem quase 5 milhões de pares de bases e vários milhares de genes codificando mais de 4000 proteínas (o genoma humano tem 3 bilhões de pares de bases e cerca de 27 mil proteínas).
Existem, enquanto parte da microbiota normal no intestino, em grandes números. Cada pessoa evacua em média, com as fezes, um trilhão de bactérias E.coli todos os dias. A doença é devida à disseminação, noutros órgãos, das estirpes intestinais normais; ou nos casos de enterite ou meningite neonatal à invasão do lúmen intestinal por estirpes diferentes daquelas normais no indivíduo.
A presença da E.coli em água ou alimentos é indicativa de contaminação com fezes humanas (ou mais raramente de outros animais). A quantidade de E.coli em cada mililitro de água é uma das principais medidas usadas no controlo da higiene da água potável municipal, preparados alimentares e água de piscinas. Esta medida é conhecida oficialmente como índice coliforme da água.
A estirpe de E.coli que existe normalmente nos intestinos de um determinado indivíduo é bem conhecida e controlada pelo seu sistema imunitário, e raramente causa problemas excepto quando há debilidade do indivíduo. A maioria das doenças é devido a E.coli vindas de indivíduos diferentes e portanto de estirpe diferente, não reconhecida pelos linfócitos. As intoxicações alimentares em particular são quase sempre devidas a bactérias de estirpes radicalmente diferentes. O subgrupo Escherichia coli enterotoxigênica (ETEC) é responsável pela grande maioria das intoxicações alimentares entre turistas e viajantes. [carece de fontes?]
No Brasil, entre 2000 e 2002 a quantidade de infectados subiu de 12% para 18% em Santa Catarina. Essa quantidade de infectados está próxima a média de outros estados.[12]
Uma de suas formas de classificar é pelos sintomas que causam quando são patogênicas:
O E. coli é transmitido por via orofecal a seres humanos e outros animais, geralmente por[15]:
A E.coli está entre as principais causas de:
O diagnóstico é feito pela cultura de amostras dos líquidos infectados e observação microscópica com análises bioquímicas. São usadas técnicas genéticas para identificar genes presentes no genoma da E.coli. Pode causar dor no final ao urinar, ardência. Causa hematuria (que é sangue), odor fétido.
A E.coli pode ser resistente a um número crescente de antibióticos, mas uma estirpe raramente é a mais de dois ou três fármacos. A Escherichia coli é mais vulnerável à amicacina (98,6 por cento), gentamicina (96,2 por cento), nitrofurantoína (96,3 por cento), e às quinolonas (90,9 por cento) e norfloxacina (89,8 por cento), sendo mais resistente à sulfametoxazol-trimetoprima (50,6 por cento). A escolha do antibiótico é feita por testes in vitro de susceptibilidade. É recomendado também repouso e beber muita água potável para repor os líquidos perdidos por diarreia, suor e vômito.[16]
Uma complicação grave possível é a síndrome hemolítico-urêmica (SHU) causada por substâncias tóxicas produzidas por algumas espécies de bactérias que destroem as células vermelhas do sangue, causando danos nos rins. Pode exigir cuidados intensivos, diálise renal e transfusões de sangue. Esse tipo de complicação é raro nos países desenvolvidos (apenas 1 caso em cada 100.000 habitantes por ano), mas são mais de 20 vezes mais comuns em países latinos e africanos.
O seu nome vem do descobridor, Theodor Escherich (1857-1911), um médico alemão-austríaco nascido na Baviera. A descoberta ocorreu em 1885 e o seu nome foi dado à bactéria em 1919.
A E.coli foi e é muito estudada enquanto modelo geral para os mecanismos biológicos das bactérias, na disciplina de biologia molecular, tendo também um papel muito importante em bioengenharia e microbiologia industrial.
A E. coli é usada pela engenharia genética para produzir proteínas recombinantes. Em 1977 foi usada pela primeira vez para produzir insulina "humana" em laboratório.
Na Coreia do Sul, um experimento na universidade KAIST (Korea Advanced Institute of Science and Technology),[17] produziu pequenas quantidades de biocombustível num processo que empregou micro-organismos.[18] [19] Anteriormente, micro-organismos geneticamente modificados geraram biodiesel, mas os pesquisadores coreanos conseguiram produzir biogasolina usando bactérias do tipo E.coli.[18] [19]
Escherichia coli (/eʃe'ɾikia 'kɔli/, mais conhecida pela abreviatura E. coli), é uma bactéria bacilar Gram-negativa que se encontra normalmente no trato gastrointestinal inferior dos organismos de sangue quente (endotérmicos). A maioria das estirpes de E. coli são inofensivas, mas alguns sorotipos podem causar graves intoxicações alimentares nos seres humanos, e são ocasionalmente responsáveis pela recolha de produtos alimentícios devido à sua contaminação. As estirpes inofensivas constituem parte da microbiota intestinal humana normal, e podem ser benéficas para os seus hospedeiros ao produzirem vitamina K2, e impedirem que ali se estabeleçam bactérias patogénicas.
E. coli e as bactérias relacionadas constituem cerca de 0,1% da microbiota intestinal, e a transmissão fecal-oral é a principal via utilizada pelas cepas patogénicas que causam doenças. As células desta bactéria podem sobreviver fora do corpo por um tempo bastante limitado, o que faz com que sejam um organismo indicador ideal para comprovação da contaminação fecal em amostras quando extraídas para o meio ambiente. Entretanto, existe um crescente número de investigações que identificaram a E. coli persistentes no meio ambiente, capazes de sobreviver por um longo período de tempo fora de um hospedeiro.
A bactéria também pode crescer e ser cultivada facilmente e a baixo custo em laboratório, e tem sido intensamente investigada há mais de 60 anos. Assim, pode-se dizer que a E. coli é o organismo modelo procariota mais estudado, e uma importante espécie no campo da biotecnologia e microbiologia, onde serviu como organismo hóspede para a maioria dos trabalhos sobre o ADN recombinante. Em condições favoráveis, leva apenas 20 minutos para se reproduzir.
Juntamente com o Staphylococcus aureus é a mais comum e uma das mais antigas bactérias simbiontes da humanidade. Foi descoberta pelo alemão-austríaco Theodor Escherich, em 1885.
Escherichia coli (nume abreviat: E. coli) este o bacterie Gram-negativă care trăiește în intestinele organismelor cu sânge cald.
E. coli este o bacterie lactozo-pozitivă (descompune lactoza), gram-negativă, oxidazo-negativă, ce apare la microscop sub formă de bastonașe (este un bacil). El face parte din grupa enterobacteriilor care trăiește ca epifit în tractusul digestiv. În unele cazuri de dezechilibrare a microflorei intestinale, aceste bacterii pot produce îmbolnăviri, printr-o înmulțire masivă sau apariția unor tulpini toxicogene. Bacteria a fost denumită în 1919 după numele bacteriologului germano-austriac Theodor Escherich(en), cel care a descoperit-o.
Bacteriile E. coli nu sunt întotdeauna limitate la intestin, și abilitatea lor de a supraviețui pentru perioade scurte de timp în afara corpului ce le face un organism-indicator ideal pentru a testa probe de mediu de contaminare fecală. Bacteria poate fi, de asemenea, cultivata cu ușurință și genetica sa este relativ simplă și ușor de manipulat printr-un proces mutagenic, făcându-l unul dintre cele mai bine studiate organisme procariote model, și o specie importanta în domeniul biotehnologiei și microbiologie.
Escherichia coli (nume abreviat: E. coli) este o bacterie Gram-negativă care trăiește în intestinele organismelor cu sânge cald.
Escherichia coli, förkortas ofta E. coli (kolibakterie), är en gramnegativ bakterieart som ingår i familjen Enterobacteriaceae. E. coli lever i de nedre delarna av tarmarna hos varmblodiga djur, inklusive fåglar och däggdjur. Det klarlades tidigt att den kan orsaka olika typer av infektioner i bland annat urinvägar, blodbanor, hjärnhinnor och tarmar. E. coli kan enkelt odlas på agarplattor och är den vanligaste bakteriella modellorganismen vilket gör den särskilt viktig inom molekylärbiologin.
E. coli är nödvändig för normal matsmältning och utgör en stor andel av tarmfloran (intestinala floran). Antalet E. coli-bakterier i en människas avföring varierar mellan 100 miljarder och 10 biljoner per gram avföring. Om E. coli påträffas i grundvatten är det troligt att detta förorenats med avföring från människa eller något annat varmblodigt djur.
Maj 2011 skedde ett utbrott av aggressiv E. coli O104:H4 i Tyskland. Senare har även personer i Frankrike och Sverige blivit smittade, med stor utredningsinsats som följd.
E. coli isolerades för första gången 1885, av Theodor Escherich,[1] en barnläkare och bakteriolog.[2]
E. coli och en mängd andra relativt närbesläktade bakteriarter finns i människans normala tarmflora. Sådana bakterier benämns ofta koliforma bakterier och brukar beskrivas som fakultativt anaeroba rörliga gramnegativa stavar. Hittills har man identifierat 4 olika så kallade ytantigensystem hos E. coli. Dessa benämns O (lipopolysackarid, cirka 190 [3] olika typer finns beskrivna)-, K (kapsel)-, H (flagell)- och F (fimbrie)-antigen. De olika antigenen ger bakterierna en del av deras karakteristiska egenskaper, exempelvis förmåga att orsaka feber (O), motstå kroppens immunförsvar (K), rörlighet (H) och förmåga att fästa på slemhinnor (F). En del av E. coli-bakterierna bildar så kallade enterotoxiner, det vill säga gifter som orsakar diarré. Vissa stammar kan orsaka blödande tarminfektioner och skada njurarna (så kallade EHEC-bakterier, Enterohemorragisk E. coli).
Livsmedel som äts utan att ha tillagats tillräckligt, eller rått, ökar risken för en infektion av E. coli, förutsatt att bakterierna förekommer i det djur från vilket livsmedlet har framställts. En livsmedelsprodukt som anses vara särskilt riskabel att konsumera är obehandlad mjölk. Det är dock ofarligt att dricka pastöriserad mjölk. En annan riskfylld produkt är vegetabiliska livsmedel. Detta då dessa kan vara förorenade med avföring från djur. Även i pälsen på djur finns mikroskopiska fekala partiklar som kan smitta via kontakt med handen, som vid ett senare tillfälle ofta kommer i kontakt med munnen. Den direkta överföringen av E. coli från en individ till en annan spelar en betydande roll. Det är nämligen tillräckligt med färre än hundra E. coli-bakterier för att en infektion ska uppstå. En person vars händer inte har tvättats tillräckligt efter ett besök på toaletten kan därför överföra bakterier till familjemedlemmar, eller andra personer, och därigenom orsaka en infektion av E. coli.[4]
Enklast diagnostiseras E. coli-infektioner genom att göra en bakteriologisk odling från den infekterade lokalen, till exempel urin eller blod. Eftersom urinvägsinfektioner oftast orsakas av E. coli behöver läkaren ofta inte företa odling av urin, utan kan behandla infektionen med antibiotika ändå.
Infektioner som orsakas av E. coli är behandlingsbara med ett flertal olika antibiotikatyper som pivmecillinam, ampicillin, cefalosporiner, trimetoprim, kinoloner. Mot ampicillin, som är ett slags penicillin, föreligger dock relativt ofta resistens, varför just detta antibiotikum används relativt sällan numera.
E. coli är en mycket vanlig modellorganism[5] som växer bra i enkelt sammansatta medier och kan enkelt odlas på agarplattor eller i flytande medier. I näringsbuljong fermenterar de oftast laktos under gasbildning inom 48 timmar vid 35 °C.
E. coli har ett 4,6 Mb stort genom som totalt innehåller ca 4400 gener.[6]
![]() Scholia har publikationer om Escherichia coli
Scholia har publikationer om Escherichia coli
Escherichia coli, förkortas ofta E. coli (kolibakterie), är en gramnegativ bakterieart som ingår i familjen Enterobacteriaceae. E. coli lever i de nedre delarna av tarmarna hos varmblodiga djur, inklusive fåglar och däggdjur. Det klarlades tidigt att den kan orsaka olika typer av infektioner i bland annat urinvägar, blodbanor, hjärnhinnor och tarmar. E. coli kan enkelt odlas på agarplattor och är den vanligaste bakteriella modellorganismen vilket gör den särskilt viktig inom molekylärbiologin.
E. coli är nödvändig för normal matsmältning och utgör en stor andel av tarmfloran (intestinala floran). Antalet E. coli-bakterier i en människas avföring varierar mellan 100 miljarder och 10 biljoner per gram avföring. Om E. coli påträffas i grundvatten är det troligt att detta förorenats med avföring från människa eller något annat varmblodigt djur.
Maj 2011 skedde ett utbrott av aggressiv E. coli O104:H4 i Tyskland. Senare har även personer i Frankrike och Sverige blivit smittade, med stor utredningsinsats som följd.
Genelde E. coli kısaltması ile veya koli basili olarak bilinen Escherichia coli (okunuşu Eşerihiya koli), memeli hayvanların kalın bağırsağında yaşayan bakteri türlerinden biridir. Normalde bağırsakta yaşadığı için, E. coli 'nin çevresel sularda varlığı dışkı kirlenmesinin bir belirtisidir.
E. coli, pediyatrist ve bakteriyolog olan Theodor Escherich tarafından bebek dışkılarında keşfedilmiştir ve adını ondan alır; coli, "kalın bağırsaktan" demektir. E. coli, genel olarak bakteri biyolojisinin anlaşılması amacıyla üzerinde sıkça çalışılmış bir model organizma olmuştur. Canlılar arasında hakkında en fazla şey bilinen organizma olduğu söylenebilir.
İnsanın bir günde dışkı yoluyla vücudundan geçen E. coli bakteri sayısı 100 milyar ila 10 trilyon arasındadır. Dışkıyı oluşturan bakteriler başlıca aerobik bakterilerdir, seçmeli anerobik E. coli hücrelerinin sayısı diğer bakteri türlerinin binde biri dolayındadır. Başka hayvanlarda etkisiz olan bazı E. coli tipleri insana bulaştıklarında hastalık yapabilirler. Bunların en ünlüsü sayılan O157:H7 adlı serotip kanlı ishale ve ölüme yol açabilir.
E. coli, normal bağırsak florasına aittir, biyolojik sınıflandırmada da bağırsaklarda yaşayan bakterilerden oluşan enterik bakteriler ailesinde yer alır. Bakteri çubuk şeklinde olup, boyutları 1-2 µm uzunluğunda ve 0.1-0.5 µm çapındadır.
E. coli Gram-negatif bir bakteri olduğundan endospor oluşturmaz, pastörizasyon veya kaynatma ile ölür. Memeli hayvanların bağırsaklarında büyümeye adapte olmuş olduğu için en iyi vücut sıcaklığında çoğalır.
Su arıtım sahasında E. coli yeni teknolojilerin gelişiminin en başından itibaren su kirliliğinin bir "göstergeci" olarak kullanılmıştır. Sudaki dışkı miktarı koliform endeks aracılığıyla ölçülmüştür. E. coli genelde zararsız olmasına rağmen kirlenmenin ölçütü olarak kullanılmasının nedeni, seçmeli (fakültatif) anaerob olmasından dolayı E. coli 'nin kolay kültürlenebilmesidir. Ayrıca, dışkıda bulunan E. coli sayısı patojen bakterilerin (örneğin tifo etmeni Salmonella typhi) sayısından çok daha fazladır. Su kirlenmesini belirlemek amacıyla yapılan nispeten basit testlerde E. coli 'ye benzeyen organizmalar için "koliform" terimi kullanılır. Ancak, bağırsaklarda yaşamayan bazı saprotrof bakteriler de koliform tanımına uyarlar. Ayrıca, dışkı kirlennmesi olmayan ortamlarda da E. coli 'nin var olabildiği gözlemlenmiştir. Buna rağmen, pratik nedenlerden dolayı dünyanın çoğu ülkesinde su temizliğiyle ilgili standartlar "koliform sayısı"na dayandırılır.
Bağırsak florasının normal bir üyesi olan E. coli ile konak organizma arasında uyumlu bir ilişki olduğundan bakteri normalde hastalık yapmaz. Ancak, ortama geçmesi halinde, ki bu aynı organizmada başka bir organ olabilir (idrar yolu enfeksiyonu ile mesaneye geçmek gibi) veya başka bir konak organizmanın bağırsağı olabilir, E. coli bir hastalık etmeni olabilir. Bazı E. coli tipleri içinde bulundukları hayvan için zararsız olmalarına rağmen insana geçtiklerinde hastalık yapabilirler. Bu hastalıklar arasında başlıca ishalli hastalıklar olmakla beraber idrar yolu enfeksiyonları, menenjit, peritonit, mastit, septisemi ve gram-negatif pnömoni de sayılabilir. E. coli 'nin, tavuk, dana ve başka hayvanlarda da hastalık yapabildiği gösterilmiştir.
E. coli içinde hastalık yapan pek çok tipi vardır. Bunlar hasta ettikleri dokular ve hastalık mekanizmalarına bağlı olarak aşağıdaki "patotip" olarak gruplandırılırlar:
İshalli hastalıklara neden olan E. coli tipleri aşağıdaki gruplara ayrılırlar:
E. coli türü içinde büyük bir çeşitlilik vardır, hatta modern tekniklerle gösterilmiştir ki Shigella ve Salmonella familyasının üyeleri aslında E. coli'nin alt-tipleridir.[4] E. coli türü içinde farklı özelliklere sahip olan, "suş" olarak adlandırılan çeşitli tipler vardır. Bunları birbirinden farklı kılan küçük mutasyonlar olabileceği gibi bütün bir genin, hatta pek çok genin, varlığı veya yokluğu, olabilir. Bu genler bakteriofaj, transpozon veya plazmidlerde bulunur ve bunlar başka bakteri türlerinden E. coli'ye iletilmiş olur.
Suşları farklı kılan genler arasında toksin ve yapışma (adezyon) faktörleri gibi hastalık (virülans) faktörleri vardır. Örneğin O157:H7 suşunun taşıdığı Şiga toksini geni, E. coli 'ye Shigella'dan geçmiştir. Aşağıda E. coli 'nin hastalık yapmasını sağlayan özelliklerin bazıları sıralanmıştır. Bunların hepsi bir arada olmaz, belli E. coli suşları bu faktörlerin belli kombinasyonlarına sahiptir.
Gıdaların yıkanması patojen E. coli enfeksiyonun yeme yoluyla yayılmasını engellemenin en etkili yoludur. E. coli bulaşmış yiyeceklerin kaynatılması da etkilidir.
Uygun tedavi, enfeksiyonun nedeni olan E. coli tipinin antibiyotik duyarlılığına bağlıdır. E. coli enfeksiyonlarını tedavide kullanılabilecek antibiyotikler arasında amoksisilin, trimethoprim-sulfamethoxazole, ciprofloxacin, nitrofurantoin sayılabilir. E. coli 'nin neden olduğu her hastalık için her antibiyotik uygun olmayabilir, bu konuda bir doktora danışmak gereklidir.
Antibiyotiğe direnç gelişmesi büyüyen bir sorundur. Bunun başlıca nedeni insanlarda antibiyotiklerin gereksiz kullanımıdır. Geniş spektrumlu beta-laktamaz üreten E.coli suşları çeşitli antibiyotiklere dayanıklı bir beta laktamaz enzimi üretirler ve bunların tedavisi çok daha zordur. Çoğu durumda bu suşlara karşı yalnızca iki oral antibiyotik ve damardan alınan sınırlı bir grup antibiyotik etkilidir.
Bakterilerde "suş", ortak özellikleri ile başka suş'lardan ayırdedilebilen bir gruptur. Bu farklılıklar genelde moleküler düzeyde algılanabilse de bakterinin fizyolojisi ve yaşam döngüsüne etki edebilirler, örneğin patojenliğe yol açabilirler. Farklı E. coli suşları farklı hayvanlarda yaşadıkları için sudaki dışkı kirlenmesinin kaynağını anlamak mümkündür. Yeni E. coli suşları doğal mutasyonlar sonucu sürekli olarak belirmektedir ve bunların bazılarının özellikleri içinde bulundukları konak hayvana zararlı olabilir. Çoğu sağlıklı insanda böylesi yeni bir E. coli suşu bir hafif bir ishale yol açsa da, küçük çocuklarda, başka bir hastalıktan dolayı zayıf düşmüşlerde veya bazı ilaçları alanlarda ciddi bir hastalık hatta ölüm meydana gelebilir.
E. coli suşlarını tanımlamanın bir yolu yüzeyindeki antijenler yoluyladır. "O" bakterinin yüzeyindeki, "H" de flagelladaki antijeni belirtir. Bu teknikle tanımlanan tiplere serotip denir. Bir serotip genetik anlamda homojen olmasa da genelde belli serotiplerin hastalık etkileri aynı olduğu için halk sağlığı ve tıbbi mikrobiyoloji açısından pratik bir sınıflandırma yöntemi olarak kullanılırlar. E. coli serotipleri arasında ölümcül olabilmesinden dolayı en ün yapmış olanı O157:H7'dir.
E. coli türünün içinde büyük bir çeşitlilik vardır. Bu, kısmen farklı ortamlarda yaşayan bakterilerin ufak mutasyonlar biriktirerek faklılaşmasından dolayı olsa da, çeşitliliğin büyük bir bölümü bazı genlerin başka bakterilerden ediniminden meydana gelir. Çeşitli patojen E. coli türlerinin farklı suşlardan kaynaklandıkları, bunların birbirlerinden bağımsız olarak virülans genlerini "yatay transfer" yoluyla elde ettikleri gösterilmiştir. Genomunda "genetik ada" olarak adlandırılan bölgelerde dışarıdan alınma genler kümelenir. Shigella türlerinin patolojik E. coli türlerinden evrimleştiği düşünülmektedir.
Yaygın bir bakteri olmasından dolayı E. coli mikrobiyolojide sıkça çalışılmıştır ve moleküler biyolojide bir gereç haline gelmiştir. Yapısı bellidir, hayat bilimlerini çalışan her seviyede ögrenci ve araştırmacı için ideal bir araştırma organizmasıdır.
Bakteriyel konjügasyon, genetik rekombinasyon, operon kavramları ilk E. coli 'de keşfedilmiştir, DNA'nın çoğalması, RNA transkripsiyonu, protein sentezi gibi, moleküler biyolojinin pek çok önemli mekanizması, metabolizmanın çoğu ayrıntısı bu organizmada yapılan araştırmalarla anlaşılmıştır. En az on Nobel Ödülü E. coli 'de yapılan araştırmalara dayanır.
Laboratuvarda kullanılan standart E. coli suşunun adı K12'dir. E. coli K12'nin ve O157:H7 serotipli bir suşun genom dizinleri çözülmüştür. K12 genomu yaklaşık 4200 genden oluşmaktadır, O157:H7 genomu ise K12'ninkiden %25 daha büyüktür. K12 suşu hastalık yapan bir faktörler taşımaz ve hatta K12'nin ilk izolasyonundan günümüze geçen yıllar zarfında kapsül yapma yeteneğini kaybederek laboratuvar ortamına uyum sağlamış, artık doğal ortamında (yani insan bağırsağında) başka E. coli türleriyle rekabet edemeyecek kadar zayıflamıştır.
E. coli modern biyoloji mühendisliğinde önemli bir yeri vardır. Araştırmacılar bu bakteriyi büyük miktarda DNA veya protein üretmek amacıyla bir fabrika gibi kullanırlar. Rekombinant DNA teknolojisinin ilk faydalı uygulamalarından biri E. coli 'nin manipüle edilerek onun diyabetli hastalar için insülin üretmesini sağlamak olmuştur.
Genelde E. coli kısaltması ile veya koli basili olarak bilinen Escherichia coli (okunuşu Eşerihiya koli), memeli hayvanların kalın bağırsağında yaşayan bakteri türlerinden biridir. Normalde bağırsakta yaşadığı için, E. coli 'nin çevresel sularda varlığı dışkı kirlenmesinin bir belirtisidir.
E. coli, pediyatrist ve bakteriyolog olan Theodor Escherich tarafından bebek dışkılarında keşfedilmiştir ve adını ondan alır; coli, "kalın bağırsaktan" demektir. E. coli, genel olarak bakteri biyolojisinin anlaşılması amacıyla üzerinde sıkça çalışılmış bir model organizma olmuştur. Canlılar arasında hakkında en fazla şey bilinen organizma olduğu söylenebilir.
İnsanın bir günde dışkı yoluyla vücudundan geçen E. coli bakteri sayısı 100 milyar ila 10 trilyon arasındadır. Dışkıyı oluşturan bakteriler başlıca aerobik bakterilerdir, seçmeli anerobik E. coli hücrelerinin sayısı diğer bakteri türlerinin binde biri dolayındadır. Başka hayvanlarda etkisiz olan bazı E. coli tipleri insana bulaştıklarında hastalık yapabilirler. Bunların en ünlüsü sayılan O157:H7 adlı serotip kanlı ishale ve ölüme yol açabilir.
E. coli, normal bağırsak florasına aittir, biyolojik sınıflandırmada da bağırsaklarda yaşayan bakterilerden oluşan enterik bakteriler ailesinde yer alır. Bakteri çubuk şeklinde olup, boyutları 1-2 µm uzunluğunda ve 0.1-0.5 µm çapındadır.
E. coli Gram-negatif bir bakteri olduğundan endospor oluşturmaz, pastörizasyon veya kaynatma ile ölür. Memeli hayvanların bağırsaklarında büyümeye adapte olmuş olduğu için en iyi vücut sıcaklığında çoğalır.
Кишкова паличка (Escherichia coli, звичайно скорочується до E. coli, від лат. coli — «кишкова») — відкрита німецьким педіатром і мікробіологом Теодором Ешеріхом (нім. Theodor Escherich) — один з головних видів бактерій, що живуть у нижніх відділах кишечнику ссавців, загалом відомих як флора кишечника. Деякі зразки також були знайдені на краю гарячих джерел. Кілька сотень штамів бактерій E. coli можуть спричинити хворобу ешерихіоз у людей[1]. Присутність цих видів на поверхні водойм — загальний індикатор забруднення води фекаліями. Назва родини, до якої належить E. coli (Enterobacteriaceae) посилається на кишечник і часто також на фекалії. E. coli — найпоширеніша бактерія в лабораторних дослідженнях і зазвичай використовується як модельний організм для вивчення всіх бактерій взагалі.
Число індивідуальних бактерій E. coli в фекаліях, що одна людина виділяє за день, може бути від 100 млрд до 10 трлн. Всі різні види фекальних (coli) бактерій, і всі пов'язані з ними бактерії, що живуть у ґрунті та гниючих рослинах, наприклад, Aerogenes enterobacter, групуються разом під назвою колідоподібних (coliform). Всі представники колідоподібних бактерій аеробні або факультативно анаеробні, не формуючі спор, грам-негативні, паличкоподібні бактерії, що ферментують лактозу з виділенням газу в межах 48 годин при температурі 35 °C. В організмі цей газ створює метеоризм. Клітини E. coli паличкоподібні, 1-2 µм завдовжки і 0.1-0.5 µм в діаметрі.
Як більшість грам-негативних бактерій, E. coli не в змозі формувати спори. Тому, обробка, яка вбиває всі активні бактерії, наприклад пастеризація або кип'ятіння, ефективні для знищення цих бактерій, без необхідності суворіших методів стерилізації, які застосовують аби вбити спори. Внаслідок адаптації до кишечників ссавців, E. coli краще всього ростуть при температурах притаманних температури тіла цих тварин, а не температурах ґрунтів та інших природних середовищ.
Штам — це сукупність особин всередині виду, яка має властивості, відмінні від властивостей інших особин. Часто такі відмінності можуть бути виявлені тільки на молекулярному рівні, проте, мають ефект на фізіологію бактерії або життєвий цикл. Різні штами E. coli часто специфічні до певних господарів, що робить можливим визначення джерела фекального зараження в зразках. Наприклад, якщо відомо, які штами E. coli представлені в зразку води, можна визначити джерело зараження, наприклад, людина, інший ссавець чи птах.
Нові штами E. coli з'являються в результаті мутацій та горизонтального переносу генів.[2] Вірулентніші штами, наприклад, O157:H7 спричиняють тяжкі форми ешерихіозу і навіть призводять до смерті літніх людей, маленьких дітей та осіб з ослабленим імунітетом.[3][4]
Кишкова паличка (Escherichia coli, звичайно скорочується до E. coli, від лат. coli — «кишкова») — відкрита німецьким педіатром і мікробіологом Теодором Ешеріхом (нім. Theodor Escherich) — один з головних видів бактерій, що живуть у нижніх відділах кишечнику ссавців, загалом відомих як флора кишечника. Деякі зразки також були знайдені на краю гарячих джерел. Кілька сотень штамів бактерій E. coli можуть спричинити хворобу ешерихіоз у людей. Присутність цих видів на поверхні водойм — загальний індикатор забруднення води фекаліями. Назва родини, до якої належить E. coli (Enterobacteriaceae) посилається на кишечник і часто також на фекалії. E. coli — найпоширеніша бактерія в лабораторних дослідженнях і зазвичай використовується як модельний організм для вивчення всіх бактерій взагалі.
Число індивідуальних бактерій E. coli в фекаліях, що одна людина виділяє за день, може бути від 100 млрд до 10 трлн. Всі різні види фекальних (coli) бактерій, і всі пов'язані з ними бактерії, що живуть у ґрунті та гниючих рослинах, наприклад, Aerogenes enterobacter, групуються разом під назвою колідоподібних (coliform). Всі представники колідоподібних бактерій аеробні або факультативно анаеробні, не формуючі спор, грам-негативні, паличкоподібні бактерії, що ферментують лактозу з виділенням газу в межах 48 годин при температурі 35 °C. В організмі цей газ створює метеоризм. Клітини E. coli паличкоподібні, 1-2 µм завдовжки і 0.1-0.5 µм в діаметрі.
Як більшість грам-негативних бактерій, E. coli не в змозі формувати спори. Тому, обробка, яка вбиває всі активні бактерії, наприклад пастеризація або кип'ятіння, ефективні для знищення цих бактерій, без необхідності суворіших методів стерилізації, які застосовують аби вбити спори. Внаслідок адаптації до кишечників ссавців, E. coli краще всього ростуть при температурах притаманних температури тіла цих тварин, а не температурах ґрунтів та інших природних середовищ.
Escherichia coli (thường ghi tắt: E. coli) là một loài vi khuẩn Gram âm, phân bố rất rộng trong môi trường sống trên Trái Đất, hay có mặt ở thực phẩm, nguồn nước, thường kí sinh trong ruột già của người và hầu hết các loài Thú đẳng nhiệt.[1][2][3] Đa số các chủng E. coli là vô hại mặc dù kí sinh, chỉ một số dòng có thể gây ngộ độc thức ăn, gây bệnh đường ruột.[4][5] Trong những trường hợp nhất định, chúng còn giúp vật chủ nhờ sản xuất vitamin K2,[6] và chống sự xâm lấn của một vài mầm bệnh khác, tạo nên một mối quan hệ cộng sinh.[7][8]
E. coli thường được nhắc đến chủ yếu vì nó là loài sinh vật mô hình rất quan trọng trong Sinh học hiện đại, đặc biệt trong Di truyền học phân tử. Ngoài ra, sự có mặt của chúng trong nguồn nước là một chỉ tiêu quan trọng để đo độ sạch của nước, do E. coli bị thải ra môi trường qua phân, có khả năng tiếp tục tạo nên các quần thể sống tự do, sinh trưởng mạnh trong phân tươi ở điều kiện yếm khí vài ba ngày rồi mới giảm tăng trưởng.[9][10]
2015: Giải Nobel Hóa học năm 2015 được trao cho Tomas Lindahl, Paul Modrich và Aziz Sancar “cho các nghiên cứu cơ học về sửa chữa DNA” khi nghiên cứu về Escherichia coli.
2008: Prôtêin huỳnh quang xanh dùng đánh dấu theo dõi các thành phần của tế bào vi khuẩn.
1999: Các chuỗi tín hiệu trên prôtêin biểu hiện phương thức tế bào tự tổ chức.
1997: Phương thức tế bào tạo ra ATP - năng lượng của sự sống.
1989: Vai trò bổ sung của ARN.
1980: Tái tổ hợp ADN.
1978: Các enzym giới hạn có vai trò như chiếc “kéo” cho phép các nhà khoa học cắt ADN.
1969: Sự nhân lên của virus trong tế bào vật chủ.
1968: Hoàn thiện về mã di truyền, "ngôn ngữ" của ADN.
1965: Điều hoà gen: gen được "bật" hoặc "tắt" như thế nào.
1959: Cơ chế nhân đôi ADN, nhờ đó sự sống được chuyển giao cho đời sau.
1958: Giới tính ở vi khuẩn và phương thức vi khuẩn chia sẻ gen với nhau.
Những nghiên cứu về loài vi khuẩn này nhiều đến mức không chỉ dẫn đến các thành tựu nổi bật đã liệt kê ở trên, mà còn tạo ra một bộ môn được coi như là một nhánh quan trọng của ngành Sinh học gọi là Sinh học E. coli (Escherichia coli Biology), bao gồm các kiến thức từ mô tả hình thái, sinh lý theo kiểu cổ điển cho đến thành phần sinh hoá, hoạt động của bộ gen ở cấp độ phân tử trong Di truyền học phân tử và Sinh hoá học của loài này.
Escherichia coli (thường ghi tắt: E. coli) là một loài vi khuẩn Gram âm, phân bố rất rộng trong môi trường sống trên Trái Đất, hay có mặt ở thực phẩm, nguồn nước, thường kí sinh trong ruột già của người và hầu hết các loài Thú đẳng nhiệt. Đa số các chủng E. coli là vô hại mặc dù kí sinh, chỉ một số dòng có thể gây ngộ độc thức ăn, gây bệnh đường ruột. Trong những trường hợp nhất định, chúng còn giúp vật chủ nhờ sản xuất vitamin K2, và chống sự xâm lấn của một vài mầm bệnh khác, tạo nên một mối quan hệ cộng sinh.
E. coli thường được nhắc đến chủ yếu vì nó là loài sinh vật mô hình rất quan trọng trong Sinh học hiện đại, đặc biệt trong Di truyền học phân tử. Ngoài ra, sự có mặt của chúng trong nguồn nước là một chỉ tiêu quan trọng để đo độ sạch của nước, do E. coli bị thải ra môi trường qua phân, có khả năng tiếp tục tạo nên các quần thể sống tự do, sinh trưởng mạnh trong phân tươi ở điều kiện yếm khí vài ba ngày rồi mới giảm tăng trưởng.
Штамм — это совокупность особей внутри вида, которая обладает свойствами, отличными от свойств других особей. Часто такие отличия могут быть обнаружены только на молекулярном уровне, однако имеют эффект на физиологию бактерии или жизненный цикл. Разные штаммы E. coli часто специфичны к определённым хозяевам, что делает возможным определение источника фекального заражения в образцах[6][7]. Например, если известно, какие штаммы E. coli представлены в образце воды, можно определить источник заражения, например, человек, другое млекопитающее или птица.
Новые штаммы E. coli появляются в результате мутаций и горизонтального переноса генов[9]. Некоторые штаммы вырабатывают особенности, губительные для организмов хозяина, такие вирулентные штаммы могут вызывать диарею, что неприятно в случае взрослых и может привести к летальному исходу у детей в развивающихся странах[10]. Более вирулентные штаммы, например, O157:H7 вызывают тяжёлые заболевания и даже приводят к смерти у пожилых людей, маленьких детей и лиц с ослабленным иммунитетом[10][11].
E. coli — грамотрицательная бактерия, факультативный анаэроб, не образует эндоспор. Клетки палочковидные, со слегка закруглёнными концами, размером 0,4—0,8 × 1—3 мкм, объём клетки составляет около 0,6—0,7 мкм³[12][13]. Кишечная палочка может жить на разных субстратах. В анаэробных условиях E. coli образует в качестве продукта жизнедеятельности лактат, сукцинат, этанол, ацетат и углекислый газ. Часто при этом образуется молекулярный водород, который мешает образованию указанных выше метаболитов, поэтому E. coli часто сосуществует с микроорганизмами, потребляющими водород — например, с метаногенами или бактериями, восстанавливающими сульфат[14].
Оптимальный рост достигается культурами E. coli при температуре 37°C, некоторые штаммы могут делиться при температурах до 49°C[15]. Рост может стимулироваться аэробным или анаэробным дыханием, различными парами окислителей и восстановителей, в том числе, окислением пирувата, формиата, водорода, аминокислот, а также восстановлением кислорода, нитрата, диметилсульфоксида и триметиламин N-оксида[16].
Штаммы, имеющие жгутики, способны передвигаться. Жгутики расположены перитрихиально[17]. На конце жгутика расположен белок FimH, который прикрепляется к молекулам сахаров на поверхности, а сам жгутик состоит из цепочки взаимосвязанных белковых сегментов, закрученных в форме тонкой длинной пружины и упруго вытягивающихся при воздействии силы[18][19].
E. coli в норме заселяет кишечник новорождённого ребёнка в течение 40 часов после рождения, поступая с пищей или от лиц, контактирующих с ребёнком, и сохраняются на протяжении жизни на уровне 106—108 КОЕ/г содержимого толстой кишки. В ЖКТ кишечные палочки прилипают к слизистым оболочкам и являются основными представителями факультативных анаэробов у человека. Так как кишечные палочки не имеют бактериофагов, кодирующих факторы вирулетности, они являются комменсалами[20]. По другим данным, микроорганизмы (в том числе E. coli) начинают заселять человеческий организм ещё в утробе матери[21].
Непатогенный штамм Escherichia coli Nissle 1917 известен как Mutaflor и используется в медицине в качестве пробиотика, в основном для лечения желудочно-кишечных заболеваний, в том числе у новорождённых[22][23].
E. coli часто используют в качестве модельного организма в микробиологических исследованиях. Культивируемые штаммы, например, E. coli K12 хорошо приспособлены к росту в лабораторных условиях, и, в отличие от штаммов дикого типа, неспособны заселять кишечник. Многие лабораторные штаммы утеряли способность образовывать биологические плёнки[24][25]. Описанные особенности предохраняют штаммы дикого типа от антител и химических агентов, но требуют больших затрат вещества и энергии.
В 1946 году Джошуа Ледерберг и Эдуард Тейтем описали явление конъюгации бактерий, используя кишечную палочку в качестве модельного организма[26]. E. coli остаётся одной из наиболее востребованных бактерий при изучении конъюгации и в настоящее время. E. coli была важным компонентом первых экспериментов по генетике бактериофагов[27], ранние исследователи, например, Сеймор Бензер, использовали E. coli и фаг T4 для изучения структуры генов[28]. До исследований Бензера не было известно, имеет ген линейную или разветвлённую структуру.
Кишечная палочка E. coli была одним из первых организмов, чей геном был полностью секвенирован. Последовательность нуклеотидов в геноме штамма К12 E. coli была опубликована в журнале Science в 1997 году[29].
Долговременный эксперимент по эволюции E. coli был начат Ричардом Ленски в 1988 году и позволил непосредственно наблюдать эволюционные изменения в лабораторных условиях[30]. В данном эксперименте одна популяция E. coli получила возможность аэробно метаболизировать цитрат. Такая способность встречается у E. coli в норме крайне редко. Неспособность к росту в аэробных условиях используют для того, чтобы отличить E. coli от других родственных бактерий, например, Salmonella. В ходе данного эксперимента в лабораторных условиях удалось наблюдать процесс видообразования.
E. coli играет важную роль в современной промышленной микробиологии и биологической инженерии[31]. Работа Стенли Нормана Коэна и Герберта Бойера на E. coli с использованием плазмид и эндонуклеаз рестрикции для создания рекомбинантной ДНК находится у истоков современной биотехнологии[32].
Кишечную палочку считают универсальным организмом для синтеза чужеродных белков[33]. В E. coli исследователи вводят гены при помощи плазмид, что позволяет осуществлять биосинтез белков для промышленной ферментации. Также разработаны системы для синтеза в E. coli рекомбинантных белков. Одним из первых примеров использования технологии рекомбинантных ДНК является синтез аналога инсулина человека[34]. Модифицированные E. coli используют при разработке вакцин, синтеза иммобилизованных ферментов и решения других задач[33]. Однако в организме E. coli невозможно получать некоторые крупные белковые комплексы, содержащие дисульфидные связи, в частности, белки, для проявления биологической активности которых требуется посттрансляционная модификация[31].
Гены кишечной палочки также используются для генетической модификации растений, в частности из нее выделяют ген устойчивости к антибиотикам неомицину и канамицину.[35]
Непатогенные бактерии E. coli, в норме в больших количествах населяющие кишечник, могут, тем не менее, вызвать развитие патологии при попадании в другие органы или полости человеческого тела. Если бактерия попадает через отверстие в ЖКТ в брюшную полость, может возникнуть перитонит. Попав и размножившись во влагалище женщины, бактерия может вызвать или осложнить кольпит. Попадание бактерии в предстательную железу мужчины может быть патогенезом острого или хронического бактериального простатита. В таких случаях в лечение включается применение антибиотиков, проводимое таким образом, чтобы не подавлять нормальную микрофлору кишечника, иначе возможно развитие дисбактериоза.
E. coli очень чувствительна к таким антибиотикам, как стрептомицин или гентамицин. Однако E. coli может быстро приобретать лекарственную устойчивость[36].
Вирулентные штаммы E. coli в норме отсутствуют в кишечнике, и заболевание наступает при заражении алиментарным путём. Передача патогенных E. coli часто происходит фекально-оральным путём[20][37][38]. Частые пути передачи могут быть вызваны: низкой гигиеной приготовления пищи[37], загрязнением продуктов навозом[39], поливом урожая загрязнённой водой или сточными водами[40], при выпасе диких свиней на пашнях[41], употреблением для питья воды, загрязнённой сточными водами[42].
Вирулентные штаммы E. coli могут вызывать гастроэнтериты, воспаления мочеполовой системы, а также менингит у новорождённых. В редких случаях вирулентные штаммы также вызывают гемолитический-уремический синдром, перитонит, мастит, сепсис и грамотрицательную пневмонию.
Некоторые штаммы E. coli, например, O157:H7, O121, O104:H4 и O104:H21, синтезируют потенциально смертельные токсины. Пищевые отравления, инфекционным агентом при которых являются вирулентные E. coli, обычно вызваны употреблением в пищу немытых овощей или непрожаренного мяса.
Первичными резервуарами E. coli O157:H7 является мясной и молочный скот[43], который может переносить бактерии бессимптомно и выделять с фекалиями[43].
В случае заболеваний кишечника у новорождённых, при болезни Крона и при неспецифическом язвенном колите обнаруживают повышенные уровни E. coli в слизистых ЖКТ[44]. Инвазивные штаммы E. coli обнаружены в воспалённых тканях, а количество бактерий в очагах воспаления коррелирует с тяжестью воспаления в кишечнике[45].
Один из серотипов Escherichia coli содержит антиген K1. Заселение кишечника новорождённого данным серотипом бактерий при попадании бактерий из влагалища матери может приводить к менингиту. В отсутствие IgM от матери, которые не способны проникать через гемато-плацентарный барьер, и потому, что организм распознаёт K1 как собственный антиген, данный серотип вызывает тяжёлые воспаления мозга.
Терапия бактериофагами для лечения патогенных бактерий была разработана более 80 лет назад в Советском Союзе, где использовалась для лечения диареи, вызванной E. coli[46]. В настоящее время фаговая терапия доступна лишь в Центре фаговой терапии в Грузии и в Польше[47].
Бактериофаг Т4 является хорошо изученным фагом, инфицирующим E. coli.
Исследователи разрабатывают эффективные вакцины для снижения количества случаев заражения патогенными штаммами E. coli по всему миру[48].
В апреле 2009 года исследователи Мичиганского университета заявили о том, что разработали вакцину для одного из штаммов E. coli. Подана заявка на патент[49].
|month= (справка) |month= (справка) |month= (справка) |month= (справка) |month= (справка) |month= (справка) |month= (справка); Проверьте дату в |month= (справка на английском) Source: National Library of Medicine — The Joshua Lederberg Papers |month= (справка) |pmc= (справка на английском). Используется устаревший параметр |month= (справка); Проверьте дату в |month= (справка на английском) |month= (справка) |month= (справка) |month= (справка) |coauthors= (справка) |coauthors= (справка) (недоступная ссылка) Штамм — это совокупность особей внутри вида, которая обладает свойствами, отличными от свойств других особей. Часто такие отличия могут быть обнаружены только на молекулярном уровне, однако имеют эффект на физиологию бактерии или жизненный цикл. Разные штаммы E. coli часто специфичны к определённым хозяевам, что делает возможным определение источника фекального заражения в образцах. Например, если известно, какие штаммы E. coli представлены в образце воды, можно определить источник заражения, например, человек, другое млекопитающее или птица.
Новые штаммы E. coli появляются в результате мутаций и горизонтального переноса генов. Некоторые штаммы вырабатывают особенности, губительные для организмов хозяина, такие вирулентные штаммы могут вызывать диарею, что неприятно в случае взрослых и может привести к летальному исходу у детей в развивающихся странах. Более вирулентные штаммы, например, O157:H7 вызывают тяжёлые заболевания и даже приводят к смерти у пожилых людей, маленьких детей и лиц с ослабленным иммунитетом.
大腸桿菌(學名:Escherichia coli,通常簡寫:E. coli)是人和动物肠道中著名的一种细菌,主要寄生于大肠内,約占腸道菌中的0.1%。大腸桿菌是一种两端钝圆、能运动、无芽孢的革蘭氏陰性短杆菌。大部分的大腸桿菌不會致病,但有些血清型會造成嚴重的食物中毒或食物污染。無害的菌株是人體腸道中正常菌叢的一部份,會製造維他命K、防止腸道中其他致病菌的生長,對人體有益。大腸桿菌經常透過糞便排放而散佈到環境中,它們會在新鮮的糞便且氧氣充足的環境中大量孳生約3天,之後菌數就會下降。
其屬名埃希氏菌(Escherichia)來源於其發現者特奥多尔·埃舍里希。1885年當埃舍里希嘗試找出霍亂病原時,他分離出大腸桿菌,并将其最初命名為Bacterium coli commune。
大腸桿菌和其他兼性厭氧性細菌組成了0.1%的腸道微生物,而糞口傳染是致病菌株的主要傳染途徑。它可以在人體外存活一段時間,因此成為環境衛生檢測中的微生物指標之一。大腸桿菌是腸桿菌科的一員,經常作爲細菌的模式生物廣泛用於科學研究。
每個人每天平均從糞便中排出1011到1013個大腸桿菌。各種糞便細菌和類似的生活在土壤或植物降解物中的細菌(最常見的是產氣腸桿菌,學名Enterobacter aerogenes)一起被歸爲“大腸桿菌群”(coliform)。大腸桿菌群不形成內孢子,可在膽鹽存在下生長,革蘭氏陽性菌則否,該菌群可以利用乳糖並產氣,這個特性可以用來當概略性菌群的判定基礎,因為大腸桿菌群容易培養,而且容易跟其他菌分辨,所以是理想的指標菌,除非有特別的異形菌存在增加鑑定的困難度外,異常的乳糖發酵代表衛生可能有問題[1]。
在水淨化和污水處理領域,因大腸桿菌在糞便中数量极多,故常用为检查水源是否被粪便污染的标志,其測量標準爲大腸菌群指數。此外大腸桿菌多數情況下無害,不會從實驗室“逃脫”而傷害人類。利用大腸桿菌作爲糞便污染的指示物也可能產生誤導性的結論,因爲其它環境如造紙廠中,大腸桿菌也可大量存在。
然而一般無害的大腸桿菌在以下三种情況下也會導致疾病:
大腸桿菌的一個株是具有某些能和其它株區分開的特徵的族群。不同的大腸桿菌菌株生活在不同動物中,因此我們可以通過其判斷糞便來源於人或者鳥類等。通過突變,新的大腸桿菌菌株不斷出現,其中一些可能對宿主動物造成損害。儘管對於大多數健康成年人,這樣的菌株可能只引起一場腹瀉,或者根本沒有症狀,但對於幼兒、大病初癒的人或者進行某些藥物治療的人來説,陌生的菌株可能引起嚴重疾病甚至死亡。大腸桿菌O157:H7就是一個毒性很強的菌株。
大腸桿菌是現代生物學中研究最多的一種細菌,作爲一種模式生物,其基因組序列已全部測出。用分子生物學方法在大腸桿菌得出的結論可用於其它生物的研究。此外,在生物工程中,大腸桿菌被廣泛用於基因複製和表達的宿主。
大腸桿菌(學名:Escherichia coli,通常簡寫:E. coli)是人和动物肠道中著名的一种细菌,主要寄生于大肠内,約占腸道菌中的0.1%。大腸桿菌是一种两端钝圆、能运动、无芽孢的革蘭氏陰性短杆菌。大部分的大腸桿菌不會致病,但有些血清型會造成嚴重的食物中毒或食物污染。無害的菌株是人體腸道中正常菌叢的一部份,會製造維他命K、防止腸道中其他致病菌的生長,對人體有益。大腸桿菌經常透過糞便排放而散佈到環境中,它們會在新鮮的糞便且氧氣充足的環境中大量孳生約3天,之後菌數就會下降。
其屬名埃希氏菌(Escherichia)來源於其發現者特奥多尔·埃舍里希。1885年當埃舍里希嘗試找出霍亂病原時,他分離出大腸桿菌,并将其最初命名為Bacterium coli commune。
大腸桿菌和其他兼性厭氧性細菌組成了0.1%的腸道微生物,而糞口傳染是致病菌株的主要傳染途徑。它可以在人體外存活一段時間,因此成為環境衛生檢測中的微生物指標之一。大腸桿菌是腸桿菌科的一員,經常作爲細菌的模式生物廣泛用於科學研究。
每個人每天平均從糞便中排出1011到1013個大腸桿菌。各種糞便細菌和類似的生活在土壤或植物降解物中的細菌(最常見的是產氣腸桿菌,學名Enterobacter aerogenes)一起被歸爲“大腸桿菌群”(coliform)。大腸桿菌群不形成內孢子,可在膽鹽存在下生長,革蘭氏陽性菌則否,該菌群可以利用乳糖並產氣,這個特性可以用來當概略性菌群的判定基礎,因為大腸桿菌群容易培養,而且容易跟其他菌分辨,所以是理想的指標菌,除非有特別的異形菌存在增加鑑定的困難度外,異常的乳糖發酵代表衛生可能有問題。
在水淨化和污水處理領域,因大腸桿菌在糞便中数量极多,故常用为检查水源是否被粪便污染的标志,其測量標準爲大腸菌群指數。此外大腸桿菌多數情況下無害,不會從實驗室“逃脫”而傷害人類。利用大腸桿菌作爲糞便污染的指示物也可能產生誤導性的結論,因爲其它環境如造紙廠中,大腸桿菌也可大量存在。
然而一般無害的大腸桿菌在以下三种情況下也會導致疾病:
當細菌離開腸道進入泌尿道可以導致感染,由於性交會導致細菌進入膀胱,有時被稱作“蜜月膀胱炎”。尿路感染儘管對女性更爲普遍,但兩性都可能發生。老年中發病男女比例大體相同。因爲細菌總是通過尿道進入泌尿系統,廁所的不衛生會升高感染機率,但其它因素也很重要(如女性懷孕,男性前列腺肥大),還有一些原因不明。 當細菌由於如潰瘍等導致的穿孔進入腹腔,通常會導致致命性的腹膜炎感染。然而,大腸桿菌對一些抗生素,如鏈黴素非常敏感,一般情況抗生素能夠有效治療。 大腸桿菌的某些株具有毒性(其中一些類似導致痢疾的毒素),可以導致食物中毒,這通常是因爲使用了被污染的肉類(通常是屠宰過程或儲藏販賣過程中的污染所致,加上食物未完全煮熟無法殺死細菌)。疾病的嚴重程度可以相差很多,尤其對兒童、老人和免疫缺失病人可以是致命的,但通常是溫和的。大腸桿菌的内毒素可能對熱穩定或不穩定。後者的結構和功能與霍亂毒素相當接近,全毒素包含一個A亞基和五個B亞基。B亞基起黏附作用,使毒素進入腸道細胞,而A亞基斷裂出來,使得細胞脫水引起腹瀉。大腸桿菌的一個株是具有某些能和其它株區分開的特徵的族群。不同的大腸桿菌菌株生活在不同動物中,因此我們可以通過其判斷糞便來源於人或者鳥類等。通過突變,新的大腸桿菌菌株不斷出現,其中一些可能對宿主動物造成損害。儘管對於大多數健康成年人,這樣的菌株可能只引起一場腹瀉,或者根本沒有症狀,但對於幼兒、大病初癒的人或者進行某些藥物治療的人來説,陌生的菌株可能引起嚴重疾病甚至死亡。大腸桿菌O157:H7就是一個毒性很強的菌株。
大腸桿菌是現代生物學中研究最多的一種細菌,作爲一種模式生物,其基因組序列已全部測出。用分子生物學方法在大腸桿菌得出的結論可用於其它生物的研究。此外,在生物工程中,大腸桿菌被廣泛用於基因複製和表達的宿主。

大腸菌(だいちょうきん、学名: Escherichia coli)は、グラム陰性の桿菌で通性嫌気性菌に属し、環境中に存在するバクテリアの主要な種の一つである。この菌は腸内細菌でもあり、温血動物(鳥類、哺乳類)の消化管内、特にヒトなどの場合大腸に生息する。アルファベットで短縮表記でE. coliとすることがある(詳しくは#学名を参照のこと)。大きさは通常短軸0.4-0.7μm、長軸2.0-4.0μmだが、長軸が短くなり球形に近いものもいる[1]。
バクテリアの代表としてモデル生物の一つとなっており、各種の研究で材料とされるほか、遺伝子を組み込んで化学物質の生産にも利用される(下図)。

 大腸菌のコロニー グラム染色像
大腸菌のコロニー グラム染色像 大腸菌はそれぞれの特徴によって「株」と呼ばれる群に分類することができる(動物でいう品種のような分類)。それぞれ異なる動物の腸内にはそれぞれの株の 大腸菌が生息していることから、環境水を汚染している糞便が人間から出たものか、鳥類から出たものかを判別することも可能である。大腸菌には非常に多数の株があり、その中には病原性を持つものも存在する。
大腸菌は、菌の表面にある抗原(O抗原とH抗原)に基づいて細かく分類されている[2]。O抗原は外膜のリポ多糖由来のもので、H抗原はべん毛由来のものである。O抗原は現在約180種類ほどに分類されている[2]。例えば「O157(オーいちごーなな)」という名称は、O抗原としては157番目に発見されたものを持つ菌ということを意味しており[2]、「O111(オーいちいちいち)」はO抗原としては111番目に発見されたものを持つ、ということを意味する。 H抗原は約70種類に分類されている。 なお、さらに細かく分けるとO抗原とH抗原の両方を考慮した分類になる。例えばO157でも、H抗原に関する違いでさらに細かく分類することができ、H7のものとH抗原を持たないものがあるので、「O157:H7」と「O157:H-」という2種類に分けることができる[2]。
ほとんどの 大腸菌は無害だが、いくつかの場合では疾患の原因となることがある。ヒトの場合、大腸内ではなく、血液中や尿路系に侵入した場合(異所感染した場合)に病原体となる。内毒素(リポ多糖)を産生するため、大腸菌による敗血症は重篤な内毒素ショック(エンドトキシンショック)を引き起こす。敗血症の原因(明らかになる場合)として最も多いのは尿路感染症であるが、大腸菌は尿路感染症の原因菌として最も多いものである。
大腸菌の株は多数報告されており、一部では動物に害となりうる性質を持つ株も存在する。大部分の健康な成人の持っている株では下痢を起こす程度で何の症状も示さないものがほとんどであるが、幼児や病気などによって衰弱している者、あるいはある種の薬物を服用している者などでは、特殊な株が病気を引き起こすことがあり、時として死亡に至ることもある。
大腸菌の株の中でも特に強い病原性を示すものは病原性大腸菌とよばれる。食品衛生学分野では病原大腸菌ともよぶ。ただし、病原性大腸菌の中でも赤痢を起こす株については特に赤痢菌とよび、衛生管理上の問題から別種扱いされる。
O111やO157などの腸管出血性大腸菌は牛の腸内に生息しているとされ、保健所は「内臓と他部位の肉は調理器具を使い分けるのが好ましい」としている。
学名(ラテン語名)は Escherichia coli で、属名は発見者のオーストリア人医学者テオドール・エシェリヒ Theodor Escherich にちなみ、これに屈折語尾を加えてラテン語化したもの。種形容語はラテン語で大腸を意味する「colon」の属格「coli」である。学名の正式な読みというものは存在しないが、語源を重視するとエシェリヒア・コリー、語源を無視して属名もラテン語読みするとエスケリキア・コリーとなる。英語ではエシェリキア・コーライと読む。全体として「大腸のエシェリヒ菌」の意を表す。
属名を省略してE. coli(イー・コライ、イー・コリー)と略す表記もある。ただし正式には、これは Escherichia 属が既出の場合に認められる略記である。最初からE. coli と略すのは、文脈から Escherichia 属のことを言っているのが明らかでも、不適切である。
大腸菌属は腸内細菌科のタイプ属として指定されているが、腸内細菌科の学名はEscherichiaceaeではなく、Enterobacteriaceaeとなっている。
ヒトに対して、大腸菌の死骸を含んだ液体(大腸菌死菌浮遊液)が、直腸部に塗布されると、白血球が呼び寄せられるため、感染防御の役に立つことが知られており、これを利用した薬剤が実用化されている[3]。また、遺伝子組み換え技術を用いて、大腸菌にヒト型インスリンを作らせる遺伝子を導入して、インスリンを生産することに利用されている。他にも、顆粒球コロニー刺激因子(G-CSF)や組織プラスミノーゲン活性化因子(t-PA)などの生産も、同様の方法で行われている。大腸菌に感光性を与えて撮像素子として利用できる研究も実施される。[4]
腸内に生息する菌であることから、この菌の存在は糞便による水の汚染を示唆し、河川、湖、海水浴場などの環境水の汚れの程度の指標として用いられる。
ヒト成人が一日に排泄する糞便中に含まれる菌体数は、平均で1011から1013個である。ただしヒトの消化管において、大腸菌が全体の微生物に占める割合は極めて少なく、ヒト腸内常在細菌の0.01%以下にすぎない(残りの大部分は、バクテロイデス Bacteroides 属やユーバクテリウム Eubacterium 属などの偏性嫌気性菌である)
水の浄化や汚水処理技術の分野では、培養可能な E. coli の量は人間の糞便の混入の程度を示唆するものとして、水の汚染レベルの指標としてかなり早い時期から用いられてきた。研究に使われている E. coli それ自体は無害であり、E. coli がこれらの指標に用いられるのは、他の病原性のある菌(サルモネラなど)よりもこれらの糞便由来の大腸菌の方が遥かに多く含まれるとされるためである。また、日本の水道法により上水道の浄水からは「検出されてはならない」とされている。
大腸菌群とは細菌学用語ではなく衛生上の用語である。ラクトース発酵(乳糖分解し、酸とガスを発生)するグラム陰性、好気性・通性嫌気性で芽胞を形成しない桿菌の全てである。E. coliであってもこれに該当しないものが多く存在する。
その多くは汚水菌(クレブジエラ属菌、サイトロバクター属菌、エンテロバクター属菌)や土壌中の非常によく似た性質のバクテリア(よく知られたものとしてはAerobacter aerogenes)が大腸菌群として分類される。なお、病原性大腸菌はこの検査法での検出は非常に困難である。また、水中に含まれる大腸菌群を数値化したものを大腸菌群数といい、水質汚濁の指標に用いられる。
食品衛生法では大腸菌群陰性とは加熱済み食品の加熱ができているか、加熱後の二次汚染がないかを確認するために食品の規格に規定されている。また、食品衛生法の規格基準にある検査法(EC培地において44.5℃で増殖し、乳糖を分解してガスを産生するグラム染色陰性、無芽胞の桿菌)で検出する菌を E. coli と記述しているが E. coli であってもこれにあてはまらない菌も多く食品衛生上の行政用語である。これは検査法では大腸菌群の培養温度が異なるだけの糞便性大腸菌群とほぼ同一の内容である。
大腸菌及び大腸菌群の検査には用途に応じて多くの培地が使用される。以下に主な物を列挙する。
大腸菌はそれぞれの特徴によって「株」と呼ばれる群に分類することができる(動物でいう品種のような分類)。それぞれ異なる動物の腸内にはそれぞれの株の 大腸菌が生息していることから、環境水を汚染している糞便が人間から出たものか、鳥類から出たものかを判別することも可能である。大腸菌には非常に多数の株があり、その中には病原性を持つものも存在する。
대장균(大腸菌, Escherichia coli, E. coli)은 온혈동물의 창자(대장과 소장)에서 많이 볼 수 있는 박테리아이다. 대장균 자체는 인체에 해롭지 않다.[1] 대부분의 대장균의 변종은 해롭지 않지만, 항원형 O157:H7등은 사람의 식중독을 일으키며, 가끔 대규모의 식품 리콜의 원인이 된다.[2] 해롭지 않은 변종은 대장의 공생미생물이며 비타민 K2 등을 생산하여 이로움을 주기도 하며,[3] 창자에서 병의 원인이 되는 박테리아의 번식을 막기도 한다.[4][5]
박테리아는 쉽게 번식하고 유전적으로 비교적 단순하고 다루기가 쉬워 가장 많이 연구된 원핵생물의 표본생물이고, 바이오테크놀로지에서도 중요하게 쓰인다. 독일의 소아과 의사이면서 박테리아 연구 학자인 테오도르 에쉐리히가 1885년에 발견하였으며,[6] 장내세균과에 속하는 감마프로테오박테리아로 분류된다.[7]
대한민국에서는 먹는 물 수질 기준으로 총대장균군이 지표 중 하나로 쓰인다. 기준을 만족하기 위해서는 총대장균군이 물 100mL 중 불검출되어야 한다.[8]
대장균(大腸菌, Escherichia coli, E. coli)은 온혈동물의 창자(대장과 소장)에서 많이 볼 수 있는 박테리아이다. 대장균 자체는 인체에 해롭지 않다. 대부분의 대장균의 변종은 해롭지 않지만, 항원형 O157:H7등은 사람의 식중독을 일으키며, 가끔 대규모의 식품 리콜의 원인이 된다. 해롭지 않은 변종은 대장의 공생미생물이며 비타민 K2 등을 생산하여 이로움을 주기도 하며, 창자에서 병의 원인이 되는 박테리아의 번식을 막기도 한다.
박테리아는 쉽게 번식하고 유전적으로 비교적 단순하고 다루기가 쉬워 가장 많이 연구된 원핵생물의 표본생물이고, 바이오테크놀로지에서도 중요하게 쓰인다. 독일의 소아과 의사이면서 박테리아 연구 학자인 테오도르 에쉐리히가 1885년에 발견하였으며, 장내세균과에 속하는 감마프로테오박테리아로 분류된다.