pms
nòm ant ël fil

Orthomyxoviridae (orthos,en grec significa "recte"; myxa, significa fong[1] és una família de virus d’ARN monocatenari (-) que inclou cinc gèneres: Influenzavirus A, Influenzavirus B, Influenzavirus C, Isavirus i Thogotovirus. Un sisè gènere també ha estat descrit recentment.[2] Els primers tres gèneres cusen grips en vertebrats incloent-hi els ocells i peixos de piscicultura .[3][4][5]
Basant-se en la filogenètica:
Orthomyxoviridae Gèneres, Espècies, i Serotipus gènere Espècies (* indica espècie tipus) Serotipus o Subtipus Hostes Influenzavirus A Influenza A virus* H1N1, H1N2, H2N2, H3N1, H3N2, H3N8, H5N1, H5N2, H5N3, H5N8, H5N9, H7N1, H7N2, H7N3, H7N4, H7N7, H9N2, H10N7 Humà, porc, au, cavall Influenzavirus B Influenza B virus* Humàn, foca sense orelles Influenzavirus C Influenza C virus* Humà, porc Isavirus Infectious salmon anemia virus* Salmó atlàntic Thogotovirus Thogoto virus* paparra, mosquit, mamífers (humans inclosos) Dhori virus Batken virus, Dhori virus Quaranfil virus, Johnston Atoll virus, Lake Chad virusOrthomyxoviridae (orthos,en grec significa "recte"; myxa, significa fong és una família de virus d’ARN monocatenari (-) que inclou cinc gèneres: Influenzavirus A, Influenzavirus B, Influenzavirus C, Isavirus i Thogotovirus. Un sisè gènere també ha estat descrit recentment. Els primers tres gèneres cusen grips en vertebrats incloent-hi els ocells i peixos de piscicultura .
Influenzavirus A caua de tots els grips pandèmics que infecten humans altres mamífers i aus Influenzavirus B infects humans i pinnípedes Influenzavirus C infecten humans i porcsOrthomyxoviridae (z řeckého orthos pro hladký a myxa pro sliz) je čeleď virů V.skupiny, která společně s čeleděmi Ophioviridae, Bunyaviridae a Arenaviridae v současnosti není zařazena do žádného řádu. Čeleď obsahuje 5 rodů (Influenza virus A, Influenza virus B, Influenza virus C, Isavirus a Togotovirus). Viry způsobující chřipku (Influenza viry) napadají obratlovce, zejména savce (člověka a prasata) a ptáky. Isavirus napadá lososovité a Togotovirus infikuje obratlovce i bezobratlé, mezi které patří moskyti a mořské vši.
Kapsid může mít sférický či vláknitý tvar; jsou obalené lipoproteinovou membránou Sférické kapsidy mají 50-120 nm v průměru, zatímco vláknité mají 20 nm v průměru a 200-300 (až 3000) nm na délku.
Hlavní glykoproteinový lektin hemaglutinin (HA, H), který zodpovídá za navázání na receptory buněk, je místy nahrazen enzymem neuraminidázou (NA, N), který zodpovídá za vypučení z buňky, v poměru 4-5:1. Hemaglutinin má za úkol se doslova nalepit na buňku a zprostředkovat tak splynutí s hostitelskou membránou, zatímco neuraminidáza štěpí glykoproteiny, díky schopnosti štěpit glykosidové vazby mezi kyselinou sialovou a galaktózou. Děje se tak proto, aby se viry, které jsou obaleny hostitelskou membránou, nepřilepily k sobě a zároveň byly schopny odpoutat se od buňky. Antivirotika se zaměřují právě na tyto dva proteiny. Zároveň jsou rozeznávány imunitním systémem, díky čemuž slouží jako antigeny. Také slouží k určování rozdílných sérotypů.
Genom Orthomyxoviridae se skládá z 6-8 segmentů lineární záporné jednořetězcové RNA.
Velikost je 12000-15000 bp. Největší (první) segment má 2300-2500 bp, druhý má 2300-2500 bp, třetí má 2200-2300 bp, čtvrtý má 1700-1800 bp, pátý má 1500-1600 bp, šestý 1400-1500 bp, sedmý 1000-1100 bp a osmý 800-900 bp. Každý segment má na obou koncích ukončující repetitivní sekvence. Na 5' konci je tato sekvence dlouhá 12-13 bp, na 3' konci je 9-11 bp dlouhá.
Chřipka se typicky mezi savci přenáší kapénkovou infekcí, mezi ptáky pak jejich výkaly. Může být přenášena také například slinami či krví. Nákaza se šíří přímým kontaktem s těmito tekutinami nebo kontaktem s kontaminovanými povrchy. Chřipkové viry dokáží týden přečkat při teplotách lidského těla, více než měsíc při teplotě 0 °C a po neurčitou dobu při velmi nízkých teplotách (jezera na severní Sibiři). Mohou být ale lehce znehodnoceny desinfekcí, detergenty, teplotou 56 °C (po dobu minimálně 60 minut) nebo pH nižším než 2.
Virus se na buňku naváže prostřednictvím HA na cukry s kyselinou sialovou na povrchu epitelu v plicích a hrdle (krok 1). Buňka poté virus transportuje do endozómu prostřednictvím endocytózy. Část HA sfůzuje s membránou a uvolní tak virovou RNA (vRNA), příslušné proteiny a RNA dependentní RNA polymerázy do cytoplasmy (krok 2). Následně se z těchto jednotek vytvoří komplex, který je transportován do jádra, kde RNA dependentní RNA polymeráza vytvoří komplementární pozitivní cRNA (krok 3a a 3b). cRNA je buď exportována do cytoplasmy a následně translatována (krok 4) nebo zůstane v jádře. Chřipkové viry nekódují aparát k vytvoření 5' čepičky na vlastní mRNA, ale odštěpují 5' čepičku z hostitelské mRNA a navážou ji na vlastní. Nově syntetizované proteiny jsou buďto díky Golgiho aparátu přeneseny na povrch buňky (jako v případě HA a NA (krok 5b)), nebo přeneseny do jádra, kde se váží na vRNA a vytvoří nové virové genové částice (krok 5a). Další virové proteiny mají mnoho funkcí, například štěpí buněčnou mRNA, aby vir získal dostatek nukleotidů k syntéze vRNA, nebo inhibují translací hostitelských mRNA.
Negativní vRNA tvořící genom budoucích virů, polymerázy a další virové proteiny utvoří virion. HA a NA se na povrchu buňky shluknou v bouli, virové jádro opustí buněčné jádro a vstoupí do tohoto výběžku (krok 6). Dospělý virus vypučí z buňky obalen lipoproteinovou membránou buňky obohacenou o HA a NA (krok 7). Cyklus znovu začíná navázáním HA na hostitelskou buňku, přičemž NA vyčistí novou membránu od receptorů vázající HA. Po uvolnění nové generace dochází ke smrti buňky.
Kvůli absenci RNA opravných enzymů zůstává chyba (chybné vložení jednoho nukleotidu každých 10 kbp) RNA dependentní RNA polymerázy neopravena. to se téměř rovná velikosti genomu chřipkových virů, což vede k tomu, že téměř každý nový virus nese mutaci. Segmentace genomu do více částí také dovoluje výměnu jednotlivých segmentů mezi více variantami virů při superinfekci (děj, kdy jednu buňku napadne více variant virů najednou). To vede k získání nových vlastností viru, například schopnosti infikovat nového hostitele nebo překonání imunitního systému starého hostitele (děj se nazývá antigenní posun).
Jde o jediný druh z rodu Influenzavirus A. Hostitelé jsou lidé, prasata, ptáci a koně. Jsou klasifikováni na základě proteinů hemaglutininu a neuramidinázy. Dodnes bylo popsáno několik sérotypů s 16 variantami HA a 9 variantami NA. Další klasifikace berou v úvahu typ viru, místo a rok první izolace a sekvenční číslo izolace (dosl. překlad sequential number of isolation): A/Brisbane/59/2007 (H1N1).
Influenzavirus A je nejvirulentnější Influenzavir a způsobuje nejvíce vážných onemocnění. Následující sérotypy jsou seřazeny podle počtu obětí:
Virion má 80-120 nm v průměru a je v drtivé většině sférický, avšak ani vláknité formy nejsou neobvyklé. Jeho genom je tvořen 8 segmenty o celkové délce 13,5 kbp, které kódují 11 proteinů:
Jde o jediný druh z rodu Influenzavirus B. Hostitelé jsou téměř výhradně lidé. Jediný další známý tvor, který je virem napadnutelný, je tuleň. Je méně častý než Influenzavirus A, má menší mutační rychlost (2-3×) a tudíž je méně geneticky rozmanitý a má jediný sérotyp. Díky tomu dochází k imunizaci v nízkém věku, avšak nestačí k trvalé ochraně. Všechny tyto aspekty (malá rozmanitost, specializace na hostitele) zabraňují Influenzaviru B vyvolat pandemie.
Jde o jediný druh z rodu Influenzavirus C. Infikuje lidi a člověka a může vyvolat vážná onemocnění a lokální epidemie. Je však ze všech Influenzavirů nejméně častý a většinou jen vyvolává mírně onemocnění u dětí.
Rod obsahuje jediný druh Infectious salmon anemia virus (ISAV), který způsobuje anemii lososovitých. Přenos je uskutečňován přímým kontaktem ryb, anebo kontaktem s jejich sekretem, kontaminovaným vybavením, či člověkem, který držel nakaženou rybu. Nedávno se však prokázal pasivní přenos skrze Lepeophtheirus salmonis, kdy virus cestuje na povrchu parazita a čeká na poškození tkání. Virus však také přežívá volně ve vodě a může tak hrozit nákazou celé rybí farmě.
Jak jméno napovídá, způsobují anémii ryb. Jejich erytrocyty totiž mají jádro s DNA a tak mohou být napadeny virem. Nemoc se projevuje bledými žábrami a lapáním po vzduchu na hladině. Někdy se ale nemoc neprojeví a může dojít k pomalé nákaze celé farmy, kdy se úmrtnost vyšplhá až na 100%. Játra a slezina otečou a částečně odumřou. Kardiovaskulární systém se zastaví, počet nezralých a poškozených erytrocytů narůstá.
Togotoviry napadají obratlovce a klíšťata, kterými jsou také obvykle přenášena. Nenakažené klíště se může nakazit od nakaženého klíštěte při společném krmení na hostiteli, který však nevykazuje příznaky infekce. Rod zahrnuje dva druhy, Thogoto a Dhori.
Řadí se do skupiny V - (-)ssRNA viry. Vir byl izolován z klíšťat v Africe a jižní Evropě. Je schopen replikovat se v lidských buňkách, lidi napadá hlavně v přírodních podmínkách. Může se přenášet mezi nakaženými a nenakaženými klíšťaty pokud se společně živí na neinfikovaných morčatech, například. Má 6 RNA segmentů. [1]
[1] Index of Viruses - Orthomyxoviridae (2006). In: ICTVdB - The Universal Virus Database, version 4. Büchen-Osmond, C (Ed), Columbia University, New York, USA External links
Vir byl izolován z klíšťat v Indii, východním Rusku a Egyptě a v jižním Portugalsku.
Orthomyxoviridae (z řeckého orthos pro hladký a myxa pro sliz) je čeleď virů V.skupiny, která společně s čeleděmi Ophioviridae, Bunyaviridae a Arenaviridae v současnosti není zařazena do žádného řádu. Čeleď obsahuje 5 rodů (Influenza virus A, Influenza virus B, Influenza virus C, Isavirus a Togotovirus). Viry způsobující chřipku (Influenza viry) napadají obratlovce, zejména savce (člověka a prasata) a ptáky. Isavirus napadá lososovité a Togotovirus infikuje obratlovce i bezobratlé, mezi které patří moskyti a mořské vši.
Orthomyxoviridae (orthos græsk for "lige" myxa græsk for "mucus")[1] Er en RNA-virus-familie med fem slægter (medlemmer): Influenzavirus A, Influenzavirus B, Influenzavirus C, Isavirus og Thogotovirus. Et sjette medlem er for nylig blevet beskrevet.[2] De tre førstnævnte medlemmer er virus som medfører influenza i vertebrater som fugle, menneske og andre pattedyr. Isavirus inficerer laks og thogotoviruses inficerer både vertebrater og invertebrater såsom myg og sølus.[3][4][5]
Orthomyxoviridae (orthos græsk for "lige" myxa græsk for "mucus") Er en RNA-virus-familie med fem slægter (medlemmer): Influenzavirus A, Influenzavirus B, Influenzavirus C, Isavirus og Thogotovirus. Et sjette medlem er for nylig blevet beskrevet. De tre førstnævnte medlemmer er virus som medfører influenza i vertebrater som fugle, menneske og andre pattedyr. Isavirus inficerer laks og thogotoviruses inficerer både vertebrater og invertebrater såsom myg og sølus.
Die Familie Orthomyxoviridae (griechisch μύξα myxa, deutsch ‚Schleim‘) umfasst behüllte Viren mit einzelsträngiger RNA mit negativer Polarität als Genom. Ihr RNA-Genom ist auf mehrere Segmente verteilt, weshalb sie im Gegensatz zu den Paramyxoviridae nicht der Ordnung Mononegavirales zugerechnet werden. Die Segmentierung ihres Genoms ermöglicht den Orthomyxoviridae eine hohe genetische Flexibilität und Anpassungsfähigkeit an neue Wirtsspezies aufgrund der Durchmischung der verschiedenen Segmente verschiedener Subtypen und Mutanten durch das sogenannte Reassortment.
Zu den Orthomyxoviridae gehören Virusgattungen, die hauptsächlich über Tröpfcheninfektion das respiratorische System eines Wirts infizieren und sich darin vermehren. Dies gilt besonders für die Gattungen der Influenzaviren, die bei Säugetieren und Vögeln symptomlose Infektionen oder schwere Erkrankungen hervorrufen können. Lediglich die Spezies der Gattung Thogotovirus verursachen keine respiratorischen Infektionen und werden durch Zecken auf Wirbeltiere übertragen.[3] Einige im Wasser lebende Wirte der Gattungen Influenzavirus A (Bartenwale)[4] und Isavirus (Lachse) werden durch kontaminiertes Wasser, direkten Kontakt oder (beim Virus der infektiösen Lachsanämie) durch Fischläuse infiziert. Als „orthomyxovirus-ähnlich“ wurde ferner das Tilapia-Teich-Virus wissenschaftlich beschrieben[5] und vom ICTV in eine neue Schwesterfamilie Amnoonviridae, Gattung Tilapinevirus gestellt.
Die Viruspartikel (Virionen) der Orthomyxoviridae sind kugelförmig bis unregelmäßig und 80–120 nm im Durchmesser groß. Auch fadenförmige (filamentöse) Formen mit Längen bis zu wenigen µm werden beobachtet. In die lipidhaltige Virushülle sind 1–3 Glykoproteine und 1–2 nicht-glykosylierte Proteine eingelagert. Diese bilden 10–14 nm lange und 4–6 nm im Durchmesser große sichtbare „Spikes“ auf der Oberfläche. In der Hülle verankert, jedoch mit dem größeren Anteil nach innen zeigend, sind sogenannte Matrixproteine, die den Raum zwischen Hülle und Kapside (Matrixraum) auskleiden.
In den Virionen finden sich je nach Segmentierung des Genoms auch mehrere helikale Kapside, an deren einem Ende jeweils mehrere Untereinheiten der viralen Polymerase-Proteine (PA, PB1 und PB2) assoziiert sind. Diese viralen Enzyme zeigen je nach Virusgattung unterschiedliche Aktivitäten, z. B. ist das PB1 bei Influenzaviren eine Endonuklease und zusätzlich bei diesen und der Gattung Thogotovirus eine RNA-Polymerase (Transkriptase). Die Kapside werden nach Freisetzung im Zytoplasma durch spezifischen Kerntransport („nuclear import“) in den Zellkern transportiert.[6]
Das (-)ssRNA-Genom ist linear und segmentiert; die Anzahl der Segmente variiert zwischen den Gattungen. So besitzen die Spezies der Gattungen Influenzavirus A, Influenzavirus B und Isavirus jeweils 8 Segmente, Influenzavirus C und aus der Gattung Thogotovirus die Spezies Dhori-Virus 7, die Spezies Thogoto-Virus 6 Segmente. Die Größe der Segmente reicht von 874 bis 2396 nt, die Gesamtgröße des Genoms von 10,0 bis 14,6 kb. Durch Verteilungs- und Synthesefehler während der Virusvermehrung besitzen viele Virionen kürzere, defekte RNA-Stücke oder verfügen über keinen kompletten Segmentsatz. Dies wird besonders bei Mutationen der Polymerase-Untereinheit PA beobachtet,[7] die für die korrekte Verpackung und Verteilung der Genomsegmente offenbar eine entscheidende Rolle spielt.
Mit Stand März 2019 ist die Systematik der Orthomyxoviridae gemäß International Committee on Taxonomy of Viruses (ICTV) wie folgt:[2]
Die einzelnen Viruslinien und weitere unklassifizierte Kandidaten finden sich beim NCBI.[8]
Die Familie Orthomyxoviridae (griechisch μύξα myxa, deutsch ‚Schleim‘) umfasst behüllte Viren mit einzelsträngiger RNA mit negativer Polarität als Genom. Ihr RNA-Genom ist auf mehrere Segmente verteilt, weshalb sie im Gegensatz zu den Paramyxoviridae nicht der Ordnung Mononegavirales zugerechnet werden. Die Segmentierung ihres Genoms ermöglicht den Orthomyxoviridae eine hohe genetische Flexibilität und Anpassungsfähigkeit an neue Wirtsspezies aufgrund der Durchmischung der verschiedenen Segmente verschiedener Subtypen und Mutanten durch das sogenannte Reassortment.
Zu den Orthomyxoviridae gehören Virusgattungen, die hauptsächlich über Tröpfcheninfektion das respiratorische System eines Wirts infizieren und sich darin vermehren. Dies gilt besonders für die Gattungen der Influenzaviren, die bei Säugetieren und Vögeln symptomlose Infektionen oder schwere Erkrankungen hervorrufen können. Lediglich die Spezies der Gattung Thogotovirus verursachen keine respiratorischen Infektionen und werden durch Zecken auf Wirbeltiere übertragen. Einige im Wasser lebende Wirte der Gattungen Influenzavirus A (Bartenwale) und Isavirus (Lachse) werden durch kontaminiertes Wasser, direkten Kontakt oder (beim Virus der infektiösen Lachsanämie) durch Fischläuse infiziert. Als „orthomyxovirus-ähnlich“ wurde ferner das Tilapia-Teich-Virus wissenschaftlich beschrieben und vom ICTV in eine neue Schwesterfamilie Amnoonviridae, Gattung Tilapinevirus gestellt.
Orthomyxoviridae iku famili virus RNA sing nyakup limang genera: Influenzavirus A, Influenzavirus B, Influenzavirus C, Thogotovirus lan Isavirus. Telu genera pisanan bisa njalari influensa ing vertebrata, kalebu manuk (uga pirsani flu burung), manungsa lan mamalia liyané. Isaviruses nginfèksi salmon; thogotoviruses nginfèksi vertebrata lan invertebrata, kaya déné lemut.[1][2][3][4]
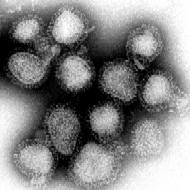
Influenza-wiirus oner't elektronenmikroskop
Süstemaatik Hoodkategorii: Wiiren Kategorii: - Famile: Orthomyxoviridae Taksonomii Genoom: (-)ssRNA, segmentiaret Baltimore-klas: Skööl 5 Sümetrii: helikal Wiirusskan: diar Wedenskapelk nööm Orthomyxoviridae (ingelsk) FerwisangenOrthomyxoviridae (faan griichisk myxa: slim) san en famile faan wiiren an hiar faan hör struktuur tu a RNA-wiiren. Fööraal a influenza-wiiren kön minsken an ööder tetjdiarten, oober uk fögler kraank maage. Jo fu diar gripe faan.
Orthomyxoviridae iku famili virus RNA sing nyakup limang genera: Influenzavirus A, Influenzavirus B, Influenzavirus C, Thogotovirus lan Isavirus. Telu genera pisanan bisa njalari influensa ing vertebrata, kalebu manuk (uga pirsani flu burung), manungsa lan mamalia liyané. Isaviruses nginfèksi salmon; thogotoviruses nginfèksi vertebrata lan invertebrata, kaya déné lemut.
Orthomyxoviridae (faan griichisk myxa: slim) san en famile faan wiiren an hiar faan hör struktuur tu a RNA-wiiren. Fööraal a influenza-wiiren kön minsken an ööder tetjdiarten, oober uk fögler kraank maage. Jo fu diar gripe faan.
Οι ιοί της γρίπης ή Ορθομυξοϊοί (Orthomyxoviridae) είναι μια οικογένεια RNA ιών που περιλαμβάνει έξι γένη: το ιό γρίπης Α, τον ιό γρίπης Β, τον ιό γρίπης C, τον ισαϊό, τον θογκοτοϊό και ένα γένος που ακόμη δεν έχει περιγραφεί. Τα τρία πρώτα γένη περιλαμβάνουν ιούς που μολύνουν τα σπονδυλωτά, συμπεριλαμβανομένων των πτηνών (βλέπε επίσης γρίπη των πτηνών), τους ανθρώπους και άλλα θηλαστικά, ο ισαϊός προσβάλει τους σολομούς, ενώ ο θογκοτοϊός προσβάλει τόσο σπονδυλωτά όσο και ασπόνδυλα.
Το ιοσωμάτιο είναι πλειομορφικό· ο φάκελος μπορεί να έχει είτε σφαιρική και νηματοειδή μορφή. Σε γενικές γραμμές, η μορφολογία του ιού είναι σφαιρικά σωματίδια με 50 έως 120 nm σε διάμετρο ή νηματοειδή ιοσωμάτια διαμέτρου 20 nm και μήκους 200 έως 300 (-3000) nm. Υπάρχουν περίπου 500 διακριτές ακιδόμορφες προβολές στην επιφάνεια του περιβλήματος, οι οποίες προεξέχουν 10 έως 14 nm από την επιφάνεια με μερικούς τύπους (π.χ. εστεράσης αιμοσυγκολλητίνης (HEF)) πυκνά διεσπαρμένους πάνω στην επιφάνεια και με τους άλλους (π.χ. αιμοσυγκολλητίνης (ΗΑ)) απέχοντα πολύ. Η μεγάλη γλυκοπρωτεΐνη (ΗΑ) παρεμβάλλεται ακανόνιστα από συστάδες νευραμινιδάσης (ΝΑ), με αναλογία ΗΑ προς ΝΑ από 4-5 έως περίπου 1. Οι μεμβρανικές λιποπρωτεΐνες επισυνάπτονται στα πυρηνικά καψίδια· νουκλεοπρωτεΐνες διαφόρων κατηγοριών μεγέθους με μια θηλιά σε κάθε άκρο. Η διάταξη εντός του λοιμογόνου παράγοντα είναι αβέβαιη. Τα πυρηνοκαψίδια είναι νηματοειδή και έχουν μήκος από 50 έως 130 nm και διάμετρο 9 έως 15 nm. Έχουν ελικοειδή συμμετρία.
Οι ιοί αυτής της οικογένειας περιέχουν 6 έως 8 τμήματα του γραμμικού μονόκλωνου RNA με αρνητική πολικότητα.[1]
Το συνολικό μήκος του γονιδιώματος είναι 12000-15000 νουκλεοτίδια (nt). Το μεγαλύτερο τμήμα είναι 2300-2500 nt, το δεύτερο μεγαλύτερο 2300-2500 nt, το τρίτο 2200-2300 nt, το τέταρτο 1700-1800 nt, το πέμπτο 1500-1600 nt, το έκτο 1400-1500 nt, το έβδομο 1000-1100 nt και το όγδοο 800-900 nt. Το γονιδίωμα έχει τερματικές επαναλαμβανόμενες αλληλουχίες, ενώ επαναλαμβάνεται σε αμφότερα τα άκρα. Οι τερματικές επαναλήψεις στο 5'-άκρο έχουν μήκος 12-13 νουκλεοτίδια. Νουκλεοτιδικές αλληλουχίες του 3'-άκρου είναι όμοιες στα γένη της οικογένειας και έχουν μήκος 9-11 νουκλεοτίδια. Το νουκλεϊκό οξύ στο καψίδιο είναι αποκλειστικά το γονιδίωμα. Κάθε τμήμα ιού μπορεί να περιέχει ελαττωματικά αντίγραφα.
Το γονιδίωμα κωδικοποιεί 11 πρωτεΐνες (HA, NA, NP, M1, M2, NS1, ΝΕΠ, PA, PB1, PB1-F2, PB2).[2] Από αυτές οι καλύτερα χαρακτηρισμένες είναι η αιμοσυγκολλητίνη (HA) και η νευραμινιδάση (NA), δύο μεγάλες γλυκοπρωτεΐνες που ανευρίσκονται στο εξωτερικό των ιικών σωματιδίων. Η νευραμινιδάση είναι ένα ένζυμο που εμπλέκεται στην απελευθέρωση των νέων ιών από τα μολυσμένα κύτταρα, με διάσπαση των σακχάρων που δεσμεύουν τα ώριμα ιοσωμάτια. Αντίθετα, η αιμοσυγκολλητίνη είναι μία λεκτίνη που μεσολαβεί πρόσδεση του ιού σε κύτταρα στόχους και την είσοδο του ιικού γονιδιώματος στο κύτταρο στόχο.[3] Η αιμοσυγκολλητίνη (H) και νευραμινιδάση (N) είναι στόχοι για αντι-ιικά φάρμακα.[4] Αυτές οι πρωτεΐνες, επίσης, αναγνωρίζονται από τα αντισώματα, δηλαδή είναι αντιγόνα. Οι αποκρίσεις των αντισωμάτων έναντι αυτών των πρωτεϊνών που χρησιμοποιούνται για την ταξινόμηση των διαφορετικών οροτύπων ιών της γρίπης Α, εξ ου και το H και Ν στην Η5Ν1.
Τυπικά, η γρίπη μεταδίδεται από μολυσμένα θηλαστικά αερογενώς, από το βήχα ή το φτέρνισμα, δημιουργώντας αερολύματα που περιέχουν τον ιό, καθώς και από μολυσμένα πτηνά μέσω των περιττωμάτων τους. Η γρίπη μπορεί επίσης να μεταδοθεί με το σάλιο, ρινικές εκκρίσεις, περιττώματα και αίμα. Οι λοιμώξεις συμβαίνουν μέσα από την επαφή με αυτά τα σωματικά υγρά ή με μολυσμένες επιφάνειες. Ιοί της γρίπης μπορούν να παραμείνουν μολυσματικοί για περίπου μία εβδομάδα σε θερμοκρασία του ανθρώπινου σώματος, πάνω από 30 ημέρες στους 0° C (32° F), και επ 'αόριστον σε πολύ χαμηλές θερμοκρασίες (όπως λίμνες στη βορειοανατολική Σιβηρία). Μπορούν να απενεργοποιηθούν εύκολα με απολυμαντικά και απορρυπαντικά.[5][6][7]
Οι ιοί προσδένονται σε ένα κύτταρο μέσω αλληλεπιδράσεων μεταξύ της γλυκοπρωτεΐνης αιμοσυγκολλητίνη και του σιαλικού οξέος στην επιφάνεια των επιθηλιακών κυττάρων στον πνεύμονα και το λαιμό (στάδιο 1).[8] Το κύτταρο εισάγει τον ιό με ενδοκυττάρωση. Στο όξινο ενδόσωμα, μέρος της πρωτεΐνης αιμοσυγκολλητίνης συγχωνεύεται το ιικό φάκελο και την μεμβράνη του κυστιδίου, απελευθερώνοντας τα μόρια του RNA του ιού (vRNA), βοηθητικές πρωτεΐνες και την RNA-εξαρτώμενη πολυμεράση RNA στο κυτταρόπλασμα (Στάδιο 2).[9] Αυτές οι πρωτεΐνες και τα vRNA σχηματίζουν ένα σύμπλοκο που μεταφέρεται εντός του πυρήνα του κυττάρου, όπου η RNA-εξαρτώμενη RNA πολυμεράση αρχίζει μεταγραφή στο συμπληρωματικό θετικής πολικότητας-cRNA (Στάδιο 3α και β).[10] Το cRNA είτε εξάγεται στο κυτταρόπλασμα και μεταφράζεται (στάδιο 4) ή παραμένει στον πυρήνα. Οι προσφάτως συντεθημένες ιικές πρωτεΐνες είτε εκκρίνονται μέσω της συσκευής Golgi πάνω στην κυτταρική επιφάνεια (στην περίπτωση της νευραμινιδάσης και της αιμοσυγκολλητίνης, στάδιο 5b) ή μεταφέρονται πίσω στον πυρήνα για να δεσμεύσουν vRNA και να σχηματίζουν νέα ιικά σωματίδια γονιδιώματος (5α βήμα). Άλλες ιικές πρωτεΐνες έχουν πολλαπλές δράσεις στο κύτταρο ξενιστή, συμπεριλαμβανομένης της αποικοδόμησης του κυτταρικού mRNA, χρησιμοποιώντας τα νουκλεοτίδια που κυκλοφορούν για την σύνθεση vRNA και επίσης αναστέλλουν μετάφραση των mRNAs του κυττάρου-ξενιστή.[11]
Τα αρνητικής πολικότητας vRNAs, που αποτελούν το γονιδιώμα των μελλοντικών ιών, η RNA-εξαρτώμενη RNA μεταγραφάση και άλλες ιικές πρωτεΐνες συναρμολογούνται σε ένα ιοσωμάτιο. Το σύμπλεγμα μορίων αιμοσυγκολλητίνης και νευραμινιδάσης δημιουργούν ένα εξόγκωμα στην κυτταρική μεμβράνη. Το vRNA και οι ιικές πρωτεΐνες του πυρήνα φεύγουν από τον πυρήνα και μεταφέονται στη προεξοχή της μεμβράνης (βήμα 6). Τα ώριμα σωμάτιου του ιού μακριά από το κύτταρο, σε μια σφαίρα από τη φωσφολιπιδική μεμβράνη του ξενιστή, η οποία στη συνέχεια αποκτά νευραμινιδάση και αιμοσυγκολιτίνη (βήμα 7).[12] Όπως και πριν, οι ιοί προσδένονται στο κύτταρο μέσω αιματογλουτινίνης. Οι ώριμοι ιούς αποκολλώνται όταν η νευραμινιδάση διασπά το σιαλικό οξύ του κυττάρου ξενιστή.[8] Μετά από την απελευθέρωση του νέου ιού της γρίπης, το κύτταρο ξενιστής πεθαίνει.
Δεδομένου ότι τα ένζυμα επιδιόρθωσης του RNA είναι απόντα, η RNA-εξαρτώμενη RNA μεταγραφάση κάνει ένα νουκλεοτιδίο σφάλμα εισαγωγής περίπου κάθε 10 χιλιάδες νουκλεοτίδια, όσο είναι κατά προσέγγιση μήκος του RNA της γρίπης. Ως εκ τούτου, σχεδόν κάθε νεοκατασκευασθής ιός της γρίπης θα περιέχει μια μετάλλαξη στο γονιδίωμά του.[13] Ο διαχωρισμός του γονιδιώματος σε οκτώ ξεχωριστά τμήματα του vRNA επιτρέπει την ανάμιξη (ανακατάταξη) των γονιδίων, εάν περισσότερες από μία ποικιλία του ιού της γρίπης έχουν μολύνει το ίδιο κύτταρο (επιμόλυνση). Η προκύπτουσα μεταβολή στα τμήματα του γονιδιώματος των νέων τους προσδίδει νέα συμπεριφορά, μερικές φορές την ικανότητα να μολύνει νέο είδος ξενιστή ή να ξεπεράσουν ανοσία του ξενιστή, η οποία βασιζόταν στο παλιό γονιδίωμα του (αυτή η περίπτωση ονομάζεται αντιγονική μετατόπιση).
Οι ιοί της γρίπης ή Ορθομυξοϊοί (Orthomyxoviridae) είναι μια οικογένεια RNA ιών που περιλαμβάνει έξι γένη: το ιό γρίπης Α, τον ιό γρίπης Β, τον ιό γρίπης C, τον ισαϊό, τον θογκοτοϊό και ένα γένος που ακόμη δεν έχει περιγραφεί. Τα τρία πρώτα γένη περιλαμβάνουν ιούς που μολύνουν τα σπονδυλωτά, συμπεριλαμβανομένων των πτηνών (βλέπε επίσης γρίπη των πτηνών), τους ανθρώπους και άλλα θηλαστικά, ο ισαϊός προσβάλει τους σολομούς, ενώ ο θογκοτοϊός προσβάλει τόσο σπονδυλωτά όσο και ασπόνδυλα.
ऑर्थोमिक्सोविरिडि (Orthomyxoviridae) (यूनानी भाषा मे: ऑर्थोस: "सीधा"; मिक्सा: "श्लेष्म") आर एन ए विषाणुओं का एक कुल है जिसमे विषाणुओं के पाँच वंश शामिल हैं: इन्फ्लुएंजाविषाणु ए, इन्फ्लुएंजाविषाणु बी, व्एंजाविषाणु सी, आइसाविषाणु और थोगोटोविषाणु। पहले तीन वंशों में वह विषाणु शामिल हैं जो कशेरुकी जन्तुओं को इन्फ्लुएंजा से संक्रमित करते हैं, जिनमे पक्षी, मनुष्य और अन्य स्तनधारी शामिल हैं। आइसाविषाणु सैमन को संक्रमित करते हैं; थोगोटोविषाणु कशेरुकी और अकशेरुकी जीवों जैसे मच्छर और समुद्री जूँ को संक्रमित करते हैं।
इन्फ्लूएंजाविषाणु के तीन वंश जिनकी पहचान उनके न्यूक्लियोप्रोटीन और मैट्रिक्स प्रोटीन में मौजूद प्रतिजनी अंतरों से होती है, कशेरुकी जीवों को निम्नानुसार संक्रमित करते हैं:
ऑर्थोमिक्सोविरिडि (Orthomyxoviridae) (यूनानी भाषा मे: ऑर्थोस: "सीधा"; मिक्सा: "श्लेष्म") आर एन ए विषाणुओं का एक कुल है जिसमे विषाणुओं के पाँच वंश शामिल हैं: इन्फ्लुएंजाविषाणु ए, इन्फ्लुएंजाविषाणु बी, व्एंजाविषाणु सी, आइसाविषाणु और थोगोटोविषाणु। पहले तीन वंशों में वह विषाणु शामिल हैं जो कशेरुकी जन्तुओं को इन्फ्लुएंजा से संक्रमित करते हैं, जिनमे पक्षी, मनुष्य और अन्य स्तनधारी शामिल हैं। आइसाविषाणु सैमन को संक्रमित करते हैं; थोगोटोविषाणु कशेरुकी और अकशेरुकी जीवों जैसे मच्छर और समुद्री जूँ को संक्रमित करते हैं।
इन्फ्लूएंजाविषाणु के तीन वंश जिनकी पहचान उनके न्यूक्लियोप्रोटीन और मैट्रिक्स प्रोटीन में मौजूद प्रतिजनी अंतरों से होती है, कशेरुकी जीवों को निम्नानुसार संक्रमित करते हैं:
इन्फ्लूएंजाविषाणु ए सभी फ्लू महामारियां का कारण मनुष्यों अन्य स्तनधारियों और पक्षियों को संक्रमित करता है इन्फ्लूएंजाविषाणु बी मानव और सील को संक्रमित करता है इन्फ्लूएंजाविषाणु सी मानव और सूअरों को संक्रमित करता है।Orthomyxoviridae (from Greek ὀρθός, orthós 'straight' + μύξα, mýxa 'mucus')[1] is a family of negative-sense RNA viruses. It includes seven genera: Alphainfluenzavirus, Betainfluenzavirus, Gammainfluenzavirus, Deltainfluenzavirus, Isavirus, Thogotovirus, and Quaranjavirus. The first four genera contain viruses that cause influenza in birds (see also avian influenza) and mammals, including humans. Isaviruses infect salmon; the thogotoviruses are arboviruses, infecting vertebrates and invertebrates (such as ticks and mosquitoes).[2][3][4] The Quaranjaviruses are also arboviruses, infecting vertebrates (birds) and invertebrates (arthropods).
The four genera of Influenza virus that infect vertebrates, which are identified by antigenic differences in their nucleoprotein and matrix protein, are as follows:
The influenzavirus virion is pleomorphic; the viral envelope can occur in spherical and filamentous forms. In general, the virus's morphology is ellipsoidal with particles 100–120 nm in diameter, or filamentous with particles 80–100 nm in diameter and up to 20 µm long.[5] There are approximately 500 distinct spike-like surface projections in the envelope each projecting 10–14 nm from the surface with varying surface densities. The major glycoprotein (HA) spike is interposed irregularly by clusters of neuraminidase (NA) spikes, with a ratio of HA to NA of about 10 to 1.[6]
The viral envelope composed of a lipid bilayer membrane in which the glycoprotein spikes are anchored encloses the nucleocapsids; nucleoproteins of different size classes with a loop at each end; the arrangement within the virion is uncertain. The ribonuclear proteins are filamentous and fall in the range of 50–130 nm long and 9–15 nm in diameter with helical symmetry.
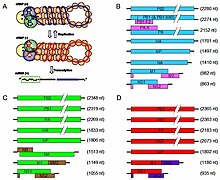
Viruses of the family Orthomyxoviridae contain six to eight segments of linear negative-sense single stranded RNA. They have a total genome length that is 10,000–14,600 nucleotides (nt).[7] The influenza A genome, for instance, has eight pieces of segmented negative-sense RNA (13.5 kilobases total).[8]
The best-characterised of the influenzavirus proteins are hemagglutinin and neuraminidase, two large glycoproteins found on the outside of the viral particles. Hemagglutinin is a lectin that mediates binding of the virus to target cells and entry of the viral genome into the target cell.[9] In contrast, neuraminidase is an enzyme involved in the release of progeny virus from infected cells, by cleaving sugars that bind the mature viral particles. The hemagglutinin (H) and neuraminidase (N) proteins are key targets for antibodies and antiviral drugs,[10][11] and they are used to classify the different serotypes of influenza A viruses, hence the H and N in H5N1.
The genome sequence has terminal repeated sequences; repeated at both ends. Terminal repeats at the 5′-end 12–13 nucleotides long. Nucleotide sequences of 3′-terminus identical; the same in genera of same family; most on RNA (segments), or on all RNA species. Terminal repeats at the 3′-end 9–11 nucleotides long. Encapsidated nucleic acid is solely genomic. Each virion may contain defective interfering copies. In Influenza A (H1N1) PB1-F2 is produced from an alternative reading frame in PB1. The M and NS genes produce two different genes via alternative splicing.[12]
Typically, influenza is transmitted from infected mammals through the air by coughs or sneezes, creating aerosols containing the virus, and from infected birds through their droppings. Influenza can also be transmitted by saliva, nasal secretions, feces and blood. Infections occur through contact with these bodily fluids or with contaminated surfaces. Out of a host, flu viruses can remain infectious for about one week at human body temperature, over 30 days at 0 °C (32 °F), and indefinitely at very low temperatures (such as lakes in northeast Siberia). They can be inactivated easily by disinfectants and detergents.[13][14][15]
The viruses bind to a cell through interactions between its hemagglutinin glycoprotein and sialic acid sugars on the surfaces of epithelial cells in the lung and throat (Stage 1 in infection figure).[16] The cell imports the virus by endocytosis. In the acidic endosome, part of the hemagglutinin protein fuses the viral envelope with the vacuole's membrane, releasing the viral RNA (vRNA) molecules, accessory proteins and RNA-dependent RNA polymerase into the cytoplasm (Stage 2).[17] These proteins and vRNA form a complex that is transported into the cell nucleus, where the RNA-dependent RNA polymerase begins transcribing complementary positive-sense cRNA (Steps 3a and b).[18] The cRNA is either exported into the cytoplasm and translated (step 4), or remains in the nucleus. Newly synthesised viral proteins are either secreted through the Golgi apparatus onto the cell surface (in the case of neuraminidase and hemagglutinin, step 5b) or transported back into the nucleus to bind vRNA and form new viral genome particles (step 5a). Other viral proteins have multiple actions in the host cell, including degrading cellular mRNA and using the released nucleotides for vRNA synthesis and also inhibiting translation of host-cell mRNAs.[19]
Negative-sense vRNAs that form the genomes of future viruses, RNA-dependent RNA transcriptase, and other viral proteins are assembled into a virion. Hemagglutinin and neuraminidase molecules cluster into a bulge in the cell membrane. The vRNA and viral core proteins leave the nucleus and enter this membrane protrusion (step 6). The mature virus buds off from the cell in a sphere of host phospholipid membrane, acquiring hemagglutinin and neuraminidase with this membrane coat (step 7).[20] As before, the viruses adhere to the cell through hemagglutinin; the mature viruses detach once their neuraminidase has cleaved sialic acid residues from the host cell.[16] After the release of new influenza virus, the host cell dies.

Orthomyxoviridae viruses are one of two RNA viruses that replicate in the nucleus (the other being retroviridae). This is because the machinery of orthomyxo viruses cannot make their own mRNAs. They use cellular RNAs as primers for initiating the viral mRNA synthesis in a process known as cap snatching.[21] Once in the nucleus, the RNA Polymerase Protein PB2 finds a cellular pre-mRNA and binds to its 5′ capped end. Then RNA Polymerase PA cleaves off the cellular mRNA near the 5′ end and uses this capped fragment as a primer for transcribing the rest of the viral RNA genome in viral mRNA.[22] This is due to the need of mRNA to have a 5′ cap in order to be recognized by the cell's ribosome for translation.
Since RNA proofreading enzymes are absent, the RNA-dependent RNA transcriptase makes a single nucleotide insertion error roughly every 10 thousand nucleotides, which is the approximate length of the influenza vRNA. Hence, nearly every newly manufactured influenza virus will contain a mutation in its genome.[23] The separation of the genome into eight separate segments of vRNA allows mixing (reassortment) of the genes if more than one variety of influenza virus has infected the same cell (superinfection). The resulting alteration in the genome segments packaged into viral progeny confers new behavior, sometimes the ability to infect new host species or to overcome protective immunity of host populations to its old genome (in which case it is called an antigenic shift).[10]
In a phylogenetic-based taxonomy, the category RNA virus includes the subcategory negative-sense ssRNA virus, which includes the order Articulavirales, and the family Orthomyxoviridae. The genera-associated species and serotypes of Orthomyxoviridae are shown in the following table.
Orthomyxovirus Genera, Species, and Serotypes Genus Species (* indicates type species) Serotypes or Subtypes Hosts Alphainfluenzavirus Influenza A virus* H1N1, H1N2, H2N2, H3N1, H3N2, H3N8, H5N1, H5N2, H5N3, H5N8, H5N9, H7N1, H7N2, H7N3, H7N4, H7N7, H7N9, H9N2, H10N7 Human, pig, bird, horse, bat Betainfluenzavirus Influenza B virus* Victoria, Yamagata[24] Human, seal Gammainfluenzavirus Influenza C virus* Human, pig Deltainfluenzavirus Influenza D virus* Pig, cattle Isavirus Infectious salmon anemia virus* Atlantic salmon Thogotovirus Thogotovirus* Tick, mosquito, mammal (including human) Dhori virus Batken virus, Bourbon virus, Jos virus Quaranjavirus[25] Quaranfil virus,* Johnston Atoll virusThere are four genera of influenza virus, each containing only a single species, or type. Influenza A and C infect a variety of species (including humans), while influenza B almost exclusively infects humans, and influenza D infects cattle and pigs.[26][27][28]
Influenza A viruses are further classified, based on the viral surface proteins hemagglutinin (HA or H) and neuraminidase (NA or N). 18 HA subtypes (or serotypes) and 11 NA subtypes of influenza A virus have been isolated in nature. Among these, the HA subtype 1-16 and NA subtype 1-9 are found in wild waterfowl and shorebirds and the HA subtypes 17-18 and NA subtypes 10-11 have only been isolated from bats.[29][30]
Further variation exists; thus, specific influenza strain isolates are identified by a standard nomenclature specifying virus type, geographical location where first isolated, sequential number of isolation, year of isolation, and HA and NA subtype.[31][32]
Examples of the nomenclature are:
The type A influenza viruses are the most virulent human pathogens among the three influenza types and cause the most severe disease. It is thought that all influenza A viruses causing outbreaks or pandemics originate from wild aquatic birds.[33] All influenza A virus pandemics since the 1900s were caused by Avian influenza, through Reassortment with human influenza strains (seasonal flu) or through adaptation in a mixing vessel (see 2009 swine flu pandemic).[34] The serotypes that have been confirmed in humans, ordered by the number of confirmed human deaths, are:
Influenza B virus is almost exclusively a human pathogen, and is less common than influenza A. The only other animal known to be susceptible to influenza B infection is the seal.[46] This type of influenza mutates at a rate 2–3 times lower than type A[47] and consequently is less genetically diverse, with only one influenza B serotype.[26] As a result of this lack of antigenic diversity, a degree of immunity to influenza B is usually acquired at an early age. However, influenza B mutates enough that lasting immunity is not possible.[48] This reduced rate of antigenic change, combined with its limited host range (inhibiting cross species antigenic shift), ensures that pandemics of influenza B do not occur.[49]
The influenza C virus infects humans and pigs, and can cause severe illness and local epidemics.[50] However, influenza C is less common than the other types and usually causes mild disease in children.[51][52]
This is a genus that was classified in 2016, the members of which were first isolated in 2011.[53] This genus appears to be most closely related to Influenza C, from which it diverged several hundred years ago.[54] There are at least two extant strains of this genus.[55] The main hosts appear to be cattle, but the virus has been known to infect pigs as well.
Mammalian influenza viruses tend to be labile, but can survive several hours in mucus.[56] Avian influenza virus can survive for 100 days in distilled water at room temperature, and 200 days at 17 °C (63 °F). The avian virus is inactivated more quickly in manure, but can survive for up to 2 weeks in feces on cages. Avian influenza viruses can survive indefinitely when frozen.[56] Influenza viruses are susceptible to bleach, 70% ethanol, aldehydes, oxidizing agents, and quaternary ammonium compounds. They are inactivated by heat of 133 °F (56 °C) for minimum of 60 minutes, as well as by low pH <2.[56]
Vaccines and drugs are available for the prophylaxis and treatment of influenza virus infections. Vaccines are composed of either inactivated or live attenuated virions of the H1N1 and H3N2 human influenza A viruses, as well as those of influenza B viruses. Because the antigenicities of the wild viruses evolve, vaccines are reformulated annually by updating the seed strains.
When the antigenicities of the seed strains and wild viruses do not match, vaccines fail to protect the vaccinees. In addition, even when they do match, escape mutants are often generated.
Drugs available for the treatment of influenza include Amantadine and Rimantadine, which inhibit the uncoating of virions by interfering with M2, and Oseltamivir (marketed under the brand name Tamiflu), Zanamivir, and Peramivir, which inhibit the release of virions from infected cells by interfering with NA. However, escape mutants are often generated for the former drug and less frequently for the latter drug.[57]
Orthomyxoviridae (from Greek ὀρθός, orthós 'straight' + μύξα, mýxa 'mucus') is a family of negative-sense RNA viruses. It includes seven genera: Alphainfluenzavirus, Betainfluenzavirus, Gammainfluenzavirus, Deltainfluenzavirus, Isavirus, Thogotovirus, and Quaranjavirus. The first four genera contain viruses that cause influenza in birds (see also avian influenza) and mammals, including humans. Isaviruses infect salmon; the thogotoviruses are arboviruses, infecting vertebrates and invertebrates (such as ticks and mosquitoes). The Quaranjaviruses are also arboviruses, infecting vertebrates (birds) and invertebrates (arthropods).
The four genera of Influenza virus that infect vertebrates, which are identified by antigenic differences in their nucleoprotein and matrix protein, are as follows:
Alphainfluenzavirus infects humans, other mammals, and birds, and causes all flu pandemics Betainfluenzavirus infects humans and seals Gammainfluenzavirus infects humans and pigs Deltainfluenzavirus infects pigs and cattle.Orthomyxoviridae es una familia de virus de ARN que infectan a los animales.[3] Incluyen a los virus causantes de la gripe.
La familia Orthomyxoviridae incluye varios géneros de virus[4] que infectan a diferentes animales:[cita requerida]
Se caracteriza por presentar envoltura y ser isométrico, con la siguiente morfología (del interior al exterior):
La hemaglutinina (HA) es un trímero formado por tres polipéptidos cada uno de los cuales tiene una secuencia señal. Tras ella, aparece un dominio rico en sitios de glicosilación, y finalmente, una zona hidrófoba: el péptido fusogénico, que va a ser el responsable de la fusión de la membrana vírica con la celular para permitir la entrada a la célula. Por último, otra zona con la que se ancla a la membrana.
Se caracteriza por tener varios puentes disulfuro internos responsables de la estructura. Para que la proteína sea funcional, debe producirse un corte justo antes del péptido fusogénico. Si no se produce este corte, la proteína es afuncional y el virus no puede infectar. Las dos partes cortadas de la molécula siguen unidas por puentes disulfuro.
La neuraminidasa (NA) es un tetrámero formado por pequeños polipéptidos con una región de anclaje a membrana y una región globular funcional. Las zonas más expuestas funcionan en el reconocimiento del hospedador, pero también como determinantes antigénicos.
El genoma de los virus de Influenza están fragmentados en 8 moléculas (7 en Influenzavirus C): Las 6 primeras codifican una proteína cada una. Las 2 últimas, codifican dos proteínas cada una.
El fragmento 7 codifica la proteína de la matriz (M1)y una segunda proteína capaz de funcionar como canal iónico en la membrana (M2).
El fragmento 8 codifica proteínas que mejoran la efectividad de la infección. Algunas zonas del genoma se aparean para ganar resistencia contra ARNasas celulares. Todos los fragmentos están unidos a nucleoproteínas. En el interior de la partícula, además, está la ARN-Pol, que es un complejo de tres proteínas codificadas por los fragmentos 1, 2 y 3.
El ARN del virus, solo funciona como molde para la ARN-Pol si está unido a las nucleoproteínas. Se producen dos procesos de polimerización: Formación de ARNm y formación de ARN (+) como intermediario replicativo.
Toda la primera fase del ciclo replicativo es estándar de acuerdo a los virus ARN con polaridad negativa. La principal particularidad es que el proceso de replicación no se lleva a cabo si se trata con amantidina (inhibe la ARN-Pol celular) o si se infectan células anucleadas. Esto se debe a que los procesos de replicación y transcripción ocurren en el núcleo.
1. En el ciclo de infección del virus, las primeras dianas son células epiteliales en las que el virus reconoce restos de ácidos siálicos de las glicoproteínas de membrana a través de la hemaglutinina. Aquí actúa la neuraminidasa, que hidroliza estos restos de ácidos siálicos de las proteínas a las que pertenecen, evitando que el virus quede anclado a los ácidos siálicos de la mucoproteínas que recubren los epitelios.
2. La entrada se produce por endocitosis.[5] El endosoma se fusiona con un lisosoma, lo que baja el pH. Esto provoca un cambio en la hemaglutinina, que va a interaccionar con la membrana del endosoma. Las cabezas globulares de la proteína se apartan hacia un lateral y queda expuesto el péptido fusogénico, que es hidrófobo. Esta secuencia hidrófoba busca su estabilidad integrándose en la membrana del endosoma. En este momento, una región bisagra de la proteína acerca ambas membranas, facilitando su fusión.
Paralelamente a esto, la proteína M2 bombea protones al interior del virión, lo que induce la liberación de la proteína de la matriz y de los complejos ribonucleoproteícos, que al fundirse las membranas, salen libres al citoplasma.
3. El ingreso del genoma del virus en el núcleo se produce gracias a mecanismos celulares. Las nucleoproteínas tienen afinidad por una serie de intermediarios que interaccionan con el complejo del poro nuclear, permitiendo el acceso al núcleo. Esto solo puede ocurrir si las nucleoproteínas están libres, es decir, no asociadas a la proteína de la matriz.
4. Una vez en el núcleo se produce la transcripción. La lleva a cabo la ARN-Pol del virus, siempre y cuando el ARN esté unido a las nucleoproteínas. La enzima necesita usar como cebador un CAP con unos cuantos nucleótidos.
Como hemos dicho, la ARN-Pol está formada por tres proteínas:
-La PB1 tiene actividad endonucleasa y ARN-Pol. Tiene 3 regiones: dos de reconocimiento de los extremos del virus y otra que es el centro activo.
-La PB2 tiene un sitio de unión al CAP, de manera que se asocia a los mensajeros celulares.
La PB2 se une al CAP de un mensajero celular. La PB1 corta este mensajero, dejando el CAP y una pequeña secuencia de nucleótidos, que será usada como cebador. Después reconoce los extremos del fragmento de ARN vírico a transcribir, e inicia el proceso. La cola de poliA se añade al final, gracias a que la polimerasa copia varias veces seguidas un mismo fragmento de poli U. De este modo, se forma una copia de ARN (+) de cada fragmento, excepto de los segmentos 7 y 8 que se copian varias veces. Estos, además, se caracterizan porque necesitan splicing ("maduración" del ARN), que puede darse de dos modos:
-En etapas tempranas: Se elimina un gran intrón y queda un mensajero no codificante.
-En etapas tardías: Se elimina un intrón mucho más pequeño y da una de las proteínas.
-Si no se procesa, da la otra proteína.
5. Una vez que tenemos los ARNm, estos van a dos lugares: citoplasma (aquellas proteínas con señales de reconocimiento nuclear, que en cuanto se sintetizan, vuelven al núcleo) o RER (aquellas proteínas con péptido señal, que serán glicosiladas y acabarán asociadas a la membrana)
6. La replicación se lleva a cabo por el mismo mecanismo enzimático, pero con la diferencia de que no se usa un cebador con CAP, ni se añade la cola de PoliA. Esto se debe a que a lo largo del proceso de transcripción, terminan por agotarse los CAP del hospedador.
7. Una vez que tenemos todas las piezas del virus, estas han de ensamblarse. En el núcleo se asocia el ARN con las nucleoproteínas, y estas, con las proteínas de la matriz, que han englobado también a la ARN-Pol. Todo el complejo, interacciona con el complejo de poro nuclear, y sale al citoplasma. El ARN del virus no vuelve a entrar al núcleo, porque ya está asociado a la proteína de la matriz.
8. Por último, se reconocen las zonas de la membrana con proteínas víricas, y se libera el virus por gemación.
Estos viriones no son capaces de infectar a otras células ya que necesitan del procesamiento de la hemaglutinina. El procesamiento depende de las proteasas que secretan algunas de las células del epitelio intestinal o respiratorio. Cuanto más eficiente sea este proceso, más o menos virulentas serán las cepas.
Una vacuna inactiva es creada cada año para combatir las cepas de Influenza que se anticipan.[3]
Orthomyxoviridae es una familia de virus de ARN que infectan a los animales. Incluyen a los virus causantes de la gripe.
Orthomyxoviridae on viiruste sugukond.
Sugukonda Orthomyxoviridae klassifitseeritakse järgmised viiruste perekonnad:
Selles artiklis on kasutatud ingliskeelset artiklit en:Orthomyxoviridae seisuga 14.02.2014.
Ortomixobirus Orthomyxoviridae familian sailkatzen diren birusak dira, gizakia zein animaliak infektatzen dituztenak. Talde honetan gripea eragiten duten birusak daude.
"Myxo" terminoak mukia esan nahi du, eta birus hauek mukiaren bitartez transmititzen direla adierazi nahi du (arnas traktuan ugaltzen baitira).
Ortomixobirusak tamaina ertaineko birusak dira (80-120 nm). Harizpi bakarreko eta polaritate negatiboko RNA dute material genetiko gisa, oso zatikatuta (gripearen birusak, adibidez, 8 zatitan banatuta du RNA molekula). RNAren zati bakoitza kapside helikoidal batez dago babestua, baina birioia oso pleomorfikoa izaten da (gehienetan esferikoa izan arren).
Ortomixobirusek kanpoko bilgarria dute kapsideraen gainetik, bi glukoproteina dituena: hemaglutinina (HA) eta neuraminidasa (NA), ezaugarri antigenikoak dituztenak.
Aipatu den bezala, birus hauen berezko ezaugarria beren material genetikoaren antolakuntzan datza: RNA ez da zati osoa, hainbat zatitan agertzen baita banaturik (8 zatitan, zehazki, giza-gripearen birusaren kasuan). Genomaren egitura berezi horrek aldakortasun genetikoa areagotzen du, RNAren zatien arteko errekonbinaketa genetikoa errazten duelako. Esaterako, genoma desberdina duten bi andui birikok zelula bera infektatzen dutenean, haien RNA molekulak elkartu eta errekonbinatu daitezke, genoma berri bat agertuz.
Honen ondorio praktikoa egitura genetiko desberdinetako etengabeko birusen agerpena izaten da, gripearen kasuan urtero txerto berri bat sorrarazten duena.
Ortomixobirus Orthomyxoviridae familian sailkatzen diren birusak dira, gizakia zein animaliak infektatzen dituztenak. Talde honetan gripea eragiten duten birusak daude.
"Myxo" terminoak mukia esan nahi du, eta birus hauek mukiaren bitartez transmititzen direla adierazi nahi du (arnas traktuan ugaltzen baitira).
Ortomixobirusak tamaina ertaineko birusak dira (80-120 nm). Harizpi bakarreko eta polaritate negatiboko RNA dute material genetiko gisa, oso zatikatuta (gripearen birusak, adibidez, 8 zatitan banatuta du RNA molekula). RNAren zati bakoitza kapside helikoidal batez dago babestua, baina birioia oso pleomorfikoa izaten da (gehienetan esferikoa izan arren).
Ortomixobirusek kanpoko bilgarria dute kapsideraen gainetik, bi glukoproteina dituena: hemaglutinina (HA) eta neuraminidasa (NA), ezaugarri antigenikoak dituztenak.
Aipatu den bezala, birus hauen berezko ezaugarria beren material genetikoaren antolakuntzan datza: RNA ez da zati osoa, hainbat zatitan agertzen baita banaturik (8 zatitan, zehazki, giza-gripearen birusaren kasuan). Genomaren egitura berezi horrek aldakortasun genetikoa areagotzen du, RNAren zatien arteko errekonbinaketa genetikoa errazten duelako. Esaterako, genoma desberdina duten bi andui birikok zelula bera infektatzen dutenean, haien RNA molekulak elkartu eta errekonbinatu daitezke, genoma berri bat agertuz.
Honen ondorio praktikoa egitura genetiko desberdinetako etengabeko birusen agerpena izaten da, gripearen kasuan urtero txerto berri bat sorrarazten duena.
(RLQ=window.RLQ||[]).push(function(){mw.log.warn("Gadget "ErrefAurrebista" was not loaded. Please migrate it to use ResourceLoader. See u003Chttps://eu.wikipedia.org/wiki/Berezi:Gadgetaku003E.");});Ortomyksovirukset (orthomyxoviridae) on virusten heimo, johon kuuluvat influenssa A, influenssa B ja influenssa C, thogotovirus ja isavirus. Ne ovat yksijuosteisia, negatiivisäikeisiä RNA-viruksia. [1]
”Myksovirus”-nimi viittaa viruksen kykyyn tarttua musiiniin. Myksoviruksiin luettiin iso joukko erilaisia vaipallisia viruksia, jotka pystyvät kiinnittymään solun pinnan glykoproteiinireseptoreihin. Myksovirukset jaettiin sittemmin ortomyksoviruksiin ja paramyksoviruksiin rakenteellisten ja replikaatiomekanismeihin liittyvien erojen perusteella. [2]
Ortomyksovirukset (orthomyxoviridae) on virusten heimo, johon kuuluvat influenssa A, influenssa B ja influenssa C, thogotovirus ja isavirus. Ne ovat yksijuosteisia, negatiivisäikeisiä RNA-viruksia.
”Myksovirus”-nimi viittaa viruksen kykyyn tarttua musiiniin. Myksoviruksiin luettiin iso joukko erilaisia vaipallisia viruksia, jotka pystyvät kiinnittymään solun pinnan glykoproteiinireseptoreihin. Myksovirukset jaettiin sittemmin ortomyksoviruksiin ja paramyksoviruksiin rakenteellisten ja replikaatiomekanismeihin liittyvien erojen perusteella.
Orthomyxoviridés
Les Orthomyxoviridae sont une famille de virus à ARN monocaténaire de polarité négative de l'ordre des Articulavirales. Ils rassemblent notamment les quatre types de virus de la grippe. Les virus de cette famille ont un génome segmenté, c'est-à-dire divisé en segments d'acides nucléiques distincts. Dans le cas des Orthomyxoviridae, il s'agit de six à huit segments d'ARN monocaténaire de polarité négative. La longueur totale de ce génome est comprise entre 12 000 et 15 000 nucléotides.
Les quatre premiers genres regroupent les virus responsables de la grippe chez les vertébrés, y compris les oiseaux[a], les porcs, les humains, et d'autres mammifères comme les pinnipèdes (phoques, otaries et morses) alors que le genre Isavirus infecte les saumons et le genre Thogotovirus infecte vertébrés et invertébrés, comme des moustiques et des parasites de poissons.
Orthomyxoviridés
Les Orthomyxoviridae sont une famille de virus à ARN monocaténaire de polarité négative de l'ordre des Articulavirales. Ils rassemblent notamment les quatre types de virus de la grippe. Les virus de cette famille ont un génome segmenté, c'est-à-dire divisé en segments d'acides nucléiques distincts. Dans le cas des Orthomyxoviridae, il s'agit de six à huit segments d'ARN monocaténaire de polarité négative. La longueur totale de ce génome est comprise entre 12 000 et 15 000 nucléotides.
Orthomyxoviridae adalah famili virus RNA yang meliputi tujuh genera: Alphainfluenzavirus, Betainfluenzavirus, Deltainfluenzavirus, Gammainfluenzavirus, Isavirus, Quaranjavirus, dan Thogotovirus. Empat genera pertama dapat menyebabkan influenza pada vertebrata, termasuk burung (lihat juga flu burung), manusia dan mamalia lainnya. Isavirus menginfeksi ikan salmon; thogotovirus menginfeksi artropoda, seperti nyamuk.[1][2][3][4]
Orthomyxoviridae adalah famili virus RNA yang meliputi tujuh genera: Alphainfluenzavirus, Betainfluenzavirus, Deltainfluenzavirus, Gammainfluenzavirus, Isavirus, Quaranjavirus, dan Thogotovirus. Empat genera pertama dapat menyebabkan influenza pada vertebrata, termasuk burung (lihat juga flu burung), manusia dan mamalia lainnya. Isavirus menginfeksi ikan salmon; thogotovirus menginfeksi artropoda, seperti nyamuk.
Gli Orthomyxoviridae (dal greco orthos per dritto e myxa per muco)[1] sono una famiglia di virus a RNA a singolo filamento a polarità negativa ((-)ssRNA) che comprende otto generi di virus, identificati attraverso le differenze nelle loro nucleoproteine e nella proteina matrice:
I primi quattro generi rappresentano i virus che provocano l'influenza nei vertebrati come gli uccelli (in questo caso si parla di influenza aviaria), gli umani ed altri mammiferi. Gli isavirus infettano i salmoni; i thogotovirus e quaranjavirus infettano i vertebrati e gli invertebrati come le zanzare e i Copepodi.[2][3][4][5]
L'influenzavirus A provoca tutte le pandemie di influenza e infetta l'uomo, i mammiferi e gli uccelli. Influenzavirus B infetta l'essere umano e i pinnipedi. Influenzavirus C infetta l'essere umano e i suini. Influenzavirus D infetta suini e bovini.
In generale la morfologia del virus è sferica con particelle di diametro da 50 a 120 nm, oppure di forma filamentosa con diametro di 20 nm e da 200 a 300 nm di lunghezza. I virus influenzali sono dotati di una envelope che protegge ulteriormente il virus e gli permette di entrare o uscire dalla cellula.
Il capside virale è a simmetria elicoidale, ovvero costituito da una spirale di proteine chiamate nucleoproteine che si avvolge in senso antiorario. Le nucleoproteine formano una struttura ad anello presso una estremità e sono legate ad una RNA-polimerasi, hanno una lunghezza compresa tra 50 e 130 nm con un diametro che va da 9 a 15 nm e rappresentano l'"impalcatura" del capside. Ogni segmento di RNA virale è legato ad una sola nucleoproteina, formando un complesso ribonucleoproteico, la cui disposizione all'interno del virione è ignota.
La seguente struttura è relativa ai virus Influenza A, anche se gli altri ceppi di influenza hanno strutture molto simili.[6]
Il virione dell'Influenza A ha un diametro di 80–120 nm e normalmente è sferico, anche se possono esistere forme filamentose.[7] Il genoma non è costituito da una singola porzione di acido nucleico, ma contiene otto porzioni di RNA segmentato a polarità negativa (13,5 migliaia di basi in totale), che codifica 11 proteine (HA, NA, NP, M1, M2, NS1, NEP, PA, PB1, PB1-F2, PB2). [8]
Tra queste proteine virali, le meglio caratterizzate sono l'emoagglutinina e la neuraminidasi, due grandi glicoproteine che si trovano sulla superficie esterna delle particelle virali. La neuraminidasi è un enzima coinvolto nel rilascio dei virioni all'esterno delle cellule infette, mentre l'emagglutinina è una lectina che media la connessione del virus e l'ingresso del genoma virale nelle cellule target.[9] L'emagglutinina (H) e la neuraminidasi (N) sono i bersagli da colpire nei farmaci antivirali.[10]
Queste due proteine sono anche riconosciute dagli anticorpi.[11] La risposta degli anticorpi a queste proteine è utilizzata per classificare i diversi sierotipi dei virus dell'influenza A.
Tipicamente, l'influenza viene trasmessa da mammiferi infetti attraverso l'aria da colpi di tosse e starnuti, che creano particelle di aerosol contenenti il virus e dai volatili infetti attraverso i loro escrementi. L'influenza può essere trasmessa anche attraverso la saliva, le secrezioni nasali, le feci e il sangue. Le infezioni avvengono attraverso il contatto con questi fluidi corporei o con superfici contaminate. I virus possono essere infettivi per circa una settimana alla temperatura del corpo umano, più di 30 giorni a 0 °C e indefinitamente a basse temperature (come nei laghi a nordest della Siberia). Possono essere inattivati facilmente con disinfettanti e detergenti[12][13][14]
Il virus si collega ad una cellula attraverso le interazioni tra la sua glicoproteina (emoagglutinina) e l'Acido sialico sulla superficie delle cellule epiteliali nel polmone e nella gola (Fase 1 dell'infezione)[15]. La cellula importa il virus attraverso l'endocitosi. Nell'endosoma, parte della proteina emoagglutinina unisce il rivestimento del virus con la membrana del vacuolo, rilasciando le molecole virali RNA (vRNA), proteine accessorie e RNA polimerasi RNA dipendente nel citoplasma (Fase 2)[16]
Queste proteine e il vRNA formano un complesso che è trasportato nel nucleo cellulare, dove l'RNA polimerasi RNA dipendente inizia la trascrizione del vRNA complementare a polarità positiva (Fasi 3a e 3b)[17].
Il vRNA viene sia trasportato nel citoplasma e tradotto (Fase 4), o resta nel nucleo. Le nuove proteine virali che sono state sintetizzate sono secrete attraverso l'apparato del Golgi nella superficie della cellula (nel caso delle neuraminidasi e emoagglutinina, Fase 5), o trasportate indietro nel nucleo per collegare il vRNA e formare nuove particelle con genoma virale (Fase 5a). Altre proteine virali possono avere diverse azioni nella cellula ospite, tra cui la degradazione dell'mRNA cellulare e l'utilizzo dei nucleotidi rilasciati per la sintesi vRNA e l'inibizione della trascrizione dell'mRNA della cellula ospite.[18].
Il vRNA a polarità negativa che forma il genoma dei virus, l'RNA polimerasi RNA dipendente e altre proteine virali sono assemblate in un virione. Le molecole di emoaglutinina e di neuraminidase si raggruppano in un rigonfiamento nella membrana cellulare. Il vRNA e le proteine virali lasciano il nucleo ed entrano in questo rigonfiamento (Fase 6). Il virus maturo germoglia dalla cellula in una sfera costituita dalla membrana fosfolipidica dell'ospite, acquisendo attraverso di essa l'emaglutinina e la neuraminidase (Fase 7)[19]. Dopo il rilascio del nuovo virus influenzale, la cellula ospite muore.
A causa dell'assenza degli enzimi di controllo dell'RNA, l'RNA polimerasi RNA dipendente genera un errore di inserimento circa ogni 10 migliaia di nucleotidi, che è la lunghezza approssimativa del vRNA dell'influenza. Quindi, quasi ogni nuovo virus dell'influenza che viene creato contiene una mutazione nel suo genoma.[20]. La separazione del genoma in otto diverse porzioni di vRNA permette il mescolamento o il riassortimento dei geni se più di una varietà di virus infetta la stessa cellula. L'alterazione che ne risulta nei segmenti del genoma viene impacchettata nella progenie virale e conferisce un nuovo comportamento, a volte anche la capacità di infettare nuove specie ospite o superare l'immunità dell'ospite al vecchio genoma (nel qual caso viene chiamato spostamento antigenico)[11].
Esistono tre generi di virus influenzali dell'uomo: Influenzavirus A, Influenzavirus B, Influenzavirus C. Ogni genere include solo una specie o tipo: virus dell'influenza A, virus dell'influenza B e virus dell'influenza C. I virus A e C infettano diverse specie, mentre il virus B infetta quasi esclusivamente l'uomo.[21][22]
I virus della specie Influenza A sono ulteriormente classificati in base alle proteine superficiali HA (o H) e NA (o N). Sono stati identificati diciotto sottotipi (o sierotipi) H e undici sottotipi N del virus Influenza A.[23] i ceppi influenzali che vengono isolati sono identificati tramite una nomenclatura standard attraverso la quale viene indicato il tipo di virus, la localizzazione geografica dove è stato isolato per la prima volta, il numero sequenziale di isolamento, l'anno di isolamento e i sottotipi HA e NA.[24][25]
Alcuni esempi di questa nomenclatura sono:
I virus di tipo A sono i più virulenti patogeni umani tra i tre tipi di influenza e provocano le patologie più gravi. I sierotipi che sono stati confermati nell'uomo, ordinati dal numero di decessi sono:
I virus Influenza B è quasi esclusivamente un patogeno umano ed è meno comune dell'Influenza A. L'unico animale conosciuto, oltre all'uomo, ad essere colpito dall'influenza B sono le foche[27]. Questo tipo di influenza muta ad un tasso da 2 a 3 volte inferiore del tipo A[28] e quindi ha una diversità genetica inferiore, con solo un sierotipo[21] Come risultato di questa scarsa diversità antigenica, normalmente si acquisisce un certo grado di immunità all'influenza B. Tuttavia il virus muta in modo tale da impedire un'immunità permanente[29]. Questo ridotto tasso di cambiamento negli antigeni, combinato con la scarsa gamma di ospiti (che impedisce lo spostamento antigenico), previene la formazione di pandemie di influenza B[30]
L'influenza C infetta l'uomo e i suini e può provocare malattie gravi e localmente epidemiche[31] Tuttavia, l'influenza C è meno comune rispetto agli altri tipi e normalmente provoca disturbi non troppo gravi nei bambini.[32][33]
Nella tassonomia filogenetica i virus RNA includono i virus con RNA a singola elica, filamento negativo (virus ssRNA), che include l'ordine Mononegavirales e la famiglia "Orthomyxoviridae" (tra le altre). Le specie degli Orthomyxoviridae associate a ciascun genere sono illustrate nella tabella seguente:
Gli Orthomyxoviridae (dal greco orthos per dritto e myxa per muco) sono una famiglia di virus a RNA a singolo filamento a polarità negativa ((-)ssRNA) che comprende otto generi di virus, identificati attraverso le differenze nelle loro nucleoproteine e nella proteina matrice:
Influenzavirus A Influenzavirus B Influenzavirus C Influenzavirus D Isavirus Quaranjavirus ThogotovirusI primi quattro generi rappresentano i virus che provocano l'influenza nei vertebrati come gli uccelli (in questo caso si parla di influenza aviaria), gli umani ed altri mammiferi. Gli isavirus infettano i salmoni; i thogotovirus e quaranjavirus infettano i vertebrati e gli invertebrati come le zanzare e i Copepodi.
L'influenzavirus A provoca tutte le pandemie di influenza e infetta l'uomo, i mammiferi e gli uccelli. Influenzavirus B infetta l'essere umano e i pinnipedi. Influenzavirus C infetta l'essere umano e i suini. Influenzavirus D infetta suini e bovini.
Orthomyxoviridae sunt familia virorum cum septem generis, quae ARN pluripartita, singulam polaritatem, negativa fila ostendunt. Segmentatione genetica causa orthomyxoviridae flexiblissimae quorumvis locorum sunt. Orbis terrarum scilicet systematum sanitariorum maximum momentum vira influentiae producunt.
Orthomyxoviridae de verbis Graecis ὀρθός, recte, et μύξα, mucus, derivatur.
Genera subdivisiones septem sunt Influenzavira A, B, C, D, Isavira, Quaraniavira, Thogotovira.
Omnia influenzavira infarctum cordis efficere possunt[1].
Adhuc singularis species nota est: influenza A virus. Nonnulli serotypi explorati sunt, per exemplum H1N1, H5N1, et alii. Influenzavirus A hominum, porcorum, avium, equorum, chiropterorum contagiosa sunt.
Adhuc singularis species nota est: influenza B virus, duo serotypi momentum habent: Victoria et Iamagata[2]. Influenzavira B hominum, phocidarum contagiosa sunt. Influenza B vira quibusdam annis damnum maximum confecierunt[3].
Adhuc singularis species nota est: influenza C virus. Influenzavira C hominum, porcorum, canum contagiosa sunt.
Adhuc singularis species nota est: influenza D virus. Influenzavira D porcorum, boum contagiosa sunt.
Infectiosus salmonis anaemia virus singulariter in hoc genere invenitur et solum salmonis contagiosum est.
Generi huic duo species sunt: Johnston Atoll virus et Quaranfil virus.
Quoque generi huic duo species insunt: Dhoris virus et Thogoto virus. Vira ixodidorum, culicum, mammaliarum contagiosa sunt. Nimirum transmissor Thogotovirorum ixodida quoque apparere possunt[4].
Orthomyxoviridae sunt familia virorum cum septem generis, quae ARN pluripartita, singulam polaritatem, negativa fila ostendunt. Segmentatione genetica causa orthomyxoviridae flexiblissimae quorumvis locorum sunt. Orbis terrarum scilicet systematum sanitariorum maximum momentum vira influentiae producunt.
Ortomiksovirusai (lot. Orthomyxoviridae) – RNR grupės apvalkalėtųjų virusų šeima. Virusų transkripcija vyksta paukščių ir žinduolių ląstelių branduoliuose ir citoplazmoje, plinta be pernešėjo, pažeidžia kvėpavimo organus. Tipiški atstovai – gripo virusai.[1]
Ortomiksovirusų šeimą sudaro 6 virusų gentys, tarp kurių pirmosioms trims gentims priklauso tipinės virusų rūšys, sukeliančios stuburinių organizmų gripą, įskaitant paukščius (žr. paukščių gripas), žmones ir kitus žinduolius:
Ortomiksovirusai (lot. Orthomyxoviridae) – RNR grupės apvalkalėtųjų virusų šeima. Virusų transkripcija vyksta paukščių ir žinduolių ląstelių branduoliuose ir citoplazmoje, plinta be pernešėjo, pažeidžia kvėpavimo organus. Tipiški atstovai – gripo virusai.
Orthomyxovirus Pengelasan virus Group: Kumpulan V ((−)ssRNA) Alam: Virus Filum: Negarnaviricota Kelas: Insthoviricetes Order: Articulavirales Keluarga: Orthomyxoviridae Genera
Orthomyxoviridae (daripada ὀρθός, orthós dan μύξα, mýxa, "lurus" dan "mukus" masing-masing dalam bahasa Yunani)[1] ialah sebuah keluaga virus RNA. Keluarga ini memiliki tujuh genus, yakni empat virus influenza, Virus influenza A, Virus influenza B, Virus influenza C serta Virus influenza D, Isavirus, Quaranjavirus dan Thogotovirus.
Genus serta serotip virus-virus di bawah Orthomyxoviridae adalah seperti yang berikut:
Genus, spesis dan serotip Orthomyxoviridae Genus Spesis Serotip atau subjenis Hos Virus influenza A Virus influenza A H1N1, H1N2, H2N2, H3N1, H3N2, H3N8, H5N1, H5N2, H5N3, H5N8, H5N9, H7N1, H7N2, H7N3, H7N4, H7N7, H7N9, H9N2, H10N7 Manusia, khinzir, burung, kuda, kelwar Influenza virus B Virus influenza B Victoria, Yamagata[2] Manusia, anjing laut Influenza virus C Virus influenza C Manusia, khinzir, anjing Influenza virus D Virus influenza D Khinzir, lembu Isavirus Virus anemia salmon berjangkit Salmon Atlantik Thogotovirus Thogotovirus Sengkenit, nyamuk, mamalia (termasuk manusia) Virus Dhori Virus Batken, Virus Bourbon, Virus Jos Quaranjavirus[3] Virus Quaranfil, Virus Atol JohnstonOrthomyxoviridae (daripada ὀρθός, orthós dan μύξα, mýxa, "lurus" dan "mukus" masing-masing dalam bahasa Yunani) ialah sebuah keluaga virus RNA. Keluarga ini memiliki tujuh genus, yakni empat virus influenza, Virus influenza A, Virus influenza B, Virus influenza C serta Virus influenza D, Isavirus, Quaranjavirus dan Thogotovirus.
Met het orthomyxovirus (van de Griekse woorden orthos (recht) en myxa (slijm)) wordt een bepaald type virussen met inbegrip van die welke influenza veroorzaken bedoeld.
De virussen zijn een familie van RNA-virussen met vijf geslachten: influenzavirus A, influenzavirus B, influenzavirus C, isavirus en thogotovirus. Een zesde virus is in 2009 ontdekt.[1]
Influensavirus er en gruppe virus som forårsaker influensa hos mennesker.
Ortomyksowirusy (łac. Ortomyxoviridae) – rodzina wirusów, charakteryzujących się następującymi cechami:
Systematyka ortomyksowirusów obejmuje rodzaje[1]:
Pierwsze trzy rodzaje to wirusy grypy – cechują się one wybitną zmiennością antygenową, co utrudnia stworzenie skutecznej szczepionki. Togotowirus nie ma większego znaczenia medycznego.
Ortomyksowirusy (łac. Ortomyxoviridae) – rodzina wirusów, charakteryzujących się następującymi cechami:
Symetria: helikalna, ale kształt całego wirionu, ze względu na obecność otoczki, może być bardzo zmienny Osłonka lipidowa: istnieje i zawiera liczne wypustki (są to główne antygeny tych wirusów), pod osłonką znajduje się warstwa białka M, która otacza helikalnie nawinięty na szkielet białkowy genom (tzw. rybonukleoproteina). W przypadku togotowirusa w obrębie jednego wirionu (otoczki) może znajdować się więcej niż jeden nukleokapsyd. Kwas nukleinowy: segmentowany ssRNA(-) Replikacja: zachodzi w jądrze zakażonej komórki Peptydy i białka: ich ilość zależna jest od liczby segmentów RNA – każdy segment koduje jedno białko Wielkość: 80-120 nm (togotowirus nawet do 300 nm) Gospodarz: kręgowce Cechy dodatkowe: najbardziej charakterystyczny jest bardzo silny pleomorfizm i silna zmienność antygenowa.Systematyka ortomyksowirusów obejmuje rodzaje:
Rodzina: Orthomyxoviridae (Ortomyksowirusy) Rodzaj: Influenzavirus A (FLUAV), zwyczajowo wirus grypy typu A – wywołuje pandemie, w tym hiszpankę, grypę ptaków i grypę świń Gatunek: Influenza A virus Rodzaj: Influenzavirus B (FLUBV), zwyczajowo wirus grypy typu B – atakuje ludzi i foki Gatunek: Influenza B virus Rodzaj: Influenzavirus C (FLUCV), zwyczajowo wirus grypy typu C – atakuje ludzi i świnie Gatunek: Influenza C virus Rodzaj: Thogotovirus – przenoszony przez kleszcze, wywołuje gorączkę i zapalenie mózgu Gatunek: Thogoto virus – gatunek typowy Gatunek: Dhori virus Rodzaj: Isavirus – atakuje łososie Gatunek: Infectious salmon anemia virusPierwsze trzy rodzaje to wirusy grypy – cechują się one wybitną zmiennością antygenową, co utrudnia stworzenie skutecznej szczepionki. Togotowirus nie ma większego znaczenia medycznego.
Orthomyxoviridae[1] é uma família de vírus RNA. Inclui sete gêneros: Influenzavirus A, Influenzavirus B, Influenzavirus C, Influenzavirus D, Isavirus, Thogotovirus e Quaranjavirus. Os primeiros quatro gêneros contêm vírus que causam gripe em vertebrados, incluindo aves (ver também: gripe aviária), humanos e outros mamíferos. O Isavirus infecta o salmão; os thogotovírus são arbovírus, infectando vertebrados e invertebrados, como carrapatos e mosquitos.[2][3][4]
Os quatro gêneros do vírus Influenza, identificados por diferenças antigênicas em suas nucleoproteínas e proteína de matriz, infectam os vertebrados da seguinte maneira:
Orthomyxoviridae é uma família de vírus RNA. Inclui sete gêneros: Influenzavirus A, Influenzavirus B, Influenzavirus C, Influenzavirus D, Isavirus, Thogotovirus e Quaranjavirus. Os primeiros quatro gêneros contêm vírus que causam gripe em vertebrados, incluindo aves (ver também: gripe aviária), humanos e outros mamíferos. O Isavirus infecta o salmão; os thogotovírus são arbovírus, infectando vertebrados e invertebrados, como carrapatos e mosquitos.
Os quatro gêneros do vírus Influenza, identificados por diferenças antigênicas em suas nucleoproteínas e proteína de matriz, infectam os vertebrados da seguinte maneira:
Influenzavirus A infecta humanos, outros mamíferos e aves e causa todas as pandemias de gripe Influenzavirus B infecta humanos e focas Influenzavirus C infecta humanos, porcos e cães. Influenzavirus D infecta suínos e bovinosOrthomyxoviridae (Ortomyxovirus) är en RNA-virusfamilj med fem olika genera. Influensa A, Influensa B och Influensa C orsakar Influensa i vertebrater där till exempel däggdjur och fåglar ingår. Isavirus infekterar lax och Thogotvirus infekterar vertebrater och invertebrater, i den senare gruppen ingår till exempel insekter. RNA-strängen är omgiven av en kapsid vilken i sin tur omges av ett hölje med två olika glykoproteiner, hemagglutinin (H) och neuroadminisad (N). Dessa kombineras på olika sätt och bildar de olika influensavarianterna (H1N1, H5N1, H5N8, H10N1 m fl). Alla tre influensagenera kan infektera människor men det är Influensa A som skapar pandemier. Det är bara vissa H och N varianter som kan infektera människa.
Det finns tre olika typer av influensavirus:
Orthomyxoviridae (Ortomyxovirus) är en RNA-virusfamilj med fem olika genera. Influensa A, Influensa B och Influensa C orsakar Influensa i vertebrater där till exempel däggdjur och fåglar ingår. Isavirus infekterar lax och Thogotvirus infekterar vertebrater och invertebrater, i den senare gruppen ingår till exempel insekter. RNA-strängen är omgiven av en kapsid vilken i sin tur omges av ett hölje med två olika glykoproteiner, hemagglutinin (H) och neuroadminisad (N). Dessa kombineras på olika sätt och bildar de olika influensavarianterna (H1N1, H5N1, H5N8, H10N1 m fl). Alla tre influensagenera kan infektera människor men det är Influensa A som skapar pandemier. Det är bara vissa H och N varianter som kan infektera människa.
Det finns tre olika typer av influensavirus:
Influensavirus typ A Influensavirus typ B Influensavirus typ CВключає 5 родів: віруси грипу А, В, С, тоготовіруси та ісавіруси. Віріони мають здебільшого сферичну форму, але зустрічаються ниткоподібні форми. Діаметр частинок 80 – 120 нм, молекулярна маса близько 250 мегадальтон, плавуча густина 1,19 г/см³ та коефіцієнт седиментації 700-800S. Віріон складається з внутрішньої нуклеопротеїдної спіралі діаметром 8 – 9 нм та оболонки, що включає два шари ліпідів, у які інтегровані молекули гемаглютиніну та нейрамінідази. Білки на поверхні ліпідного шару утворюють ворсинки довжиною 10 - 14 нм та діаметром до 4 нм. Віруси не мають високої резистентності до фізичних та хімічних факторів, зокрема гинуть при температурі 56°C, чутливі до ефіру та дезоксихолату. Містять кілька антигенів: нуклеокапсидний (Np), мембранний (М) антигени знаходяться в середині віріону, є також поверхневі антигени - гемаглютинін (Н) та нейрамінідаза (N). Геном представлений одноланцюговою лінійною РНК, що складається з кількох фрагментів, загальною масою до 4 мегадальтон.
Віруси аглютинують еритроцити людини, морських свинок, курей та ін. Містять фермент, що руйнує мукопротеїдні рецептори клітини. Гемоліз не властивий. До цієї групи входять численні віруси, що викликають респіраторні та генералізовані захворювання людини, ссавців та птиці. Зокрема це віруси, що викликають грип людини, класичну чуму птахів, грип качок, ластівок, свиней, коней.
Діаметр вірусної частинки 80-120 нм. Віріон має сферичну форму. В центрі віріону розміщений нуклеокапсид, який має спіральний тип симетрії. Геном представлений спіральною одноланцюговою сегментованою мінус РНК. Капсид складається з білка нуклеопротеїну, а також білків полімеразного комплексу. Сегментована РНК здатна до генетичних рекомбінацій, а також до зміни антигенної структури. Нуклеокапсид оточений шаром матриксних і мембранних білків, які беруть участь в збирані віріону. Поверх цих структур знаходиться ліпопротеїнова оболонка, яка має клітинне походження. Вона має на своїй поверхні вирости – шипики, довжиною близько 10 нм.
При температурі 55°С ВГП інактивується за годину, при 60°С - за 10 хв., при 65-70°С - за 2-5 хв. При низьких температурах і в ліофілізованому стані (при -30°С в запаяних ампулах у темному місці) зберігається до 2 років. У присутності MgCl2 вірус інактивується швидше, a MgSO4 стабілізує його. ГА і інфекційність вірусу зберігаються при - 60°С кілька років, а при 4°С - кілька тижнів. ГА властивості більш стабільні, чим інфекційні. Інфекційність вірусу втрачається при обробці його формальдегідом, детергентами, оксидованими агентами, слабкими кислотами, додецилсульфатом, гідроксил аміном, іонами амонію.
Віруси не мають високої резистентності до фізичних та хімічних факторів, зокрема гинуть при температурі 56°C, чутливі до ефіру та дезоксихолату.
Геном представлений сегментованою одноланцюговою (-)РНК. Розмір геному 13.5 Kb. Стратегія реплікації: Ядерна
Як правило, грип передається від інфікованих ссавців аерогенним механізмом, повітряно-краплинним шляхом, та від інфікованих птахів через їх послід. Грип може передаватися через слину, виділення з носа, кал і кров. Інфікування відбувається через контакт з цими біологічними рідинами або з забрудненими поверхнями. Віруси грипу можуть залишатися заразними протягом одного тижня при температурі тіла людини, більше 30 днів при 0 °C ( 32 °F ), і на невизначений термін при дуже низьких температурах (наприклад, в озерах північно-східного Сибіру). Вони можуть бути легко інактивовані дезинфікуючими і миючими засобами.
Віруси приєднуються до клітини через взаємодію між його гемаглютинуючим глікопротеїном і поліцукрами сіалової кислоти на поверхні епітеліальних клітин у легенях і глотці (крок 1). Всередину клітини вірус проникає шляхом ендоцитозу. Далі частина білка гемаглютиніну розрізає мембрану вакуолі навколо вірусної часточки, звільняючи вірусну РНК ( вРНК ), допоміжні білки і РНК-залежну РНК-полімеразу в цитоплазму (крок 2). Ці білки і вРНК утворюють комплекс, який транспортується в ядро клітини, де РНК-залежна РНК-полімераза починає транскрибувати комплементарну + РНК (кроки 3a і б). Ця РНК або експортується в цитоплазму і транслюється (крок 4), або залишається в ядрі. Заново синтезовані вірусні білки або переносяться через апарат Гольджі на поверхню клітини (наприклад нейрамінідази і гемаглютиніни, крок 5b), або ж повертаються назад в ядро, щоб зв'язати вРНК і сформувати нові вірусні частки (крок 5а). Інші вірусні білки мають численні методи впливу на клітину-хазяїна, включаючи розщеплення клітинної мРНК з подальшим використанням утворених нуклеотидів для синтезу власної вРНК, а також інгібування трансляції власної мРНК клітини-хазяїна.
- вРНК, які утворюють геноми майбутніх вірусів, РНК-залежна РНК транскриптаза та інші вірусні білки збираються у віріони. Гемаглютиніни і нейрамінідаза утворюють молекулярні кластери в клітинній мембрані. вРНК та вірусні білки, покинувши ядро, входять в цей мембранний виступ (крок 6). Дозрілі віріони покидають клітину в суперкапсиді з фосфоліпідної мембрани клітини-господаря, набуваючи гемаглютинуючих і нейрамінідазних властивостей (крок 7). Зрілі віруси відокремлюються від клітини-хазяїна, як тільки їх нейрамінідаза розщеплює залишки сіалової кислоти. Після випуску новоствореного вірусу, клітина-господар гине.
Оскільки РНК-коректуючі ферменти відсутні, РНК-залежна РНК-транскриптаза робить помилку вставки нуклеотиду приблизно через кожні 10 тис. нуклеотидів, що приблизно відповідає довжині геному вірусу грипу. Таким чином, майже кожен заново створений вірус грипу буде містити мутацію в своєму геномі. Поділ геному на вісім окремих сегментів вРНК дозволяє відбуватися перемішуванню (перестановці) генів, якщо одну і ту ж клітину заразив не один вірус (суперінфекція). У результаті зміни в сегментах геному, нове покоління вірусу виявляє нову поведінку, іноді отримуючи здатність інфікувати нові види або подолати захисний імунітет населення створений до свого старого генома (в цьому випадку він називається антигенним зсувом).
7 білків. P1, P2, P3 названі за першою буквою слова «полімераза». P1- РНК-полімераза, P3 має властивості ендонуклеази. P1, P3 – основні властивості, P2 – кислі. Входять до складу нуклеокапсиду і утворюють комплекс, зв'язаний з геномом.
Гемаглютинін - один з найбільших білків у віріоні, на його частку припадає 25-35% білків. Він відповідає за прикріплення вірусної частки до клітини хазяїна й бере участь у початковій стадії інфекції. Саме проти ГА спрямовані антитіла, що нейтралізують інфекційність вірусу грипу. Через важливу роль ГА в розвитку інфекції, структура цього білка привернула увагу багатьох дослідників, і до дійсного часу в структурному відношенні він найбільш вивчений.
Нейрамінідаза - інший поверхневий антиген вірусу грипу. Цей фермент у складі вірусу грипу відомий з 1942 р., однак функціональна роль його в репродукції вірусу дотепер повною мірою не з'ясована. Передбачається, що головна функція нейрамінідази - запобігання агрегації вірусних часток на поверхні клітин. Також відомо, що нейрамінідаза відіграє важливу роль в імунології і патогенезі захворювання.
Нуклеопротеїд – основний внутрішній білок вірусу, що формує субодиниці капсиду. Він зв'язаний з геномом протягом всього періоду репродукції вірусу, але не перешкоджає експресії генома. Є типоспецифічним антигеном.
Матриксний білок – низькомолекулярний білок у складі віріона, локалізований на внутрішній поверхні ліпопротеїдної мембрани вірусу. Так само як і НП є типоспецифічним антигеном.
Білки NS1 і NS2 – неструктурні вірусні білки, кодуються однією восьмою всього генома, мають унікальні послідовності амінокислот завдяки трансляції зі зсувом рамки.
Ліпіди знаходяться в оболонці віріону (18-37%). Це модифіковані ліпіди клітини, вилучені з її плазматичної мембрани.
Вуглеводи знаходяться у складі глікопротеїдів.
В даний час віруси грипу А птахів на підставі їх поверхневих АГ - ГА (Н) і нейрамінідази (N) - розділені на 13 по Н-АГ і 9 варіантів по N-AT. Крім 13 підтипів ГА, усі штами ВГП містять у своїй структурі НА чи пташиного, чи людського, чи кінського походження. Власне пташиного НА відомо 6 типів.
Дикі водоплавні птахи - резервуар різних підтипів ГА (Н) і нейрамінідази (N) вірусу грипу. У цих популяціях зараження часто відбуваються там, де водоплавні птахи, свині і люди живуть близько і за рахунок генної реасортації можуть утворюватися нові більш патогенні штами вірусу грипу.
Віруси грипу індукують в організмі хворої і перехворілої птиці AT, що володіють анти-ГА - і КЗ властивостями. В експериментальних умовах специфічні AT можна одержати на кроликах, пацюках і морських свинках як на нативний вірулентний вірус, так і на інактивований бета-пропіолактоном.
Вірус типу А може інфікувати людей, птахів, свиней, коней і китів. Віруси типу В виділяються винятково в людей, вірус типу С – у людей та свиней.
Шляхи передачі в природі: фекально-оральний (пташиний грип), повітряно-крапельний (іспанка). Вірус надзвичайно поширений, паразитує на хребетних тваринах, які є переносниками та поширювачами збудників, а також «живими лабораторіями» з виготовлення нових штамів – при змішаному зараженні віруси можуть обмінюватися сегментами геному.
Тропізм: Миготливий епітелій верхніх дихальних шляхів.
Географічне розповсюдження зафіксовано по всій земній кулі.
Патогенез: Вірус потрапляє в організм через дихальні шляхи, якщо проникне прямо в дихальні шляхи це може призвести до первинної гострої пневмонії. Первина репродукція вірусу проходить в клітинах дихальних шляхів. Інфіковані клітини починають виробляти інтерферон. Відбувається десквамація клітин поверхневого шару. Через пошкоджені клітини потрапляє в кров і викликає віремію. Всмоктування продуктів розпаду клітин викликає токсичну і сенсибілізуючу дію. Вірус активує систему протеолізу і викликає пошкодження ендотелію капілярів. Це в свою чергу викликає геморагії і порушення гемодинаміки з розладами мікроциркуляції. Також розвивається транзисторний імунодефіцит, що призводить до вторинної бактеріальної інфекції.
У людини хворої на грип проявляються симптоми гострого респіраторного захворювання, яке пов'язане з руйнуванням клітин миготливого епітелію дихальних шляхів. Відбувається відшарування епітелію, як наслідок – бронхіт, можуть бути тромбози та некрози судин. Небезпека грипу полягає у великій вірогідності післягрипозних ускладнень.
Як правило перебіг хвороби доброякісний і потребує симптоматичного лікування (застосовують жарознижуючі, судинозвужуючі, антигістамінні препарати, вітаміни, імуномодулятори, ангіопротектори, інгібітори протеолізу).
Для профілактики приймають протиепідемічні заходи, які обмежують поширення вірусів грипу аерогенно і контактно (ізоляція хворих, карантин в дитячих закладах і лікувальних закладах).
Orthomyxoviridae
Группа по БалтиморуОртомиксовирусы[2] (лат. Orthomyxoviridae) — семейство РНК-содержащих вирусов из порядка Articulavirales. Диаметр вирусных частиц 80—120 нм. Нуклеокапсид спиральный, заключён в липопротеидную оболочку. Содержат 7—8 фрагментов одноцепочечной линейной РНК (общая мол. м. 5 000 000). Вирионная РНК неинфекционна и комплементарна информационной РНК. Размножаются в ядре и цитоплазме клеток птиц, млекопитающих; созревают путём почкования на плазматической мембране клеток. Распространяются без переносчика. Поражают дыхательные органы. Типичные представители — вирусы гриппа (Influenza A—D virus).
На январь 2019 года к семейству относят 9 видов в 7 родах[3]:
Ортомиксовирусы (лат. Orthomyxoviridae) — семейство РНК-содержащих вирусов из порядка Articulavirales. Диаметр вирусных частиц 80—120 нм. Нуклеокапсид спиральный, заключён в липопротеидную оболочку. Содержат 7—8 фрагментов одноцепочечной линейной РНК (общая мол. м. 5 000 000). Вирионная РНК неинфекционна и комплементарна информационной РНК. Размножаются в ядре и цитоплазме клеток птиц, млекопитающих; созревают путём почкования на плазматической мембране клеток. Распространяются без переносчика. Поражают дыхательные органы. Типичные представители — вирусы гриппа (Influenza A—D virus).
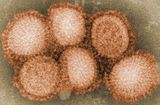 類型
類型 正黏液病毒科(Orthomyxoviridae,希臘文Orthos有「正確,直」之意;myxo有「粘液」之意[1])屬於負鏈RNA病毒,共有六個屬,此科的病毒可感染脊椎動物。造成流行性感冒的病毒正是正黏液病毒科的一員,屬於負鏈RNA病毒。正黏液病毒科可分為四屬病毒,三屬為流行性感冒病毒,分為A型、B型及C型流行性感冒病毒,以及传染性鲑鱼贫血病毒屬和托高土病毒屬。流行性感冒病毒可以感染人、馬、豬及禽類(參見禽流感),其中A型流行性感冒病毒可以感染人、豬、馬及鳥類,而B型流行性感冒,僅可感染人類。C型流行性感冒病毒僅感染人、豬,造成之病害較少。 A型及B型流行性感冒病毒之感染力非常強,傳播速度非常快,可以稱之為最重要的疾病之一,感染範圍遍及世界各大洲。水禽類,尤其是候鳥,為各不同血清型病毒之帶原者及病毒重組之動物。传染性鲑鱼贫血病毒則鮭魚,托高土病毒則可以感染脊椎動物及非脊椎動物,如蚊子和海蝨[2][3][4]。
本科中有三屬為流感病毒,其分類方法依照其表面NP及M抗原相異性:
按系統分類學,此科屬於RNA病毒的ssRNA(-),其關聯物種及血清型如下表所示:
正黏液病毒屬別、種及血清型 屬 種(*為模式種) 血清型或亞型 寄主 A型流感病毒 A型流感病毒* H1N1、H1N2、H2N2、H3N1、H3N2、H3N8流行性感冒病毒係依其基質蛋白(matrix protein)及內部核糖核蛋白(ribonucleoprotein;可溶性抗原)之抗原特性而分為A、B及C三種,外層蛋白包括血球凝集素(hemagglutinin,HA)及神經氨酸酶(neuraminidase, NA)兩種,也為組成流行性感冒的抗原成份。流行性感冒病毒依其血球凝集素及神經胺酸酶之抗原性不同可再分為數種亞型,現今已知血球凝集素已有十六種(H1-H16),神經胺酸酶則有九種。C型流行性感冒之化學性及結構性與 A 及 B 型流行性感冒皆十分相似,但它缺少神經胺酸酶,只有血球凝集素酶一種,在病毒表面上具有一特殊的接受器(receptor),使病毒表面呈蜂窩狀,而 A 型及 B 型流行性感冒很少呈蜂窩狀。
流行性感冒病毒在電子顯微鏡下有絲狀及多形性二種,但大部份為多形性,負染色時常為 70 - 120 nm 之圓形顆粒。新分離的流行性感冒病毒顆粒,其大小及形狀皆不一致,絲狀者經常可長達幾百奈米(nanometer),甚至長達 4,000 nm,有時可以見到一些奇形怪狀者,流行性感冒病毒與其它病毒不同的是它的整個病毒表面有約略等距間隔的突起,一種為長約 10 - 14 nm,寬約 4 nm ,橫切面為三角形,含有血球凝集素的桿狀突起,另一為長約 9 nm,寬約 5 nm,含有神經胺酸酶的圓柱狀突起。
在針狀突起層下面為一包圍基質蛋白及核酸蛋白之脂質被膜, A型流行性感冒病毒之核蛋白衣(nucleocapsid)含有與病毒 RNA 相連的 RNA 聚合酶, B 型及 C 型流行性感冒病毒之核蛋白衣亦可能有相同的結構。病毒封套(envelope)厚度為 6 至 8 nm,當封套破裂或染色液滲入時,可能會看到核酸呈摺疊狀平行的條狀結構。以清潔劑處理也可能會得到大小不等的極度卷曲形狀的核蛋白衣。
C 型流行性感冒病毒外型與 A或B型結構大致相同,不容易區分,有時可見病毒表面有六角型的結構,通常在電子顯微鏡下,同一視野中可能會同時見到這兩種不同形狀的病毒顆粒。
血球凝集素可附著於不同動物的紅血球,而使紅血球凝集,而神經胺酸酶與病毒離開寄主細胞有關,神經胺酸酶會分解膜上的神經胺酸,而使病毒由寄主細胞脫離感染其他細胞。 C 型流行性感冒病毒也含有可破壞流行性感冒接受器的酶,但它與神經胺酸酶不同。
Viruses of this family contain 6 to 8 segments of linear negative-sense single stranded RNA.[7]
The total genome length is 12000–15000 nucleotides (nt). The largest segment 2300–2500 nt; of second largest 2300–2500 nt; of third 2200–2300 nt; of fourth 1700–1800 nt; of fifth 1500–1600 nt; of sixth 1400–1500 nt; of seventh 1000–1100 nt; of eighth 800–900 nt. Genome sequence has terminal repeated sequences; repeated at both ends. Terminal repeats at the 5'-end 12–13 nucleotides long. Nucleotide sequences of 3'-terminus identical; the same in genera of same family; most on RNA (segments), or on all RNA species. Terminal repeats at the 3'-end 9–11 nucleotides long. Encapsidated nucleic acid is solely genomic. Each virion may contain defective interfering copies.
The following applies for Influenza A viruses, although other influenza strains are very similar in structure:[8]
The influenza A virus particle or virion is 80–120 nm in diameter and usually roughly spherical, although filamentous forms can occur.[9] Unusually for a virus, the influenza A genome is not a single piece of nucleic acid; instead, it contains eight pieces of segmented negative-sense RNA (13.5 kilobases total), which encode 11 proteins (HA, NA, NP, M1, M2, NS1, NEP, PA, PB1, PB1-F2, PB2).[10] The best-characterised of these viral proteins are hemagglutinin and neuraminidase, two large glycoproteins found on the outside of the viral particles. Neuraminidase is an enzyme involved in the release of progeny virus from infected cells, by cleaving sugars that bind the mature viral particles. By contrast, hemagglutinin is a lectin that mediates binding of the virus to target cells and entry of the viral genome into the target cell.[11] The hemagglutinin (H) and neuraminidase (N) proteins are targets for antiviral drugs.[12] These proteins are also recognised by antibodies, i.e. they are antigens.[13] The responses of antibodies to these proteins are used to classify the different serotypes of influenza A viruses, hence the H and N in H5N1.
Typically, influenza is transmitted from infected mammals through the air by coughs or sneezes, creating aerosols containing the virus, and from infected birds through their droppings. Influenza can also be transmitted by saliva, nasal secretions, feces and blood. Infections occur through contact with these bodily fluids or with contaminated surfaces. Flu viruses can remain infectious for about one week at human body temperature, over 30 days at 0 °C(32 °F), and indefinitely at very low temperatures (such as lakes in northeast Siberia). They can be inactivated easily by disinfectants and detergents.[14][15][16]
The viruses bind to a cell through interactions between its hemagglutinin glycoprotein and sialic acid sugars on the surfaces of epithelial cells in the lung and throat (Stage 1 in infection figure).[17] The cell imports the virus by endocytosis. In the acidic endosome, part of the haemagglutinin protein fuses the viral envelope with the vacuole's membrane, releasing the viral RNA (vRNA) molecules, accessory proteins and RNA-dependent RNA polymerase into the cytoplasm (Stage 2).[18] These proteins and vRNA form a complex that is transported into the cell nucleus, where the RNA-dependent RNA polymerase begins transcribing complementary positive-sense cRNA (Steps 3a and b).[19] The cRNA is either exported into the cytoplasm and translated (step 4), or remains in the nucleus. Newly synthesised viral proteins are either secreted through the Golgi apparatus onto the cell surface (in the case of neuraminidase and hemagglutinin, step 5b) or transported back into the nucleus to bind vRNA and form new viral genome particles (step 5a). Other viral proteins have multiple actions in the host cell, including degrading cellular mRNA and using the released nucleotides for vRNA synthesis and also inhibiting translation of host-cell mRNAs.[20]
Negative-sense vRNAs that form the genomes of future viruses, RNA-dependent RNA transcriptase, and other viral proteins are assembled into a virion. Hemagglutinin and neuraminidase molecules cluster into a bulge in the cell membrane. The vRNA and viral core proteins leave the nucleus and enter this membrane protrusion (step 6). The mature virus buds off from the cell in a sphere of host phospholipid membrane, acquiring hemagglutinin and neuraminidase with this membrane coat (step 7).[21] As before, the viruses adhere to the cell through hemagglutinin; the mature viruses detach once their neuraminidase has cleaved sialic acid residues from the host cell.[17] After the release of new influenza virus, the host cell dies.
Orthomyxoviridae viruses are one of the only RNA viruses that replicate in the nucleus. This is because the machinery of orthomyxo viruses cannot make their own mRNAs. They use cellular RNAs as primers for initiating the viral mRNA synthesis in a process known as cap-snatching.[22] Once in the nucleus, the RNA Polymerase Protein PB2 finds a cellular pre-mRNA and binds to its 5' capped end. Then RNA Polymerase PA cleaves off the cellular mRNA near the 5' end and uses this capped fragment as a primer for transcribing the rest of the viral RNA genome in viral mRNA.[23] This is due to the need of mRNA to have a 5' cap in order to be recognized by the cell's ribosome for translation.
Since RNA proofreading enzymes are absent, the RNA-dependent RNA transcriptase makes a single nucleotide insertion error roughly every 10 thousand nucleotides, which is the approximate length of the influenza vRNA. Hence, nearly every newly manufactured influenza virus will contain a mutation in its genome.[24] The separation of the genome into eight separate segments of vRNA allows mixing (reassortment) of the genes if more than one variety of influenza virus has infected the same cell (superinfection). The resulting alteration in the genome segments packaged into viral progeny confers new behavior, sometimes the ability to infect new host species or to overcome protective immunity of host populations to its old genome (in which case it is called an antigenic shift).[13]
There are three genera of influenza virus: Influenzavirus A, Influenzavirus B and Influenzavirus C. Each genus includes only one species, or type: Influenza A virus, Influenza B virus, and Influenza C virus, respectively. Influenza A and C infect multiple species, while influenza B almost exclusively infects humans.[25][26]
Influenza A viruses are further classified, based on the viral surface proteins hemagglutinin (HA or H) and neuraminidase (NA or N). Sixteen H subtypes (or serotypes) and nine N subtypes of influenza A virus have been identified.
Further variation exists; thus, specific influenza strain isolates are identified by a standard nomenclature specifying virus type, geographical location where first isolated, sequential number of isolation, year of isolation, and HA and NA subtype.[27][28]
Examples of the nomenclature are:
The type A viruses are the most virulent human pathogens among the three influenza types and cause the most severe disease. The serotypes that have been confirmed in humans, ordered by the number of known human pandemic deaths, are:
Influenza B virus is almost exclusively a human pathogen, and is less common than influenza A. The only other animal known to be susceptible to influenza B infection is the seal.[37] This type of influenza mutates at a rate 2–3 times lower than type A[38] and consequently is less genetically diverse, with only one influenza B serotype.[25] As a result of this lack of antigenic diversity, a degree of immunity to influenza B is usually acquired at an early age. However, influenza B mutates enough that lasting immunity is not possible.[39] This reduced rate of antigenic change, combined with its limited host range (inhibiting cross species antigenic shift), ensures that pandemics of influenza B do not occur.[40]
The influenza C virus infects humans and pigs, and can cause severe illness and local epidemics.[41] However, influenza C is less common than the other types and usually seems to cause mild disease in children.[42][43]
Mammalian influenza viruses tend to be labile, but can survive several hours in mucus.[44] Avian influenza virus can survive for 100 days in distilled water at room temperature, and 200 days at 17 °C(63 °F). The avian virus is inactivated more quickly in manure, but can survive for up to 2 weeks in feces on cages. Avian influenza viruses can survive indefinitely when frozen.[44] Influenza viruses are susceptible to bleach, 70% ethanol, aldehydes, oxidizing agents, and quaternary ammonium compounds. They are inactivated by heat of 133 °F(56 °C) for minimum of 60 minutes, as well as by low pH <2.[44]
Vaccines and drugs are available for the prophylaxis and treatment of influenza virus infections. Vaccines are composed of either inactivated or live attenuated virions of the H1N1 and H3N2 human influenza A viruses, as well as those of influenza B viruses. Because the antigenicities of the wild viruses evolve, vaccines are reformulated annually by updating the seed strains. However, when the antigenicities of the seed strains and wild viruses do not match, vaccines fail to protect the vaccinees. In addition, even when they do match, escape mutants are often generated. Drugs available for the treatment of influenza include Amantadine and Rimantadine, which inhibit the uncoating of virions by interfering with M2, and Oseltamivir (marketed under the brand name Tamiflu), Zanamivir, and Peramivir, which inhibit the release of virions from infected cells by interfering with NA. However, escape mutants are often generated for the former drug and less frequently for the latter drug.[45]
台灣於1972年時首次發現家禽流行性感冒病毒感染症,血清型為H6N1 ,現今在台灣H血清型共十型 (H1,2,3,4,6,7,8,10,11,14), N血清型共八型 (N1,2,3,4,6,7,8,9) ,其組合共十五種。A型流行性感冒病毒對豬而言,可造成嚴重的增殖性壞死性肺炎。
|author-separator= (帮助) 正黏液病毒科(Orthomyxoviridae,希臘文Orthos有「正確,直」之意;myxo有「粘液」之意)屬於負鏈RNA病毒,共有六個屬,此科的病毒可感染脊椎動物。造成流行性感冒的病毒正是正黏液病毒科的一員,屬於負鏈RNA病毒。正黏液病毒科可分為四屬病毒,三屬為流行性感冒病毒,分為A型、B型及C型流行性感冒病毒,以及传染性鲑鱼贫血病毒屬和托高土病毒屬。流行性感冒病毒可以感染人、馬、豬及禽類(參見禽流感),其中A型流行性感冒病毒可以感染人、豬、馬及鳥類,而B型流行性感冒,僅可感染人類。C型流行性感冒病毒僅感染人、豬,造成之病害較少。 A型及B型流行性感冒病毒之感染力非常強,傳播速度非常快,可以稱之為最重要的疾病之一,感染範圍遍及世界各大洲。水禽類,尤其是候鳥,為各不同血清型病毒之帶原者及病毒重組之動物。传染性鲑鱼贫血病毒則鮭魚,托高土病毒則可以感染脊椎動物及非脊椎動物,如蚊子和海蝨。
本科中有三屬為流感病毒,其分類方法依照其表面NP及M抗原相異性:
甲型流感病毒:感染人、鳥,和其他爬蟲類,是大流行的元兇。 乙型流感病毒:人、海豹 丙型流感病毒:感染人、豬、狗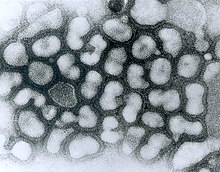
オルトミクソウイルス科またはオルソミクソウイルス科(Orthomyxoviridae)はRNAウイルスの科の一つ。呼吸器感染症を引き起こすインフルエンザウイルス(Influenza virus)を含む。
本科のウイルスは以下のように分類される。
オルトミクソウイルス科またはオルソミクソウイルス科(Orthomyxoviridae)はRNAウイルスの科の一つ。呼吸器感染症を引き起こすインフルエンザウイルス(Influenza virus)を含む。
오르토믹소바이러스과(영어: Orthomyxoviridae)는 인플루엔자 바이러스가 포함되어 있는 RNA 바이러스의 한 과이다. 인플루엔자바이러스 A형, 인플루엔자바이러스 B형, 인플루엔자바이러스 C형, 인플루엔자바이러스 D형, 연어빈혈증바이러스, 콰란자바이러스, 토고토바이러스의 7개의 속을 포함하고 있다. 처음 4개 바이러스는 새(조류인플루엔자 참고), 사람, 다른 포유류를 포함한 척추동물들에게 인플루엔자를 감염시킨다. 연어빈혈증바이러스는 연어를 감염시킨다. 토고토 바이러스는 척추동물 뿐 아니라, 진드기와 모기와 같은 무척추 동물을 감염시키는 아르보 바이러스 입니다.
척추동물을 감염시키는 4개의 인플루엔자는 핵산 단백질과 매트릭스 단백질의 항원 차이로 식별할 수 있습니다.
네 개의 인플루엔자 바이러스 속이 존재하는데, 각각의 속은 한가지 종류씩 있다. 인플루엔자 A와 C는 여러 종을 감염시킨다. 인플루엔자 B는 거의 사람들만 특정해서 감염이 된다. 인플루엔자 D는 소 떼나 돼지 등에 감염된다.[3][4][5]
포유류와 조류를 숙주로 삼으며, 대부분의 경우 비병원성이지만 일부 아종의 경우 숙주에게 인플루엔자를 일으킨다.[6] 인플루엔자바이러스 A형은 보통 큰 병으로 확산되지 않는데, 특정 변이가 닭이나 오리 등의 가정 가금류와 인간에게 심각한 질병을 가져오는 경우가 있다. 때때로 바이러스는 야생 수생 조류에서 가금류로 전염되고, 이는 인플루엔자 전염병을 일으킬 수 있다.[7][8] 이들은 유전물질로 단일가닥, 분절형의 RNA를 가지고 있다.
오로지 사람과 물개만을 숙주로 삼으며[9], 숙주에게 인플루엔자를 일으킨다. 숙주의 범위가 좁기 때문에 다양한 숙주를 가질 수 있는 A형 인플루엔자 바이러스에 비해 범유행을 일으키는 경우가 매우 적다.[10][11][12]
인플루엔자바이러스 A형이나 B형에 비해 발병 빈도는 적지만, 일단 감염되면 심각한 상태를 일으키고, 국소적인 유행을 일으킬 수도 있다. 그러나 다른 인플루엔자 바이러스에 비해서 변이 속도가 느리기 때문에 범유행을 일으키지는 않는다. 인플루엔자바이러스 C형에 대한 백신은 2010년 3월 현재 개발된 것이 없다.[13]
2011년에 돼지에게서 처음으로 분리되어 2016년에 오르토믹소바이러스과가 만들어지며 새로 분류되었다.[14][15] 이전에는 인플루엔자바이러스 C형의 아류로 여겨지며 인플루엔자바이러스 C형속에 속하였었다.[14]
오르토믹소바이러스과(영어: Orthomyxoviridae)는 인플루엔자 바이러스가 포함되어 있는 RNA 바이러스의 한 과이다. 인플루엔자바이러스 A형, 인플루엔자바이러스 B형, 인플루엔자바이러스 C형, 인플루엔자바이러스 D형, 연어빈혈증바이러스, 콰란자바이러스, 토고토바이러스의 7개의 속을 포함하고 있다. 처음 4개 바이러스는 새(조류인플루엔자 참고), 사람, 다른 포유류를 포함한 척추동물들에게 인플루엔자를 감염시킨다. 연어빈혈증바이러스는 연어를 감염시킨다. 토고토 바이러스는 척추동물 뿐 아니라, 진드기와 모기와 같은 무척추 동물을 감염시키는 아르보 바이러스 입니다.
척추동물을 감염시키는 4개의 인플루엔자는 핵산 단백질과 매트릭스 단백질의 항원 차이로 식별할 수 있습니다.
인플루엔자바이러스 A형은 사람이나 다른 포유류, 조류 및 모든 독감 유형의 대유행을 일으킨다. 인플루엔자바이러스 B형은 사람과 물개를 감염시킨다. 인플루엔자바이러스 C형은 사람과 돼지 및 개를 감염시킨다. 인플루엔자바이러스 D형은 돼지와 소를 감염시킨다.