fr
noms dans le fil d’Ariane

Ashbya gossypii (auch Eremothecium gossypii) ist ein Schimmelpilz, der mit Bäckerhefe nahe verwandt ist. Er wurde 1926 erstmals von Ashby und Nowell als Pathogen von Baumwollpflanzen beschrieben, bei denen er die so genannte Stigmatomykose verursacht. Die Krankheit betrifft die Entwicklung von Haarzellen in den Samenkapseln von Baumwolle und kann auf Zitrusfrüchte übertragen werden, die daraufhin austrocknen und zusammenfallen (Trockenfäule). A. gossypii ist ein natürlicher Überproduzent von Riboflavin (Vitamin B2), weshalb er auch industriell interessant wurde.[1][2]
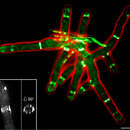
Der Lebenszyklus von A. gossypii fängt mit dem nach allen Seiten gleichmäßigen (Isotropie) Wachstum der haploiden Spore, ihrer Keimung an. Darauf folgt apikales Wachstum, das zwei Keimröhren nacheinander auf gegenüberliegenden Seiten der Keimblase hervorbringt. Weitere Achsen werden beim lateralen Abzweigen in jungem Myzel gebildet. Die Reifung ist gekennzeichnet durch apikale Abzweigungen (Spaltung von Spitzen) und einem dramatischen Ansteigen der Wachstumsgeschwindigkeit (bis zu 200 μm/h bei 30 °C), was es A. gossypii ermöglicht, eine Petrischale mit 8 cm Durchmesser in etwa 7 Tagen auszufüllen. Die Sporulation wird wahrscheinlich durch Nahrungsentzug hervorgerufen und führt zu einer Kontraktion des Septum, Zytokinese und nachfolgender Absonderung der Sporangien, die bis zu acht haploide Sporen enthalten können. Die Hyphen selbst sind im Allgemeinen durch Septa unterteilt, die zunächst als Ring erscheinen, der den Transfer von Kernmaterial zulässt; später ändert sich das Aussehen der Septa zu einer geschlossenen Scheibe. Jedes Hyphenkompartiment enthält etwa acht Zellkerne.[3]
Die genetische Information von A. gossypii liegt im Zellkern in 7 Autosomen sowie im Kern der Mitochondrien vor. Das Genom des Stamms ATCC 10895 wurde im Jahr 2004 erstmals vollständig analysiert; es besteht aus 9 Millionen Basenpaaren und geschätzten 4.750 Genen. Es ist damit das kleinste bekannte Genom eines frei lebenden Eukaryoten. Neunzig Prozent der Gene in A. gossypii sind sowohl homolog als auch in derselben Reihenfolge (Syntänie) wie in Saccharomyces cerevisiae. Der Vorfahr beider Organismen lebte vor 100 Millionen Jahren. Während A. gossypii sich nur wenig veränderte, ereignete sich bei der folgenden Evolution der Bäckerhefe eine Genduplikation.[4][5][6]
A. gossypii und zwei weitere Pilze, die Stigmatomykose verursachen (Eremothecium coryli, Aureobasidium pullulans), machten es in der ersten Hälfte des 20. Jahrhunderts nahezu unmöglich, Baumwolle in manchen subtropischen Regionen zu kultivieren. Erst die Kontrolle der sporenübertragenden Insektenarten Dysdercus suturellus und Antestia angulosa konnte daraufhin die Infektion der Baumwolle verhindern.[7]
Die erste gezielte Einbringung von DNA (Transformation) als Voraussetzung für weitere molekulargenetische Arbeiten zeigte, dass im Gegensatz zu anderen filamentösen Pilzen ringförmige Plasmide in A.gossypii frei replizieren[8]. Mit Plasmiden, die A.gossypii-DNA enthalten und in dieser Region linearisiert werden, wird homologe Integration erreicht[9]. Aufgrund seines kleinen und außerdem haploiden Genoms und bestehender effizienter Methoden zum Gene-Targeting ist A. gossypii anerkannt als Modellorganismus, insbesondere zur Untersuchung des Wachstums langer und mehrkerniger Pilzzellen. Es wird allgemein angenommen, dass ein besseres Verständnis des Hyphenwachstums die Entwicklung neuartiger Fungizide ermöglichen wird. Besonders vielversprechend ist Ashbya gossypii als Modellorganismus auch aufgrund der hohen Konservierung der Genreihenfolge (Syntänie) zwischen den Genomen von A. gossypii und Saccharomyces cerevisiae.[6]
A. gosyypii produziert große Mengen an Riboflavin. A.gossypii diente zur Aufklärung des Riboflavinbiosyntheseweges[10]. Der Organismus wird zur industriellen Produktion von Riboflavin genutzt und wurde dazu weiter optimiert[11][12].
In neueren Werken sind die Gattungen Ashbya, Holleya, und Nematospora synonym zu Eremothecium. Die vier Gattungen zeichnen sich durch nadelförmige Ascosporen aus, die gerade oder gebogen sein können. Die Taxa sind pathogen an verschiedenen Pflanzenarten. Phylogenetische Analysen ribosomaler RNA und DNA in den 1990er Jahren sind sich in der Auffassung einig, dass die vier Arten Kongenere sind.[13][14][15]
Ashbya gossypii (auch Eremothecium gossypii) ist ein Schimmelpilz, der mit Bäckerhefe nahe verwandt ist. Er wurde 1926 erstmals von Ashby und Nowell als Pathogen von Baumwollpflanzen beschrieben, bei denen er die so genannte Stigmatomykose verursacht. Die Krankheit betrifft die Entwicklung von Haarzellen in den Samenkapseln von Baumwolle und kann auf Zitrusfrüchte übertragen werden, die daraufhin austrocknen und zusammenfallen (Trockenfäule). A. gossypii ist ein natürlicher Überproduzent von Riboflavin (Vitamin B2), weshalb er auch industriell interessant wurde.
Eremothecium gossypii[1] (also known as Ashbya gossypii[2]) is a filamentous fungus or mold closely related to yeast, but growing exclusively in a filamentous way. It was originally isolated from cotton as a pathogen causing stigmatomycosis by Ashby and Nowell in 1926. This disease affects the development of hair cells in cotton bolls and can be transmitted to citrus fruits, which thereupon dry out and collapse (dry rot disease). In the first part of the 20th century, E. gossypii and two other fungi causing stigmatomycosis (Eremothecium coryli, Aureobasidium pullulans) made it virtually impossible to grow cotton in certain regions of the subtropics, causing severe economical losses. Control of the spore-transmitting insects - cotton stainer (Dysdercus suturellus) and Antestiopsis (antestia bugs) - permitted full eradication of infections. E. gossypii was recognized as a natural overproducer of riboflavin (vitamin B2), which protects its spores against ultraviolet light. This made it an interesting organism for industries, where genetically modified strains are still used to produce this vitamin.
A few years ago, E. gossypii became recognized as an attractive model to study the growth of long and multinucleate fungal cells (hyphae) because of its small genome, haploid nuclei, and efficient gene targeting methods. It is generally assumed that a better understanding of filamentous fungal growth will greatly stimulate the development of novel fungicides. Its use as a model organism is particularly promising because of the high level of gene order conservation (synteny) between the genomes of E. gossypii and the yeast Saccharomyces cerevisiae.
The complete sequencing and annotation of the entire E. gossypii genome, as published in 2004, was initiated when a significant degree of gene synteny was observed in preliminary studies in comparison to the genome of budding yeast, Saccharomyces cerevisiae. This not only helped to improve gene annotation of S. cerevisiae, but also allowed the reconstruction of the evolutionary history of both organisms. E. gossypii and S. cerevisiae originated from a common ancestor which carried about 5000 genes. Divergence of these two close relatives started some 100 million years ago. One branch of evolution involving up to 100 viable genome rearrangements (translocations and inversions), a few million base pair changes, and a limited number of gene deletions, duplications and additions lead to modern E. gossypii with its 4718 protein-coding genes and 9.2 million base pairs (smallest genome of a free-living eukaryote yet characterized) spread over seven chromosomes. The genome of S. cerevisiae underwent a more eventful evolution, which includes a whole-genome duplication.
Despite the long evolutionary history of the two organisms and fundamentally different ways of growth and development, the complete synteny map of both genomes reveals 95% of E. gossypii genes are orthologs of S. cerevisiae genes, and 90% map within blocks of synteny (syntenic homologs).

The E. gossypii life cycle starts with the only known phase of isotropic growth in wild type: germination of the haploid spore to form a germ bubble. This is followed by apical growth, extending two germ tubes in succession on opposing sites of the germ bubble. More axes of polarity are established with lateral branch formation in young mycelium. Maturation is characterized by apical branching (tip splitting) and a dramatic increase of growth speed (up to 200 μm/h at 30 °C), which enables it to cover an 8 cm Petri dish of full medium in about seven days. Sporulation is thought to be induced by nutrient deprivation, leading to contraction at the septa, cytokinesis and subsequent abscission of sporangia which contain up to eight haploid spores. Hyphae are compartmentalized by septa, which in young parts appear as rings that allow transfer of nuclei and in older parts may appear as closed discs. Compartments typically contain around eight nuclei.
Eremothecium gossypii (also known as Ashbya gossypii) is a filamentous fungus or mold closely related to yeast, but growing exclusively in a filamentous way. It was originally isolated from cotton as a pathogen causing stigmatomycosis by Ashby and Nowell in 1926. This disease affects the development of hair cells in cotton bolls and can be transmitted to citrus fruits, which thereupon dry out and collapse (dry rot disease). In the first part of the 20th century, E. gossypii and two other fungi causing stigmatomycosis (Eremothecium coryli, Aureobasidium pullulans) made it virtually impossible to grow cotton in certain regions of the subtropics, causing severe economical losses. Control of the spore-transmitting insects - cotton stainer (Dysdercus suturellus) and Antestiopsis (antestia bugs) - permitted full eradication of infections. E. gossypii was recognized as a natural overproducer of riboflavin (vitamin B2), which protects its spores against ultraviolet light. This made it an interesting organism for industries, where genetically modified strains are still used to produce this vitamin.
Eremothecium gossypii je grzib[5], co go nojprzōd ôpisoł S.F. Ashby & W. Nowell, a terŏźnõ nazwã doł mu Kurtzman 1995. Eremothecium gossypii nŏleży do zorty Eremothecium i familije Eremotheciaceae.[6][7] Żŏdne podgatōnki niy sōm wymianowane we Catalogue of Life.[6]
Eremothecium gossypii je grzib, co go nojprzōd ôpisoł S.F. Ashby & W. Nowell, a terŏźnõ nazwã doł mu Kurtzman 1995. Eremothecium gossypii nŏleży do zorty Eremothecium i familije Eremotheciaceae. Żŏdne podgatōnki niy sōm wymianowane we Catalogue of Life.