fi
nimet breadcrumb-navigoinnissa

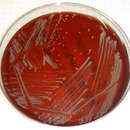
Mycobacterium smegmatis ist ein Bakterium aus der Gruppe der Mykobakterien. Es handelt sich um stäbchenförmige Bakterien mit einer Länge von 3 bis 5 µm.[1] Es zählt zur Normalflora des Smegmas im menschlichen Genitalbereich, also sowohl im Bereich der weiblichen Vulva und der Klitorisvorhaut, Praeputium clitoridis, als auch beim Mann unter der Penisvorhaut, Praeputium penis.
Auch in Bodenproben wurde es häufig gefunden.[2] Es ist nur in seltenen Fällen pathogen[3] und zählt zu den sogenannten nichttuberkulösen Mykobakterien (MOTT). Mycobacterium smegmatis zählt hier zu den schnellwachsenden Mykobakterien, die auf agarhaltigen Nährmedien schon innerhalb einer Woche gut sichtbare Kolonien bilden. Deshalb gilt es auch als Modellorganismus für Mykobakterien und wird oft in Labors für Stoffwechseluntersuchungen verwendet.[4] Die elektronenmikroskopische Charakterisierung erfolgte unter Einsatz der Kryo-Elektronenmikroskopie.[5]
Das Genom wurde vom J. Craig Venter Institute sequenziert.[6] Auch die Mykolsäuren wurden, wie auch von anderen Arten von Mycobacterium, analysiert.[7]
Mycobacterium smegmatis ist eine Art in der Gattung Mycobacterium in der Familie der Mycobacteriaceae, die zur Abteilung Actinobacteria gezählt wird. Zusammen mit Mycobacterium goodii wird es in die Mycobacterium smegmatis-Clade gestellt.[1]
Mycobacterium smegmatis ist ein Bakterium aus der Gruppe der Mykobakterien. Es handelt sich um stäbchenförmige Bakterien mit einer Länge von 3 bis 5 µm. Es zählt zur Normalflora des Smegmas im menschlichen Genitalbereich, also sowohl im Bereich der weiblichen Vulva und der Klitorisvorhaut, Praeputium clitoridis, als auch beim Mann unter der Penisvorhaut, Praeputium penis.
Auch in Bodenproben wurde es häufig gefunden. Es ist nur in seltenen Fällen pathogen und zählt zu den sogenannten nichttuberkulösen Mykobakterien (MOTT). Mycobacterium smegmatis zählt hier zu den schnellwachsenden Mykobakterien, die auf agarhaltigen Nährmedien schon innerhalb einer Woche gut sichtbare Kolonien bilden. Deshalb gilt es auch als Modellorganismus für Mykobakterien und wird oft in Labors für Stoffwechseluntersuchungen verwendet. Die elektronenmikroskopische Charakterisierung erfolgte unter Einsatz der Kryo-Elektronenmikroskopie.
Das Genom wurde vom J. Craig Venter Institute sequenziert. Auch die Mykolsäuren wurden, wie auch von anderen Arten von Mycobacterium, analysiert.
Mycobateriun smegmatis ye ua spece batéria álcol-ácido resistente de l género Mycobateriun. Eilha fui purmeiramente çcrita an nobembre de 1884 por Lustgarten qu'ancuontrou un bacilo cula aparéncia quelorada de tubercle bacilli (bacilos tuberculosos) an cancros sifilíticos. Mais tarde, Albareç i Table ancontrórun ourganismos similares aqueilhes çcritos por Lustgarten assi cumo an secreçones genitales normales (smegma). Este ourganismo fui mais tarde chamado M. smegmatis.
Mycobacterium smegmatis is an acid-fast bacterial species in the phylum Actinomycetota and the genus Mycobacterium. It is 3.0 to 5.0 µm long with a bacillus shape and can be stained by Ziehl–Neelsen method and the auramine-rhodamine fluorescent method. It was first reported in November 1884 by Lustgarten, who found a bacillus with the staining appearance of tubercle bacilli in syphilitic chancres. Subsequent to this, Alvarez and Tavel found organisms similar to that described by Lustgarten also in normal genital secretions (smegma). This organism was later named M. smegmatis.[1]
Some species of the genus Mycobacterium have recently been renamed to Mycolicibacterium, so that M. smegmatis is now Mycolicibacterium smegmatis.[2][3]
M. smegmatis, which was previously considered a nonmotile organism, uses a sliding mechanism that allows it to move around its environment. Henrichsen[4] defines it as, “a kind of surface translocation produced by the expansive forces in a growing culture in combination with special surface properties of the cells resulting in reduced friction between cell and substrate”. Essentially, the bacteria form a single-layered sheet and are able to move slowly together without the use of any extracellular structures, like flagella or pili. Although it hasn’t been determined exactly how this mechanism works, the surface properties of the unique cell wall (Figure 1) of M. smegmatis have been found to play a role. For example, this sliding ability is correlated with the presence of glycopeptidolipids (GLPs) on the outermost part of the cell wall. GLPs are amphiphilic molecules that could potentially decrease surface interactions or create a conditioning film that allows movement. Although the exact role of GLPs in sliding is not known, without them M. smegmatis does not have the ability to translocate.[5]
M. smegmatis is generally considered a non-pathogenic microorganism; however, in some very rare cases, it may cause disease.[6]
Mycobacterium smegmatis is useful for the research analysis of other Mycobacteria species in laboratory experiments. M. smegmatis is commonly used in work on the Mycobacterium genus due to it being a "fast grower" and non-pathogenic. M. smegmatis is a simple model that is easy to work with, i.e., with a fast doubling time and only requires a biosafety level 1 laboratory. The time and heavy infrastructure needed to work with pathogenic species prompted researchers to use M. smegmatis as a model for mycobacterial species.
Mycobacterium smegmatis shares the same peculiar cell wall structure of M. tuberculosis and other mycobacterial species.[7] It is also capable of oxidizing carbon monoxide aerobically, as is M. tuberculosis.
Bacterial secretion systems are specialized protein complexes and pathways that allow bacterial pathogens to secrete proteins across their cell membranes and, ultimately, to host cells. These effector proteins are important virulence factors, which allow the pathogen to survive inside of the host. There are many different kinds of specific secretion systems, and M. tuberculosis has an Snm (secretion in mycobacteria) protein secretion system. Although the Snm secretion system is a key in determining M. tuberculosis virulence, all mycobacteria have genes encoding the components of this system. This area of the genome is referred to as the RD1 locus. M. smegmatis is commonly used to study Snm secretion because of its genetic similarities and analogous function to M. tuberculosis, as well as ease of growing in the lab. One example of how this can be applied in research is the identification of gene products required for Snm secretion. By knocking out genes in the RD1 locus of M. smegmatis and testing efficiency of Snm secretion before and after gene knockout, specific genes can be identified as necessary for Snm secretion. These findings can be applied to the Snm secretion system of M. tuberculosis.[8]
Mycobacterium smegmatis is readily cultivatable in most synthetic or complex laboratory media, where it can form visible colonies in 3–5 days. These properties make it a very attractive model organism for M. tuberculosis and other mycobacterial pathogens. M. smegmatis mc2155 is also used for the cultivation of mycobacteriophage.
Like many other bacteria, M. smegmatis is known to use the trace levels of hydrogen in the atmosphere as an energy source. In 2023, researchers reported extracting from M. smegmatis a hydrogenase called Huc, which is highly efficient at oxidizing hydrogen gas—and thus creating an electric current—while also being insensitive to the presence of oxygen, which typically obstructs catalysis.[9] This discovery offers significant potential for green energy.
The genomes of multiple strains of M. smegmatis have been sequenced by TIGR and other laboratories, including the "wild-type" (mc2 155) and some antibiotic-resistant strains (4XR1/R2).[10] The genome of strain mc2155 is ~6,9 Mbp long and encodes ~6400 proteins[11] which is relatively large for bacteria (for comparison, the genome of E. coli encodes about 4000 proteins).
This species shares more than 2000 homologous genes with M. tuberculosis and thus is a good model organism to study mycobacteria in general and the highly pathogenic M. tuberculosis in particular.
The discovery of plasmids, phages, and mobile genetic elements has enabled the construction of dedicated gene-inactivation and gene reporter systems. The M. smegmatis mc2155 strain is hypertransformable, and is now the work-horse of mycobacterial genetics.
Transformation is a process by which a bacterial cell takes up DNA that had been released by another cell into the surrounding medium, and then incorporates that DNA into its own genome by homologous recombination (see Transformation (genetics)). Strains of M. smegmatis that have particularly efficient DNA repair machinery, as indicated by their greater resistance to the DNA damaging effects of agents such as UV and mitomycin C, proved to be the most capable of undergoing transformation.[12] This suggests that transformation in M. smegmatis is a DNA repair process, presumably a recombinational repair process, as it is in other bacterial species.[13]
Conjugal DNA transfer in M. smegmatis requires stable and extended contact between a donor and a recipient strain, is DNase resistant, and the transferred DNA is incorporated into the recipient’s chromosome by homologous recombination. However, in contrast to the well-known E. coli Hfr conjugation system, in M. smegmatis all regions of the chromosome are transferred with comparable efficiencies and mycobacterial conjugation is chromosome, rather than plasmid based. Gray et al.[14] reported substantial blending of the parental genomes resulting from conjugation and referred to this blending as reminiscent of that seen in the meiotic products of sexual reproduction (see Origin of sexual reproduction).
Mycobacterium smegmatis relies on DNA repair pathways to resist DNA damage. Double-strand breaks are especially threatening to bacterial viability. M. smegmatis has three options for repairing double-strand breaks; homologous recombination (HR), non-homologous end joining (NHEJ), and single-strand annealing (SSA).[15] The HR pathway of M. smegmatis is the major determinant of resistance to ionizing radiation and oxidative DNA damage. This pathway involves exchange of information between a damaged chromosome and another homologous chromosome in the same cell. It depends on the RecA protein that catalyzes strand exchange and the ADN protein that acts as a presynaptic nuclease.[15] HR is an accurate repair process and is the preferred pathway during logarithmic growth.[16]
The NHEJ pathway for repairing double-strand breaks involves the rejoining of the broken ends. It does not depend on a second homologous chromosome. This pathway requires the Ku protein and a specialized poly-functional ATP-dependent DNA ligase (ligase D).[17] NHEJ is efficient but inaccurate. Sealing of blunt DNA ends within a functional gene sequence occurs with a mutation frequency of about 50%.[17] NHEJ is the preferred pathway during stationary phase, and it protects M. smegmatis against the harmful effects of desiccation.[16]
SSA is employed as a repair pathway when a double-strand break arises between direct repeat sequences in DNA. SSA involves single-strand resection, annealing of the repeats, flap removal, gap filling and ligation. In M. smegmatis the SSA pathway depends on the RecBCD helicase-nuclease.[15]
Mycobacterium smegmatis is an acid-fast bacterial species in the phylum Actinomycetota and the genus Mycobacterium. It is 3.0 to 5.0 µm long with a bacillus shape and can be stained by Ziehl–Neelsen method and the auramine-rhodamine fluorescent method. It was first reported in November 1884 by Lustgarten, who found a bacillus with the staining appearance of tubercle bacilli in syphilitic chancres. Subsequent to this, Alvarez and Tavel found organisms similar to that described by Lustgarten also in normal genital secretions (smegma). This organism was later named M. smegmatis.
Some species of the genus Mycobacterium have recently been renamed to Mycolicibacterium, so that M. smegmatis is now Mycolicibacterium smegmatis.
M. smegmatis, which was previously considered a nonmotile organism, uses a sliding mechanism that allows it to move around its environment. Henrichsen defines it as, “a kind of surface translocation produced by the expansive forces in a growing culture in combination with special surface properties of the cells resulting in reduced friction between cell and substrate”. Essentially, the bacteria form a single-layered sheet and are able to move slowly together without the use of any extracellular structures, like flagella or pili. Although it hasn’t been determined exactly how this mechanism works, the surface properties of the unique cell wall (Figure 1) of M. smegmatis have been found to play a role. For example, this sliding ability is correlated with the presence of glycopeptidolipids (GLPs) on the outermost part of the cell wall. GLPs are amphiphilic molecules that could potentially decrease surface interactions or create a conditioning film that allows movement. Although the exact role of GLPs in sliding is not known, without them M. smegmatis does not have the ability to translocate.
 Plaques from a virus isolated from a compost heap near UCLA. The bacterium is M. smegmatis
Plaques from a virus isolated from a compost heap near UCLA. The bacterium is M. smegmatis
Mycobacterium smegmatis es un bacilo de 3 a 5 µm de longitud ácido-alcohol resistente perteneciente al filo Actinobacteria. Se tiñe con las tinciones de Ziehl-Neelsen y auramina. Fue descubierto en 1884 por Lustgarten en un chancro sifilítico. Posteriormente, Álvarez y Tavel encontraron organismos similares en el esmegma normal. M. smegmatis se considera una microorganismo no patógeno, aunque puede causar enfermedad en animales inmunodeprimidos.
Mycobacterium smegmatis est une espèce bactérienne acido-résistante du phylum Actinobacteria et du genre Mycobacterium. Ce microbe est long de 5 μm et présente une forme de bacille ; il peut être coloré par la méthode Ziehl-Neelsen et la méthode fluorescente auramine-rhodamine. Il a été signalé pour la première fois en novembre 1884 par Lustgarten, qui a trouvé un bacille avec la coloration apparente de bacilles tuberculeux dans les chancres syphilitiques. Par la suite, Alvarez et Tavel ont trouvé des organismes similaires à ceux décrits par Lustgarten également dans les sécrétions génitales normales (smegma). Cet organisme a été nommé plus tard M. smegmatis[1].
M. smegmatis est généralement considéré comme un micro-organisme non pathogène ; cependant, dans certains cas très rares, il peut provoquer une maladie[2].
M. smegmatis est utile pour l'analyse de recherche d'autres espèces de Mycobactéria dans des expériences de laboratoire. M. smegmatis est couramment utilisé dans les travaux sur le genre Mycobacterium car il est du type « croissance rapide » et non pathogène. M. smegmatis est un modèle simple, facile à travailler, c'est-à-dire avec un temps de doublement rapide et ne nécessite qu'un laboratoire de niveau de biosécurité 1. Le temps et les infrastructures lourdes nécessaires pour travailler avec des espèces pathogènes ont incité les chercheurs à utiliser M. smegmatis comme modèle pour les espèces mycobactériennes. Cette espèce partage plus de 2000 gènes homologues avec M. tuberculosis et partage la même structure de paroi cellulaire particulière que M. tuberculosis et d'autres espèces mycobactériennes[3]. Il est également capable d'oxyder le monoxyde de carbone en aérobie, tout comme M. tuberculosis.
La découverte de plasmides, de phages et d'éléments génétiques mobiles a permis la construction de systèmes dédiés d'inactivation de gènes et de rapporteurs de gènes. La souche M. smegmatis mc2155 est hypertransformable et est désormais le cheval de bataille de la génétique mycobactérienne. De plus, il est facilement cultivable dans la plupart des milieux de laboratoire synthétiques ou complexes, où il peut former des colonies visibles en 3 à 5 jours. Ces propriétés en font un organisme modèle très attractif pour M. tuberculosis et d'autres pathogènes mycobactériens. M. smegmatis mc2155 est également utilisé pour la culture de mycobactériophages. Le génome complet de M. smegmatis a été séquencé par TIGR, et des puces à ADN ont été produites par le programme PFGRC (http://pfgrc.tigr.org/descriptionPages.shtml), ajoutant encore à son utilisation comme système modèle pour étudier les mycobactéries.
La transformation est un processus par lequel une cellule bactérienne absorbe l'ADN qui avait été libéré par une autre cellule dans le milieu environnant, puis incorpore cet ADN dans son propre génome par recombinaison homologue (voir Transformation (génétique)). Ce sont les souches de M. smegmatis qui ont des machineries de réparation de l'ADN particulièrement efficaces, comme l'indique leur plus grande résistance aux effets néfastes de l'ADN d'agents tels que les UV et la mitomycine C, qui se sont révélées être les plus capables de subir une transformation[4]. Cela suggère que la transformation de M. smegmatis est un processus de réparation de l'ADN, vraisemblablement un processus de réparation recombinant, comme c'est le cas pour d'autres espèces bactériennes[5].
Le transfert d'ADN conjugal chez M. smegmatis nécessite un contact stable et prolongé entre un donneur et une souche receveuse, est résistant à la DNase et l'ADN transféré est incorporé dans le chromosome du receveur par recombinaison homologue. Cependant, contrairement au système de conjugaison bien connu de E. coli Hfr, chez M. smegmatis, toutes les régions du chromosome sont transférées avec des efficacités comparables et la conjugaison mycobactérienne est chromosomique plutôt que plasmidique. Gray et al.[6] rapportèrent un mélange substantiel des génomes parentaux résultant de la conjugaison et ont fait référence à ce mélange comme rappelant celui observé dans les produits méiotiques de la reproduction sexuelle (voir Origine de la reproduction sexuelle).
M. smegmatis s'appuie sur les voies de réparation de l'ADN pour résister aux dommages de l'ADN. Les ruptures double brin menacent particulièrement la viabilité bactérienne. M. smegmatis a trois options pour réparer les ruptures double brin : recombinaison homologue (RH), jonction d'extrémité non homologue (NHEJ) et l'annelage brin-unique (SSA, de l'anglais : single-strand annealing)[7]. La voie RH de M. smegmatis est le principal déterminant de la résistance aux rayonnements ionisants et aux dommages oxydatifs à l'ADN. Cette voie implique un échange d'informations entre un chromosome endommagé et un autre chromosome homologue dans la même cellule. Cela dépend de la protéine RecA qui catalyse l'échange de brins et de la protéine ADN qui agit comme une nucléase présynaptique. La recombinaison homologue (RH) est un processus de réparation précis, c'est la voie privilégiée au cours d'une croissance logarithmique[8].
La voie NHEJ pour réparer les ruptures à double brin implique la jonction des extrémités cassées. Il ne dépend pas d'un second chromosome homologue. Cette voie nécessite la protéine Ku et une ADN ligase poly-fonctionnelle ATP-dépendante spécialisée (ligase D)[9]. NHEJ est efficace mais inexacte. La ligature d'extrémités franches d'ADN dans une séquence de gène fonctionnelle se produit avec une fréquence de mutation d'environ 50 %. Le NHEJ est la voie privilégiée en phase stationnaire et protège M. smegmatis contre les effets nocifs de la dessiccation[8].
Le mécanisme SSA (single-strand annealing) est utilisé comme voie de réparation lorsqu'une rupture double brin survient entre des séquences de répétition directes dans l'ADN. Le SSA implique la résection simple brin, la fixation par annelage des répétitions, l'élimination des lambeaux, le remplissage des espaces et la ligature. Chez M. smegmatis, la voie SSA dépend de l'hélicase-nucléase RecBCD[7].
Mycobacterium smegmatis est une espèce bactérienne acido-résistante du phylum Actinobacteria et du genre Mycobacterium. Ce microbe est long de 3 à 5 μm et présente une forme de bacille ; il peut être coloré par la méthode Ziehl-Neelsen et la méthode fluorescente auramine-rhodamine. Il a été signalé pour la première fois en novembre 1884 par Lustgarten, qui a trouvé un bacille avec la coloration apparente de bacilles tuberculeux dans les chancres syphilitiques. Par la suite, Alvarez et Tavel ont trouvé des organismes similaires à ceux décrits par Lustgarten également dans les sécrétions génitales normales (smegma). Cet organisme a été nommé plus tard M. smegmatis.
Mycobacterium smegmatis é uma espécie bactéria álcool-ácido resistente do gênero Mycobacterium. Ela foi primeiramente descrita em novembro de 1884 por Lustgarten que encontrou um bacilo com a aparência corada de tubercle bacilli (bacilos tuberculosos) em cancros sifilíticos. Posteriormente, Alvarez e Tavel encontraram organismos similares aqueles descritos por Lustgarten assim como em secreções genitais normais (esmegma). Este organismo foi posteriormente denominado M. smegmatis.